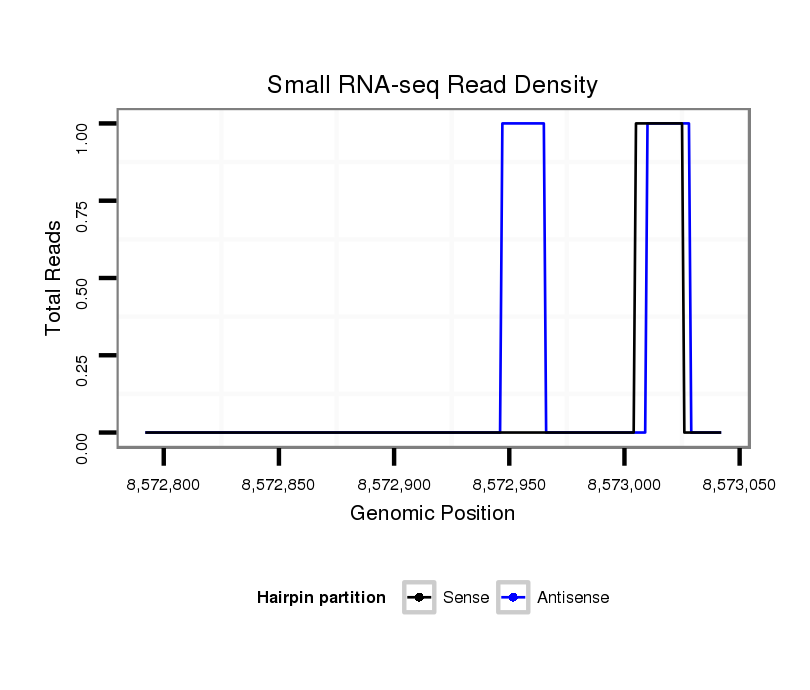
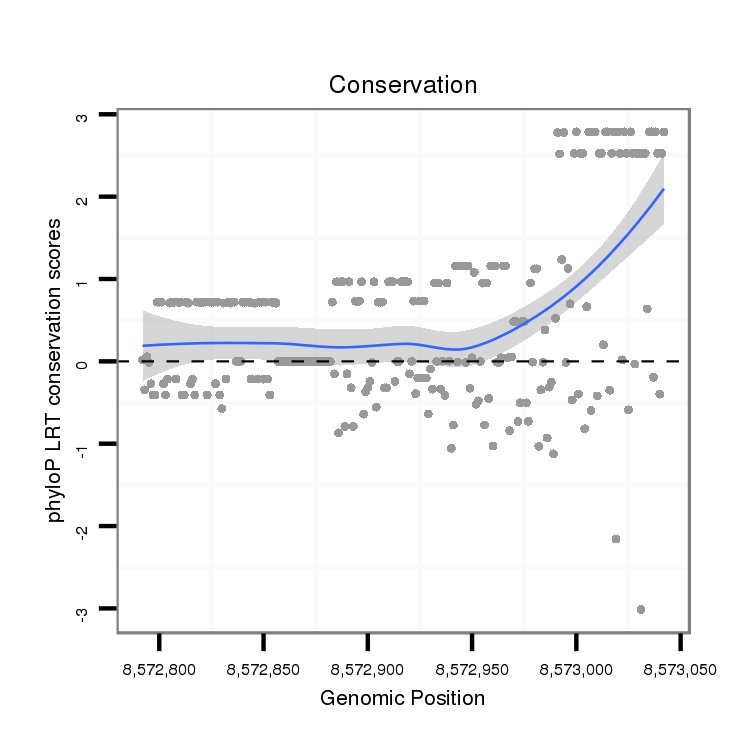
ID:dvi_10966 |
Coordinate:scaffold_12963:8572842-8572992 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ17226-cds]; exon [dvir_GLEANR_1831:3]; intron [Dvir\GJ17226-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TAACAGATAATCGCTTGATTTTCTAAAAAAATAAATATCGATCTTGGGATTAGCCTAAGATATTGCTAAGGGTTATAGCTAGCTCGACTTATCTCATCGATCTGATTAATATTATATATATACCATATCCTGCAGTCACTAGTTAGTTGGAGATACTTTCCCCTCTCACAAGATATCTATCCATCTATATCGTTTCCTCAGGTGAACGAACCGCTATACCCTATCGTATGCTCCACGGCTGCCAAGACCGA **************************************************.......(((((((....((((.(((..((((((.((((...((((..(((.((....((((.....))))..)).))).)).)).....))))))))))..))).....)))).......))))))).......................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060665 9_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR1106723 embryo_12-14h |
SRR060657 140_testes_total |
SRR060673 9_ovaries_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............................................................................................................................................AGTTAGTTCGTGATACTTTC........................................................................................... | 20 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................CATCTGTATCGATTCCTCAGG................................................. | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................CTATACCCTATCGTATGCTCC................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................TCTTTCCATCAATATTGTTTCCTC.................................................... | 24 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................TGTTGGAGATACTTACACCT....................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................................CGATATTTTCCCCTCTTACA................................................................................. | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........................................................ATATGGCTAAGGTTTATA.............................................................................................................................................................................. | 18 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ATTGTCTATTAGCGAACTAAAAGATTTTTTTATTTATAGCTAGAACCCTAATCGGATTCTATAACGATTCCCAATATCGATCGAGCTGAATAGAGTAGCTAGACTAATTATAATATATATATGGTATAGGACGTCAGTGATCAATCAACCTCTATGAAAGGGGAGAGTGTTCTATAGATAGGTAGATATAGCAAAGGAGTCCACTTGCTTGGCGATATGGGATAGCATACGAGGTGCCGACGGTTCTGGCT
**************************************************.......(((((((....((((.(((..((((((.((((...((((..(((.((....((((.....))))..)).))).)).)).....))))))))))..))).....)))).......))))))).......................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060655 9x160_testes_total |
V053 head |
M047 female body |
SRR060666 160_males_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
M027 male body |
SRR060668 160x9_males_carcasses_total |
M028 head |
SRR060664 9_males_carcasses_total |
SRR060671 9x160_males_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
V116 male body |
GSM1528803 follicle cells |
SRR060672 9x160_females_carcasses_total |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................................................................ATAGGTAGATATAACACA........................................................ | 18 | 2 | 20 | 1.75 | 35 | 0 | 0 | 1 | 5 | 7 | 2 | 5 | 4 | 3 | 2 | 2 | 2 | 0 | 0 | 1 | 1 |
| ............................................................................................................................................................................................................CTGCTTGGCGATATGGGGTAGC......................... | 22 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................GAAAGGGGAGAGTGTTCTA............................................................................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................CTGCTTGGCGATATGGGATAGC......................... | 22 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................GGGATAGCATACGAGGTGC.............. | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................TAAGCATAGGTAGATATAGCA.......................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................CATAGGTAGATATAGCACAC....................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................AAGCATAGGTAGATATAGCA.......................................................... | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................AGTGCTAGAACCCTAATTG..................................................................................................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................TAAACATAGGTAGATATAACA.......................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................................................................................TGAAAGGGGAGAATGTC................................................................................ | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................................ACGCCAGTGATTAATCTAC...................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:8572792-8573042 + | dvi_10966 | TAACAGATAATCGCTTGATTTTCTAAAAAA-----ATAAATATCGATCTTGGGATTAGCCTAAGATATTGCTAAGGGTTATAGCTAGCTCGACTTATCTCATCGATCTGATTAATATTATATATATACCATATCCTGCAGTCACTAGTTAGTTGG-------------------AGA-TACTTTCCCCTCTC-AC------------------------AAGATATCTATCCATCTATATCG--TT-TCCTCAGGTGAACGAACCGCTATACCCTATCGTATGCTCCACGGCTGCCAAGACCGA |
| droMoj3 | scaffold_6500:1184562-1184682 + | AATT------------------------------------------------------------------------------------------------------------------------------------------CCCAGTCAATTCC-------------------AGAATATTCTCTACTGTATAA------------------------ATAATATACATATATTGATATAT--TTCACTGTAGGTAACCGAACCTCTCTATCCCATTGTCTGCTCCACCGCAGCCAAAACGGA | |
| droGri2 | scaffold_15252:11768733-11768797 + | AT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATTT--TT-CTCGCAGGTGACTGAGCCGCTTTATCCCATTGTCTGCTCGACGGCCGCCAAGACGGA | |
| droWil2 | scf2_1100000004585:8373428-8373486 + | T-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTATTTAGGTTAAAGAGCCGCTCTACCCAATTGTTTGTTCAACAGCGGCTAAAACCGA | |
| dp5 | 4_group4:1560479-1560530 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGATTGAGGAACCTCTTTATCCCATCGTATGCTCGACAGCCGCCAAAACGGA | |
| droPer2 | scaffold_10:565086-565129 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAACCTCTCTATCCCATCGTATGCTCGACAGCCGCCAAAACGGA | |
| droAna3 | scaffold_12916:13671916-13671968 + | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGATTGAGGAGCCACTGTATCCCATTGTCTGCTCCACGGCAGCCAAAACAGA | |
| droBip1 | scf7180000396535:641747-641811 + | AT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTTCC-CTCACAGATTGATGAGCCACTGTATCCCATTGTCTGCTCCACGGCGGCCAAAACCGA | |
| droKik1 | scf7180000302271:164299-164496 + | TAATTTCTAAAAACTTAATGGTCAGCAAAAGTCAAAGAAAAAGGGGTCTT---ATTAACCCAAAACCTTG--------------------------TTTTATAAATA---TATATTTTATATATATATTATATTT---------------------------------------ATA-TATTTTCT-----T-AA------------AA--TTTAATTCATAATATTT------------AT--CCCTCTTCAGATTGACGAACCCCTGTACCCCATTGTGTGCTCCACGGCCGCCAAGACGGA | |
| droFic1 | scf7180000453842:1207569-1207626 - | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCTCCAGATTGATGAGCCTTTGTACCCCATTGTCTGCTCAACGGCGGCCAAAACGGA | |
| droEle1 | scf7180000491273:189265-189459 - | TTAT---------------------------------------------------------------------------------------------CTGATGGAAGTGATGATCTTG---ATTATGTTATATCTTTTAATTAATAATTGATCAGTAATTGATTGTTTCGGAACAGA-TATT-ACCCTTCTT-ATACATTTAAAAAAAGTCCTTTATTCATATTAACT--------AAATCC--TCCTCTCCAGATCGATGAACCGCTGTACCCCATTGTATGCTCCACGGCGGCCAAAACGGA | |
| droRho1 | scf7180000777055:112522-112578 + | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCCAGATTGATGAACCGCTGTACCCAATCGTATGTTCCACGGCCGCCAAAACGGA | |
| droBia1 | scf7180000302261:469285-469337 + | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGATCGATGAGCCCCTGTACCCCATTGTGTGTTCCACGGCCGCCAAGACGGA | |
| droTak1 | scf7180000415204:107278-107334 - | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCCAGATTGATGAGCCCCTGTATCCCATTGTCTGCTCCACGGCGGCCAAAACGGA | |
| droEug1 | scf7180000409005:924476-924528 + | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGATTGATGAACCCCTATATCCCATCGTGTGCTCTACGGCTGCCAAGACAGA | |
| dm3 | chr2L:5039806-5039858 + | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGATTGATGAGCCCCTGTACCCCATCGTATGCTCTACTGCCGCCAAAACGGA | |
| droSim2 | 2l:4853134-4853186 + | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGATTGATGAGCCCCTGTACCCCATCGTATGCTCTACTGCCGCCAAAACGGA | |
| droSec2 | scaffold_5:3126326-3126378 + | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGATTGATGAGCCCCTGTACCCCATCGTATGTTCTACTGCCGCCAAAACGGA | |
| droYak3 | 2L:10485770-10485828 - | T-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCCTTCAGATTGATGAGCCCTTGTACCCCATTGTATGCTCTACTGCCGCCAAAACGGA | |
| droEre2 | scaffold_4929:5118902-5118957 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCCAGATTGATGAGCCCCTGTACCCCATCGTATGCTCTACTGCCGCGAAAACGGA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:21 AM