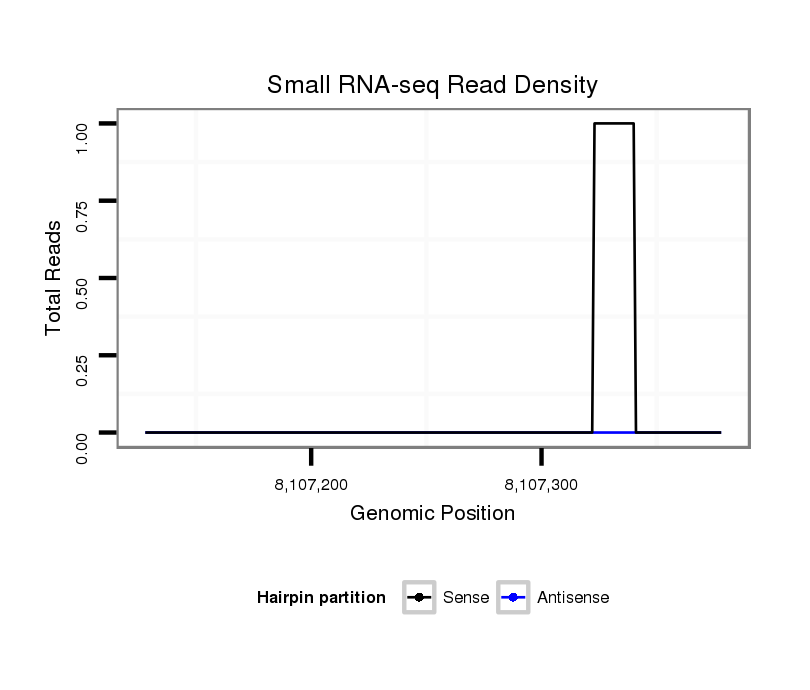
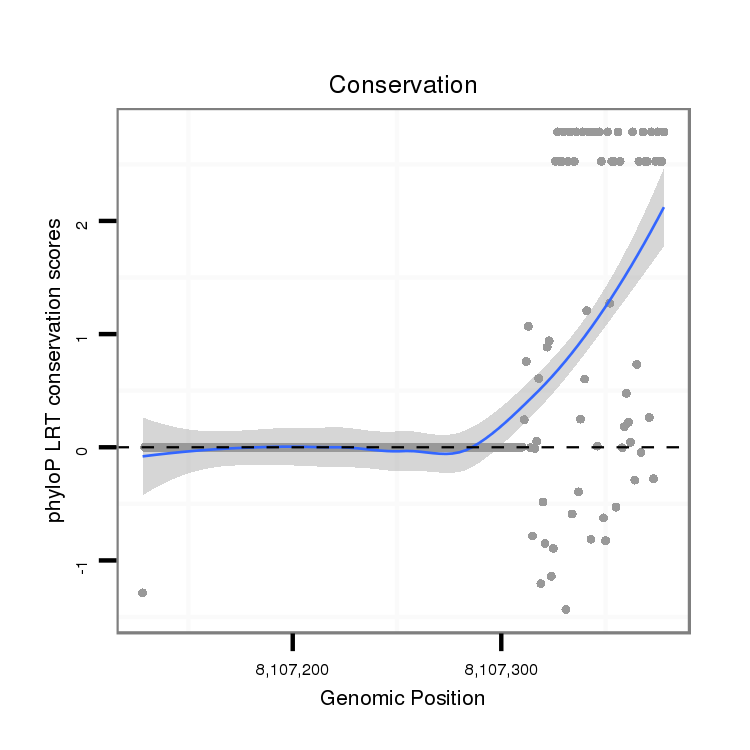
ID:dvi_10866 |
Coordinate:scaffold_12963:8107178-8107328 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_1792:5]; CDS [Dvir\GJ17183-cds]; intron [Dvir\GJ17183-in]
| Name | Class | Family | Strand |
| AT_rich | Low_complexity | Low_complexity | + |
| AT_rich | Low_complexity | Low_complexity | + |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CAATTAAACAATTTGCAAGAAGTTCAGAATTATGCAGAGTTATGCAATTCAGCGAATCCTGCAGTCGACAGACTATTATTTACTTGATGGCATACTAACTTTGCTTAACTGAATGAAAATATAAATATTATAATTATGTTCTGAGGGAAATAGGACTAATTATAATAAATTGAATTAATAATATTGATTGTTTGTTGGCAGCTGCATGACCTGCTCTACTCGTTGGCCTCGAGCGTCTCCTGGCTGCTGCT **************************************************.(((.....))).((((((((((..(((((((.(((..((((.........))))))).))))))).........(((((((((((.((((((.......))))))))))))))))).....(((((((...)))))))))))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
M028 head |
SRR060689 160x9_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060655 9x160_testes_total |
V047 embryo |
SRR060660 Argentina_ovaries_total |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................................................................................................................................................................................TGGCAGCTTCGTTACCTGCTCT.................................. | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................TGGCAGCTTCATTACCTGCTCT.................................. | 22 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................GTTCAGAATTATGTGGAGTAATG............................................................................................................................................................................................................... | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................TGGCAGCTGCATGACCTG...................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................TAGCAGCTTCATTACCTGCTCT.................................. | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................TGGCAGCTTCATACCCTGCTC................................... | 21 | 3 | 4 | 0.75 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................TGGCAGCTTCATACCCTGCTCT.................................. | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................TGGCAACTGCATTACCTGGTCT.................................. | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................TGGCAGCTTCATTCCCTGCTCT.................................. | 22 | 3 | 5 | 0.40 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................TAGCAGCTTCATAACCTGCTCT.................................. | 22 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................CTGAGGGAGATTGGACTACT........................................................................................... | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................................GCTCTAGCGTCTCCTGACT...... | 19 | 3 | 14 | 0.21 | 3 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................GCTTCATGACCTGCTCTC................................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................................................................................................................GAAGTGATAATATTGATGGTT........................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................GGTATGCAATTGAGCGAA................................................................................................................................................................................................... | 18 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GTTAATTTGTTAAACGTTCTTCAAGTCTTAATACGTCTCAATACGTTAAGTCGCTTAGGACGTCAGCTGTCTGATAATAAATGAACTACCGTATGATTGAAACGAATTGACTTACTTTTATATTTATAATATTAATACAAGACTCCCTTTATCCTGATTAATATTATTTAACTTAATTATTATAACTAACAAACAACCGTCGACGTACTGGACGAGATGAGCAACCGGAGCTCGCAGAGGACCGACGACGA
**************************************************.(((.....))).((((((((((..(((((((.(((..((((.........))))))).))))))).........(((((((((((.((((((.......))))))))))))))))).....(((((((...)))))))))))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060677 Argx9_ovaries_total |
V053 head |
SRR060672 9x160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
M047 female body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......CTGTTAAACGTTCTTCAAGTCTTAAT........................................................................................................................................................................................................................... | 26 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................TGGACGATATGAGCAACCGC....................... | 20 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................GAACTACCCTATGATAGA....................................................................................................................................................... | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................GTGGACGTACTGGACGAGG.................................. | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........TAAACGTTATTCAGGTCTT.............................................................................................................................................................................................................................. | 19 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................................AAGTCGCTTTGGCCGTCCGC........................................................................................................................................................................................ | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................................................ACCATGATTAACATTATTTA................................................................................. | 20 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 2 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:8107128-8107378 + | dvi_10866 | CAATTAAACAATTTGCAAGAAGTTCAGAATTATGCAGAGTTATGCAATTCAGCGAATCCTGCAGTCGACAGACTATTATTTACTTGATGGCATACTAACTTTGCTTAACTGAATGAAAATATAAATATTATAATTATGTTCTGAGGGAAATAGGACTAATTATAATAAATTGAATTAATAATATTGAT----------------------------------------T----------------------------GTTTGTTGGCAGCTGCATGACCTGCTCTACTCGTTGGCCTCGAGCGTCTCCTGGCTGCTGCT |
| droMoj3 | scaffold_6500:729801-729865 + | A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T--------------------------------------------T----------------------------GGATTTTCACAGCTGCACGACTTGCTATATTCAGTGGCCTCGGGCGTTTCTTGGCTGCTGCT | |
| droGri2 | scaffold_15252:7535776-7535843 + | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T-GGT----------------------------------------T----------------------------GTCTATTGACAGCTGCACGACTTGCTTTATTCAATGGCATCGTGCATATCCTGGCTGCTGCT | |
| droWil2 | scf2_1100000004945:767355-767418 + | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T-------------------------------------------------------------------------CTTTTTCTACAGCTTCACGATGTTCTTTACTCATTGGCGTCTGCCGTTTCCTGGTTGCTGCT | |
| dp5 | 4_group3:6267063-6267115 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTTCATGATGTGCTCTACTCGCTGGCCTCTGCGTTCTCCTGGCTGCTGCT | |
| droPer2 | scaffold_1:7759469-7759521 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTTCATGATGTGCTCTACTCGCTGGCCTCTGCGTTCTCCTGGCTGCTGCT | |
| droAna3 | scaffold_12916:13414311-13414393 - | A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A---------------------------------------TCTATAAATTCATTTTT--TTTT-------------AATATTTCAGCTCCACGATTTGCTCTACTCACTAGCCTCCAGCTTTGCCTGGCTCCTGCT | |
| droBip1 | scf7180000396580:45598-45650 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTACACGACTTGCTGTACTCACTAGCCTCAAGCTTTGCATGGCTCCTGCT | |
| droKik1 | scf7180000302679:1523889-1523941 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTGCATGACTTACTGTATTCCCTGGCATCTGCCTTCTCCTGGCTGCTGCT | |
| droFic1 | scf7180000453924:1300277-1300337 + | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTGTTGACAGCTGCACGATTTGCTGTACTCGCTGGCTTCTGCCGTTGCCTGGTTGCTGCT | |
| droEle1 | scf7180000491046:2021417-2021480 + | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T-------------------------------------------------------------------------ATCCTTTGGCAGCTGCACGATTTGCTGTACTCAGTGGCTTCTGCCGTTTCGTGGCTTCTGCT | |
| droRho1 | scf7180000777227:161140-161196 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGGCAGCTCCACGATTTGCTGTACTCACTGGCATCTGCCGTTTCCTGGCTACTGCT | |
| droBia1 | scf7180000301468:1213910-1213962 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTCCATGATTTGCTGTACTCGCTGGCATCTGCTGTTTCCTGGCTGCTGCT | |
| droTak1 | scf7180000413924:29830-29957 - | C--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T-GAGTGGAATGATTAATATTATACGAGGGCATATGAAAATTAATCCC--------GCTTTTGAATGTTCGGTAATCCTTTTGCAGCTCCATGACTTGCTGTACTCCCTGGCATCTGCTGTTTCCTGGTTGCTGCT | |
| droEug1 | scf7180000409554:2776344-2776415 - | A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A---------------------------------------TTAATTAC--------------------------ATTATTTTCCAGCTTCATGATTTGCTGTATTCATTGGCATCTGCTGTTTCCTGGCTGCTGCT | |
| dm3 | chr2L:3322688-3322740 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTCCACGACTTGCTCTACTCATTGGCATCCGCCGTTTCGTGGCTTCTGCT | |
| droSim2 | 2l:3182679-3182731 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTCCACGACTTGCTGTACTCACTGGCATCCGCTGTTTCGTGGCTTCTGCT | |
| droSec2 | scaffold_5:1465287-1465339 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTCCACGACTTGCTGTACTCACTGGCATCCGCTGTTTCGTGGCTTCTGCT | |
| droYak3 | 2L:3307157-3307209 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGCTCCATGACTTGTTGTACTCACTGGCATCTGCTGTATCGTGGCTCCTGCT | |
| droEre2 | scaffold_4929:3353374-3353444 + | A--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGAC----------------------------------------TGC--------------------------ATTTCTTGGCAGCTCCACGACTTGTTGTACTCACTGGCATCTGCTGTTTCGTGGCTCCTGCT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:35 PM