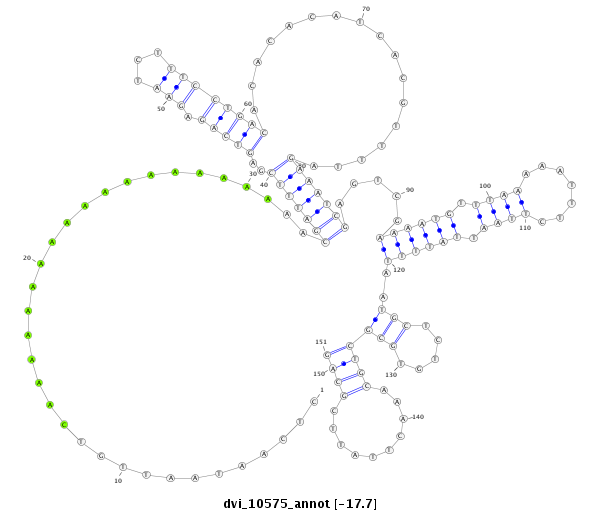
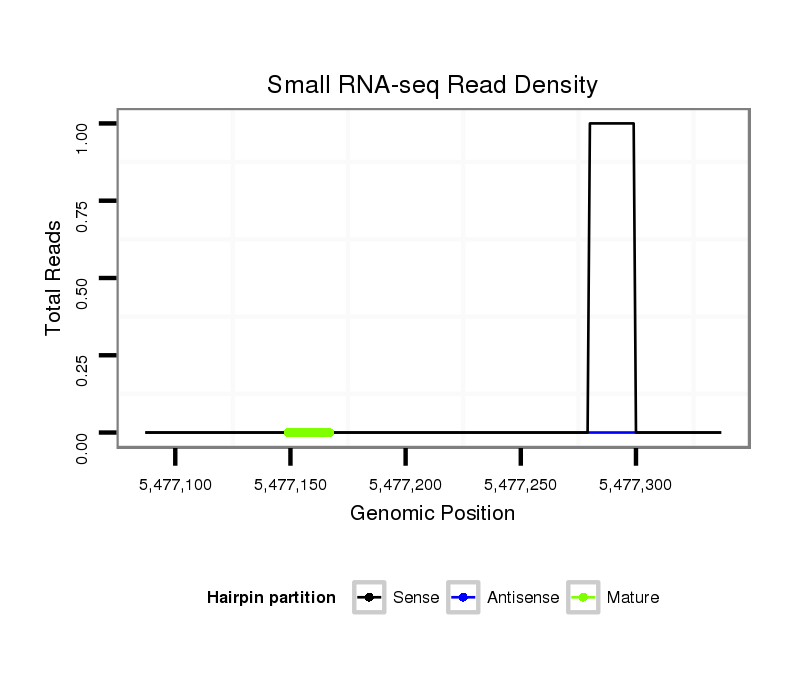
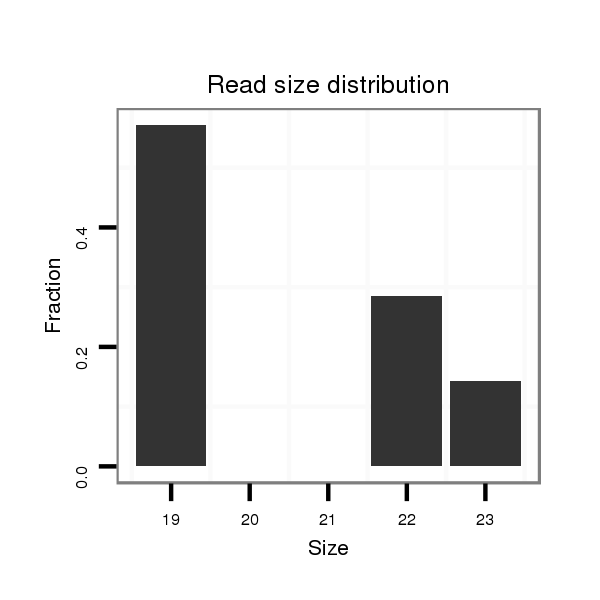
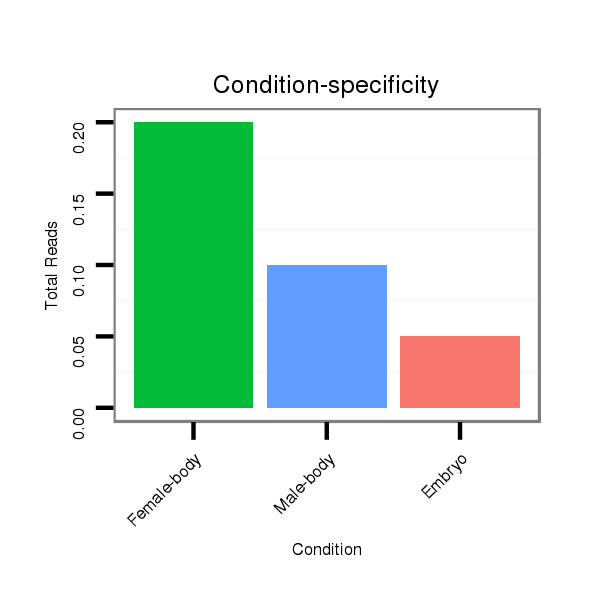
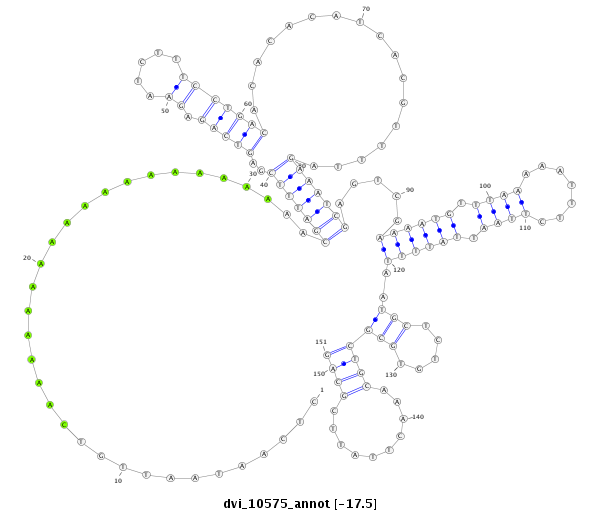
ID:dvi_10575 |
Coordinate:scaffold_12963:5477137-5477287 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
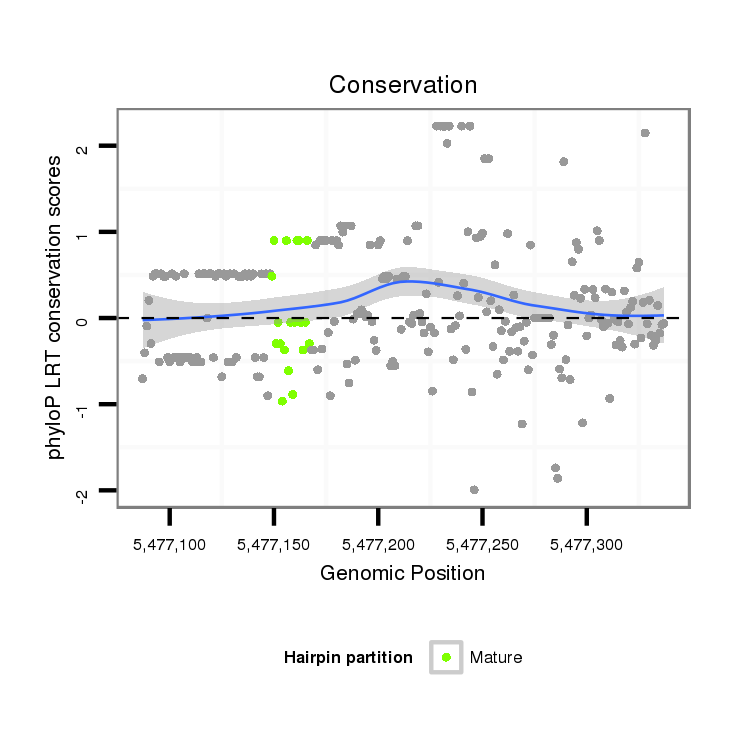
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -17.5 | -17.5 | -17.3 |
 |
 |
 |
exon [dvir_GLEANR_1687:2]; CDS [Dvir\GJ16363-cds]; intron [Dvir\GJ16363-in]
| Name | Class | Family | Strand |
| (A)n | Simple_repeat | Simple_repeat | + |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AAAATCATATCCGTTTCTTCAGAAATCAAATAATGCTTTTCTAAAACCGTCTCAATAATTGTCAAAAAAAAAAAAAAAAAAAACGATTTCGAGTCAGAGAATCTTTCCTGACACACACATCACGTTTTAGAAATCGAGTCGAAAATGTTTAAAAATTTCTTAATTATTTTAATGCTCTGTGCGCTGCAAACTTATTCGCAGTCTGTGGAGCTGTCCAAGGGGCTGGGCGCAAAAGAGAATGAAGCCAGCAA **************************************************.................................(((((((..(((((.(((...)))))))).................))))))).....((((((.((((.......)))).))))))..(((.....)))((((..........))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
V116 male body |
SRR060655 9x160_testes_total |
SRR060680 9xArg_testes_total |
SRR060681 Argx9_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060675 140x9_ovaries_total |
SRR060686 Argx9_0-2h_embryos_total |
M047 female body |
SRR060666 160_males_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060678 9x140_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................................................................................................TAATTATTTTAATGCTCTGTGCGCTCC................................................................ | 27 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................GCAAAAAAGAATGAAGCCAGC.. | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................TCTCAGTCTGTGGAGCTG...................................... | 18 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................ATTCGCAGTCTGTGGAGCTG...................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................TGCTCTGTGCGCTCCAAACTTATTCGC.................................................... | 27 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................AATCATTGTCGCAAAAAAAAAAAAAAA........................................................................................................................................................................... | 27 | 3 | 5 | 0.80 | 4 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................ATAGTTGTCAAAAAAAAAAAA................................................................................................................................................................................ | 21 | 1 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................TAATCATTGTCGAAAAAAAAAAAAAAAAA.......................................................................................................................................................................... | 29 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................AATCATTGTCGCAAAAAAAAAAAAA............................................................................................................................................................................. | 25 | 3 | 14 | 0.29 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 2 | 0 | 0 | 0 | 0 | 0 |
| .....................................................AATGATTGTCAAAAAAAAA................................................................................................................................................................................... | 19 | 1 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................CGAGCGGTCCAAGGGCCTG.......................... | 19 | 3 | 20 | 0.20 | 4 | 0 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................AATAAATGTCAAAAAAAAAAAAAAAA............................................................................................................................................................................ | 26 | 1 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................CAAAAAAAAAAAAAAAAAA.......................................................................................................................................................................... | 19 | 0 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 |
| .....................................................AATCATTGTCGCAAAAAAAAAAAAAA............................................................................................................................................................................ | 26 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................AATCATTGTCGAAAAAAAAAAA................................................................................................................................................................................ | 22 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................GTCAAAAAAAAAAAAAAAAAAA......................................................................................................................................................................... | 22 | 0 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................ATAATTGTGAAAAAAAAAAA................................................................................................................................................................................. | 20 | 1 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................TAATGGTCGAAAAAACAAAAA............................................................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ............................................................GTCAAAAAAAAAAAAAAAA............................................................................................................................................................................ | 19 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................AATGGTCAAAAAAAAAAAAAAAA............................................................................................................................................................................ | 23 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................................................GTCAAAAAAAAAAAAAAAAAAAA........................................................................................................................................................................ | 23 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
TTTTAGTATAGGCAAAGAAGTCTTTAGTTTATTACGAAAAGATTTTGGCAGAGTTATTAACAGTTTTTTTTTTTTTTTTTTTTGCTAAAGCTCAGTCTCTTAGAAAGGACTGTGTGTGTAGTGCAAAATCTTTAGCTCAGCTTTTACAAATTTTTAAAGAATTAATAAAATTACGAGACACGCGACGTTTGAATAAGCGTCAGACACCTCGACAGGTTCCCCGACCCGCGTTTTCTCTTACTTCGGTCGTT
**************************************************.................................(((((((..(((((.(((...)))))))).................))))))).....((((((.((((.......)))).))))))..(((.....)))((((..........))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
V053 head |
M027 male body |
SRR060683 160_testes_total |
|---|---|---|---|---|---|---|---|---|---|
| ..........................GTTTGTTACGAAAAGATTTG............................................................................................................................................................................................................. | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................GACGTTTGAAAAAGCGTCAGGT.............................................. | 22 | 3 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................................................................................GTCAGACACCTCGACGTG................................... | 18 | 2 | 13 | 0.08 | 1 | 0 | 0 | 1 | 0 |
| ................................................................................................................................................................................................................................CCCGCGTTGTCCCTAACTT........ | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:5477087-5477337 + | dvi_10575 | AAAATCATATCCGTTTCTTCAGAAATCAAATAATGCTTT-------TCTAAAACC-GTCTCAATAATTGTCAAAAAAAAAAAAAAAAAAAACGAT-----------------------------------------------------TTCGAGTCAGAGAATCTTTCCTGACACACACATCACGTTTTAGAAATCGA-G---TCGAAAATGTTTAAAAATTTCTTAATTATTTTAATGCTCTGTGCGC-----TGCAAACTTATTC-GCA---------GTCTGTGGAGCTGTCCAAGGGGCTGGG----------------CGCAAAAGAGAATGAAGCCAGCAA- |
| droMoj3 | scaffold_6500:17128155-17128305 - | AGAC--------------------------------------------------------------------------------------------------------------------------------------------------------AGATCATCCCTTCTGACTCACACAATTCGTTTTAGAAAACAT-G---TCGAAAATGTTGAACAAATTTGTAATTATTCTGATGGTCTCTACAC-----------------TCGCC---------AGCAATCAAGTCAC-TGCTGCTCGTGC---TCCTGCTCCTGCTCCTGCTGC----------TGGCCC- | |
| droGri2 | scaffold_15252:8094468-8094750 + | GCAACCATTTCCAATCCCATAAGGTATATAT-ATACTTGATCAACATCAATAGCCGGTCTCAGGCACTCTCAGAGCTACAGAAAATAAGTTCTAATATGCCATAGACATAGTATGCTTTGACGCACTCAAATATTCTGTTGACTTTCGTTTCAGTCAGAGAAGTTTTCCT--------------------GAAATCAAAT---CGAAAAATGTTTAAAAAATTTCTAACTTTTCTTGGGCTCTTTGCTT-----TTCC----------AAAACTTTTGCTATCT--------GA-CGATAATTTCAG----------------GGAGCTGAGCACTCACAGCGCTAC- | |
| droWil2 | scf2_1100000004884:272003-272164 - | TTTTT------------------------------------------------------------------AATAGAAATCTAATATAAAACTAA-----------------------------------------------------TTC-AATCAGAACATATTCCAGGTGACA---------G--TTCCAATAAA-T---TCAAAAATGTTTAAAATTTTTGGAAT---GGTGGCATATTTGTGTCTGTGCATCC----------AAA-------------ATGTAGGTGC-TAATGATCTAGT----------------CACACA-------AAGGCAAATGA- | |
| dp5 | 3:17351788-17351896 - | AATT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AA-G---TAGAAAATGTGGTTTAAGTTGCTTATTATTGCAACGTTCGTTGCGT-----------------TGAGC---------AGCTGCGAAATCGT-TATTGGCCAGGA----------------CGAGCTGGTGGATGAGGTCTCAGGA | |
| droPer2 | scaffold_31:438727-438834 + | AATT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AA-G---TAGAAAATGTGGTTTAAGTTGCTTATTATTGCAACGTTCGTTGCGT-----------------TGAGC---------AGCTGCGAAATCGT-TATTGGCCAGGA----------------CGAGCTGGTGGATGAGGTCTCAGG- | |
| droAna3 | scaffold_13266:15487803-15487910 + | AATT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GA-T---CCAAAAATGTGCCTAAAGTTTATTGTTTTTGCGGCTCTTATTTATT-----------------CAGGG---------GCCGGCGAAGTGGT-CATTGGCCAGGA----------------CGAGCTAGTGGACGAGGTCTCTGG- | |
| droBip1 | scf7180000396547:1559672-1559779 - | AATT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GA-T---CCGAAAATGTGCCTAAAGTTTATTATTTTTGCGGCTCTTATTTATT-----------------TAGGG---------GCCAGTGAAGTGAT-CATTGGCCAGGA----------------TGAGCTAGTAGATGAGGTCTCTGG- | |
| droEle1 | scf7180000491107:907427-907470 - | G----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGTGGT-CATTGGCCAGGA----------------TGAACTGGTGGACGAGGTCTCCGG- | |
| droRho1 | scf7180000779911:195822-195931 + | AAAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGGGC---CGAAAAATGTGCTTAAAGTTCGTTATTTTCGCCGCCCTGTTGGCCC-----------------TAGCC---------ACCAGTGAGGTTGT-CATTGGCCAAGA----------------CGAACTGGTGGACGAAGTCTCCGG- | |
| droBia1 | scf7180000301506:2842944-2843045 - | G----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCAATAATGTTCTTGAAGTTACTTATTTTCACGGCGCTCCTCGCCC-----------------TGACC---------AGCTGCGAAGTTGT-CATTGGCCAGGA----------------TGAACTGGTGGACGAGGTCTCCGG- | |
| droTak1 | scf7180000415795:659681-659791 + | AAAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGGAC---TGAAGAATGTGCTTAAAGTTACTTATTTTCACGGGTCTCCTCGCCC-----------------TGGTG---------GCCTCCGAAGTTGT-CATTGGCCAGGA----------------TGAGCTGGTGGACGAGGTGTCCGGC | |
| droEug1 | scf7180000409752:286252-286362 - | TTTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AG-CCTTCGAAAAATGTTTAAAATTATTGTGAT---------TCTAT--TAAT-----------------AGGAA---------GCCTGGGAAGTGGC-C-TTGGTAGCCACGAACTTGTACC----TCGGAAAGTTGAAGAGCCAGCCCG- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/19/2015 at 04:25 PM