
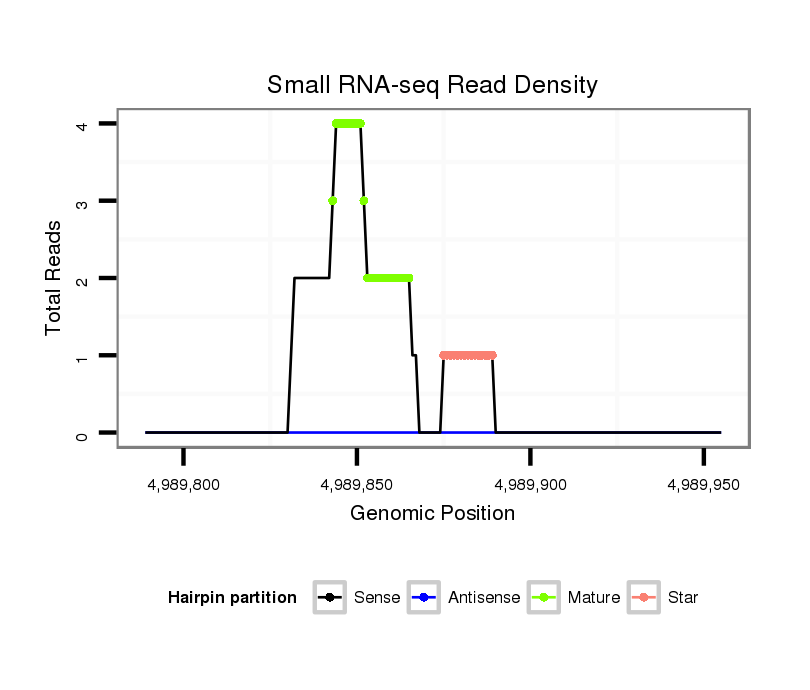
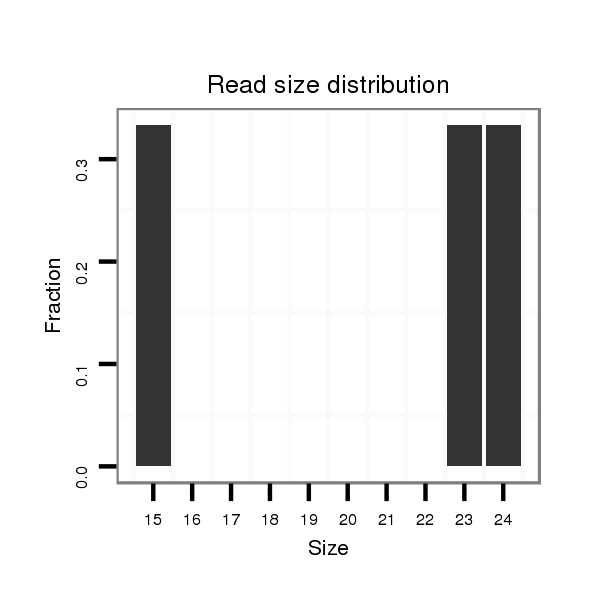
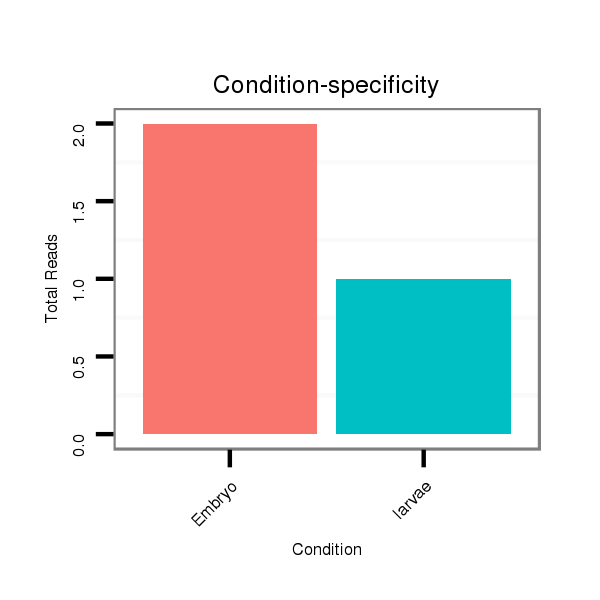
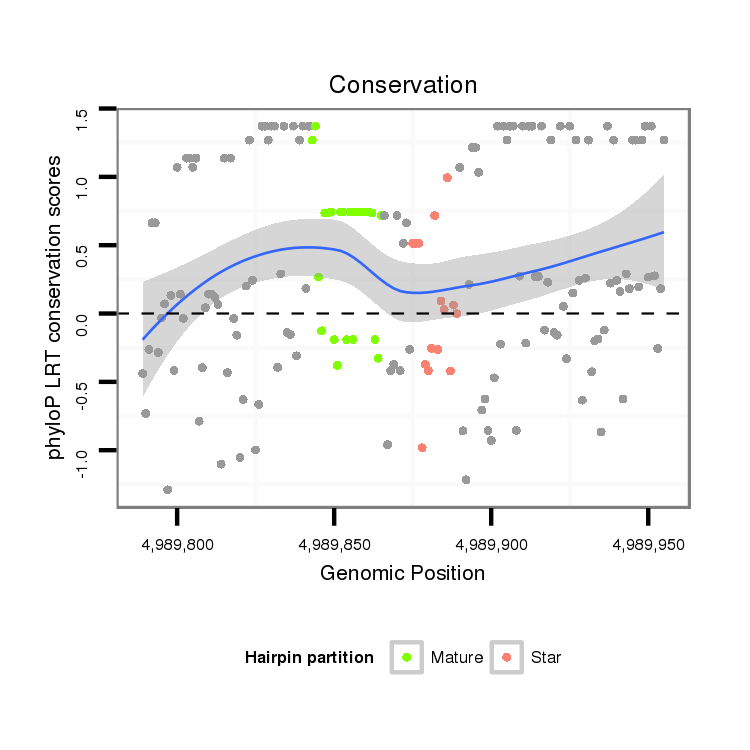
ID:dvi_10523 |
Coordinate:scaffold_12963:4989839-4989905 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -1.8 | -1.8 | -1.7 | -1.5 |
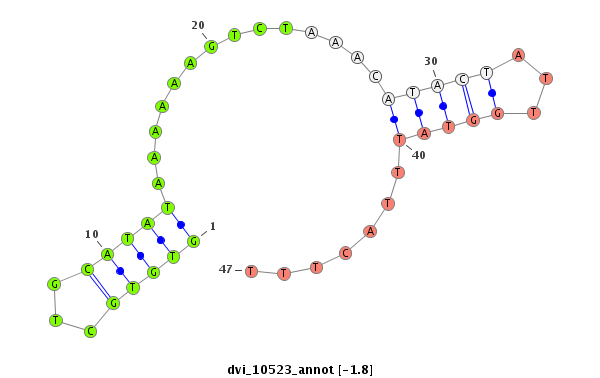 |
 |
 |
 |
CDS [Dvir\GJ16100-cds]; exon [dvir_GLEANR_1653:1]; exon [dvir_GLEANR_1653:2]; CDS [Dvir\GJ16100-cds]; intron [Dvir\GJ16100-in]
No Repeatable elements found
| --------------------------------------############-------------------------------------------------------------------################################################## GCAGCACAGTTGAGTTCAGAGTTGAAAGAGCAGTCGCTATGAAGTTGCTGGTGAGTGTGCTGCATATAAAAAAGTCTAAACATACTATTGGTATTTACTTTTGAATTTCGCGGACAGATCATTTTATTTGCGCTATTTGCTGCGGTCTTGGCAGTGCCGCAGTACGG ******************************************************.............((............(((((...))))).....))****************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M061 embryo |
SRR060664 9_males_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR1106728 larvae |
V116 male body |
SRR060660 Argentina_ovaries_total |
SRR060659 Argentina_testes_total |
M047 female body |
SRR060672 9x160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................GTGTGCTGCATATAAAAAAGTCT.......................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................GTTGCTGGTGAGTGTGCTGC........................................................................................................ | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................TGTGCTGCATATAAAAAAGTCTAA........................................................................................ | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................AGTTGCTGGTGAGTGTGCTGCA....................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................GCTATGAAGGTGCTGGTG.................................................................................................................. | 18 | 1 | 3 | 1.00 | 3 | 0 | 0 | 0 | 0 | 0 | 2 | 1 | 0 | 0 | 0 |
| ......................................................................................ATTGGTATTTACTTT.................................................................. | 15 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................GTCGCTATGAAGCTGCTGCA................................................................................................................... | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................GTTGGTATTTACGTTTGAG.............................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ........................................................................................................................GTTTTATTTGCGCTACTTTC........................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
CGTCGTGTCAACTCAAGTCTCAACTTTCTCGTCAGCGATACTTCAACGACCACTCACACGACGTATATTTTTTCAGATTTGTATGATAACCATAAATGAAAACTTAAAGCGCCTGTCTAGTAAAATAAACGCGATAAACGACGCCAGAACCGTCACGGCGTCATGCC
******************************************************************.............((............(((((...))))).....))****************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
|---|---|---|---|---|---|---|
| ......................................................................................................................................TAGACCACGCCAGAACCGCC............. | 20 | 3 | 5 | 0.20 | 1 | 1 |
| .................TTTCAACTTTCTCGACAG.................................................................................................................................... | 18 | 2 | 19 | 0.05 | 1 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:4989789-4989955 + | dvi_10523 | GCAGCACAGTTGAGTTCAGAGTTGAAAGAGCAGTCGCTATGAAGTTGCTGGTGAGTGT------------------------GCTGCATATAAAAAAGT------------------------------------CTAAA--CATACTATTGGTAT-TT---ACTTTTGAATTTCGCGGACAGATCATTTTATTTGCGCTATTTG---------CTGCGGTCTTGGCAGTGCCGCAG---------------------------TACGG |
| droMoj3 | scaffold_6500:8884300-8884484 + | AGTGCGCTCTAGAGTTCATTGTTGGCAGAGTCGTCGTCATGAAACTGTTGGTCAGTATAAT---------------ATTTTG--------------------------GTGGAGTATAT-----------AGAGTATATTGCTAA-CA---AAAGTTATATTTCAATTATATT-TGAATATAGATAATATTATTTTCGTTACTTGCGGTTTTCGCCACTTCATTGGCAGCGCCCCAA---------------------------TATGG | |
| droGri2 | scaffold_15252:480897-481085 + | ACAGCGGATTTGAGTTCATAGTTGAGAAAGCATTCGATATGAAATTGTTTGTGAGTAT---------------TCTTGTTCT--------------------------GCAGAGGCTCTCGGTCTCTGTAAGAGGCTAGTTGTAAACTATTTAAATATTATTTCTTGTTCA---TTTTGACAGATTATATTATTTGCGCTGTTTG---------CGGCTGCTTTTGCATTGCCCCAA---------------------------TTTGG | |
| dp5 | 4_group1:3039047-3039179 - | dps_1387 | AG----------------------------CCAACGACATGAAGTTTCTGGTGAGTAGATCGGAAAACTGAATTCTTGAACA--------------------------GCAA----------------------------------------------AAACTCATTTATATTTCTTCTATAGATTATTTTATTCGCTCTGCTGG---------CCGCTGCCCTGGCCTTGCCCCAA---------------------------TATGG |
| droPer2 | scaffold_5:2181556-2181688 + | AG----------------------------CCAACGACATGAAGTTTCTGGTGAGTAGATCGGAAAACTGAATTCTTGAACA--------------------------GCAA----------------------------------------------AAACTCATTTATATTTCTTCTATAGATTATTTTATTCGCTCTGCTGG---------CCGCTGCCCTGGCCTTGCCCCAA---------------------------TATGG | |
| droBia1 | scf7180000302292:1124770-1124952 - | CG----CAGACGGCTTCAGTTCCAAAATACTGTTCATAATGAAATTACTTGTGAGTAAATTAG---------------T---GCTAAATGTGAAAAAGCATCCGTAACGTAAAG-------------------------------------------------------GCTTTACCCTATAGATCGTTTTTATAGCATTGTTTG---------TCGCCATTATGGTCCTTCCCCAAGACGCACTATGTAGACATCATCATCACTACTG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
Generated: 05/17/2015 at 03:55 AM