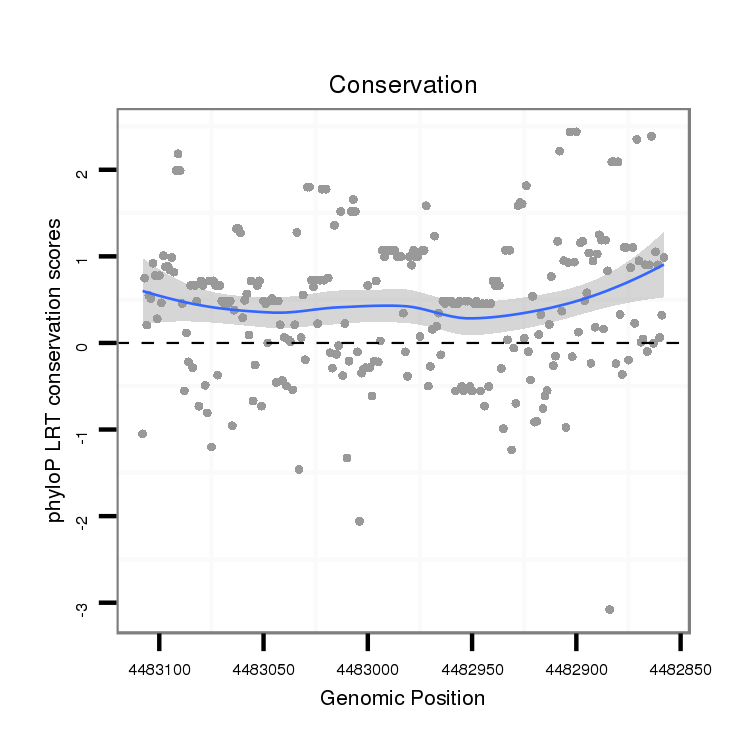
ID:dvi_10478 |
Coordinate:scaffold_12963:4482908-4483058 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_1194:2]; CDS [Dvir\GJ11932-cds]; intron [Dvir\GJ11932-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GTTTTACGCATGGGTGCAGCTAAGTCAAAGAGAGAGGGAGAGAGAGCAGCACACACACACGCACGCATATGCATACACGTTAGAGAGAGAGCGCGCAATTGAGTTGAACAGTTGAACAAAATGGCCGCCAAGCCAATGAGCAGCACATACTCATAAACAGACACCCGCTCACCTTCTGCTTCTCTCTCGCACATTTTGCAGGCACACGAGAAGACACACAAATCACCGCGCTACGAATGCTCCGACTGTGG **************************************************...................((...........(((((((((((((....(((((......((((..((...((((......)))).))..))))....)))))............))).........))).)))))))..........)).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060684 140x9_0-2h_embryos_total |
M047 female body |
V116 male body |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................................CCAAGCCAAAGAGCAGCACAAAA..................................................................................................... | 23 | 3 | 2 | 1.00 | 2 | 2 | 0 | 0 | 0 |
| .....................AAGTCATAGAGAGAGGGAGAG................................................................................................................................................................................................................. | 21 | 1 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 |
| ..............................................................................................................................ACCAAGCCAAAGAGCAGCAC......................................................................................................... | 20 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 |
| ............GGTGCAGCTACGTCAACGA............................................................................................................................................................................................................................ | 19 | 2 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 |
| .......GCATGGGTACAGCAAAGTCGA............................................................................................................................................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 1 | 0 | 0 |
| ..............................................................................................................................ACCAAGCCAAAGAGCAGCACAA....................................................................................................... | 22 | 3 | 10 | 0.10 | 1 | 1 | 0 | 0 | 0 |
|
CAAAATGCGTACCCACGTCGATTCAGTTTCTCTCTCCCTCTCTCTCGTCGTGTGTGTGTGCGTGCGTATACGTATGTGCAATCTCTCTCTCGCGCGTTAACTCAACTTGTCAACTTGTTTTACCGGCGGTTCGGTTACTCGTCGTGTATGAGTATTTGTCTGTGGGCGAGTGGAAGACGAAGAGAGAGCGTGTAAAACGTCCGTGTGCTCTTCTGTGTGTTTAGTGGCGCGATGCTTACGAGGCTGACACC
**************************************************...................((...........(((((((((((((....(((((......((((..((...((((......)))).))..))))....)))))............))).........))).)))))))..........)).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
SRR060686 Argx9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
M047 female body |
SRR060662 9x160_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
SRR060675 140x9_ovaries_total |
V053 head |
SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
V047 embryo |
SRR060680 9xArg_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060658 140_ovaries_total |
SRR060689 160x9_testes_total |
SRR060674 9x140_ovaries_total |
SRR060677 Argx9_ovaries_total |
M027 male body |
SRR060669 160x9_females_carcasses_total |
SRR1106727 larvae |
SRR1106729 mixed whole adult body |
SRR060664 9_males_carcasses_total |
SRR060673 9_ovaries_total |
SRR060661 160x9_0-2h_embryos_total |
M061 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................................CGAGTGGAATCCTAAGAGAG................................................................. | 20 | 3 | 2 | 81.50 | 163 | 56 | 28 | 28 | 14 | 7 | 7 | 3 | 5 | 4 | 0 | 1 | 1 | 0 | 1 | 2 | 1 | 0 | 2 | 0 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................CGAGTGGAATCCTAAGAGA.................................................................. | 19 | 3 | 5 | 25.60 | 128 | 33 | 23 | 24 | 11 | 9 | 9 | 3 | 5 | 2 | 0 | 4 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................CGAGTGGAATCCTAAGAG................................................................... | 18 | 3 | 20 | 15.85 | 317 | 3 | 71 | 92 | 45 | 29 | 13 | 20 | 10 | 12 | 0 | 4 | 0 | 0 | 3 | 1 | 7 | 0 | 1 | 0 | 2 | 0 | 0 | 0 | 1 | 0 | 1 | 2 | 0 |
| .....................................................................................................................................................................GCGAGTGGAATCCTAAGAGA.................................................................. | 20 | 3 | 1 | 12.00 | 12 | 4 | 3 | 3 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................GCGAGTGGAATCCTAAGAGAG................................................................. | 21 | 3 | 1 | 10.00 | 10 | 5 | 2 | 1 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................GGTTCGGTTTCTCGTCGTGTTT...................................................................................................... | 22 | 2 | 1 | 7.00 | 7 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................GCGAGTGGAATCCTAAGAG................................................................... | 19 | 3 | 8 | 5.25 | 42 | 7 | 9 | 13 | 7 | 1 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................GGTTCGGTTTCTCGTCGTGTTTT..................................................................................................... | 23 | 3 | 2 | 3.50 | 7 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................TGGTTCGGTTTCTCGTCGTGTTT...................................................................................................... | 23 | 3 | 2 | 1.50 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................GTGGAATCCTAAGAGAGAGC.............................................................. | 20 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................TGGGCGAGTGGAATCCTAAGAGA.................................................................. | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................GAGTGGAATCCTAAGAGAG................................................................. | 19 | 3 | 11 | 0.82 | 9 | 3 | 2 | 0 | 1 | 0 | 0 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................GTTCGGTTTCTCGTCGTGTTT...................................................................................................... | 21 | 2 | 4 | 0.75 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................GGTTCGGTTTCTCGTCGTGTT....................................................................................................... | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................TATACGTATGTGCAATAACT..................................................................................................................................................................... | 20 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................TTGTCAACTTGTTTTGCCGGT............................................................................................................................ | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................TGAGCGAGTGGAATCCGAAGAG................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................GCGAGTGGAATCCTAAGA.................................................................... | 18 | 3 | 20 | 0.35 | 7 | 0 | 2 | 0 | 0 | 0 | 0 | 3 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................TAGTGGCGTAATGCTTACG........... | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........ACCCAGGTCGACCCAGTTTCT............................................................................................................................................................................................................................ | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................TGTGGGCTAGTGGCAGATG........................................................................ | 19 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................GTGTGTGTGCGTGCGTAT...................................................................................................................................................................................... | 18 | 0 | 20 | 0.10 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................................................ACCGGCGGTTCGGTCAAT................................................................................................................ | 18 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................................................................................................TGAGTCTTTGTCTGTGTACGA.................................................................................. | 21 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................TGTGTGTGTGCGTGCGTA....................................................................................................................................................................................... | 18 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................CGTTTCTTTAGTGGCGCGA................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................................GTGTGTGTGTGCGTGCGTAT...................................................................................................................................................................................... | 20 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................TGTGTGTGTGCGTGCGTAT...................................................................................................................................................................................... | 19 | 0 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:4482858-4483108 - | dvi_10478 | GTTTTACGCATGGGTGCAGCTA-AGTCAAAGAGAGAGGGAGAG-AGA-------------GCAGCAC-----------ACACACACGCACGCATATGCATACACGTTAGAGA----------GAGAGCG--CGCAATTGAG---------------------------------------------TTG------A------------------------------------------------------------------------------ACAGTTGAACAAAATGGCCG------------------CCAAGCCAATGAGCAGC----------------------------ACATACTCATAAACAGACACCCGCTCACCTT--------------CTGCTTCTCTC----TCGCACATTTTGCAGGCACACGA--GAAGACACACAAATCACC-GCGCTACGAATGCTCCGACTGTGG |
| droMoj3 | scaffold_6500:8387013-8387296 - | GTTTTACGCATGGGTGCAGCAGCAGCCACAGCCAAAGAGAGAGCACA-------------GC-----------AACACAAGCAAAC------GAGAGTACAGCCGTTAGAAA----------GAGAG----CGCAATTGAG---------------------------------------------C---------------------------------------------------------------------------------------GCGCTTAAACAAAATGGCCG------------------TCAAGCCAATGAGCAGCATTGCTCAATGCTCCCTTTCCCACACACACATACACACTAATTGACTCACACTCACCATCTCTACCGTTCTCTCTCTCTCTCTC----TCTATCCCATTGTAGGCGCACGA--GAAGACACACAAATCGCC-GCGCTACGAATGCTCCGACTGTGG | |
| droGri2 | scaffold_15252:954768-954943 + | GTTTTACGCATGGGTGCAG--GCTGCC--AGCCACAGAG-------A-------------GCAACAC---G--AACACAAACA----CACACACAAGTGTA-GCATTAGAAA----------GAGAG----CGCAGTTGAG---------------------------------------------AGC-------------------------------------------------------------------------------------GCGGCTGTACAAAATGGCCG------------------CC-AGCCAATAGGCAG-----------------------------------------------------TCACAGT--------------TTTCATCTCTTTCTTCC-------ACTTAGCCAGAAGA--GTAGACA------------------------------------ | |
| droWil2 | scf2_1100000004577:3743197-3743370 + | GTTTTACGCATGGGTGCAG--AT-------------------------------------------A--------------------------------------------------------------------------------------------------------------------------------GACA---------------CACACACAGACACCT------------TGAAGTTGGCTAAC--------------------------AACAAAATGGCCTACAACACGCAAAGTAAACTCAAGCTAATA----------------------------------------------------------------T--------------CTGTTTCTTTT----TCTTTTGATTTGCAGAACCACGA--AAAGACTCACAAATCACC-GCGCTATGAATGTTCGGATTGCGG | |
| dp5 | 4_group1:2491732-2491920 - | GTTTTACGCATGGGTGCAG--GG-----------------------CA---------------GATAC-AGAT-----------------------------ACATTAGAAACAGCAAGAGAGAGAGGG-------------------------------------------------------------AGACAAGCAGTCTACGAAAGAGAT---AACGAGACCCCTACAAAATGGC-----------------------------------------------------CAAAAACAT--------------------TGAACGAT---------------------------------------------------------------------------ATTTCTCTC----CC-------TTTCAGGCGCACGA--GAAGACACACAAATCGCC-ACGTTACGAGTGCTCCGATTGCGG | |
| droPer2 | scaffold_5:2708528-2708716 + | GTTTTACGCATGGGTGCAG--GG-----------------------CA---------------GATAC-AGAT-----------------------------ACATTAGAAACAGCAAGAGAGAGAGGG-------------------------------------------------------------AGACAAGCAGTCTACGAAAGAGAT---AACGAGACCCCTACAAAATGGC-----------------------------------------------------CAAAAACAT--------------------TGAACGAT---------------------------------------------------------------------------ATTTCTCTC----CC-------TTTCAGGCGCACGA--GAAGACACACAAATCGCC-ACGTTACGAGTGCTCCGATTGCGG | |
| droAna3 | scaffold_12916:10770979-10771034 - | TT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAGGCTCATGA--AAAGACGCACAAATCGCC-CCGTTATGAGTGCTCGGACTGCGG | |
| droBip1 | scf7180000396572:434946-435001 - | TT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAGGCTCATGA--AAAGACACACAAATCCCC-CCGCTACGAGTGCTCGGACTGCGG | |
| droKik1 | scf7180000302441:2769655-2769893 - | AGGAC----AAGGGCACAG---------------------------------------------CTCACAATC----------------------------------CACAC----------GCGAATGCACACGGGAGAGCGACCCTTTAAGTGTACCATCTGCCCAAAGGCGTACACGCACGGTCCGACGCTAAGGA---------------------------TGCACATGCTGACCCATGAGACAAAGGCCCACAAGTGCTTGGATTGCG------------------AGAAGTCCT--------------------TTTACAGC---------------------------------------------------------------------------ATCTC-------------CAACCTGAGGGCCCACCAGCGATGGCATTCGGGCGACCGACCTTACCAGTGTCCCGACTGCCC | |
| droFic1 | scf7180000453316:168355-168427 + | TGTAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCTT----TCAA--AAATTTTAGGCTCATGA--AAAAACGCACAAGTCTCC-ACGTTATGAGTGCGCGGACTGCGG | |
| droEle1 | scf7180000491028:944707-944813 - | GTTTTACGCATGGGTGCAG--GG-----------------------C-------------GCAGCTACCAGGT-------------------ATATTTCTATATATTAGAAA----------GAGAAGG-------------------------------------------------------------CGACAGGCA---------------------------CTCACAAAATGGCCCCGGAGAAAAACAACA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAAAT----------------------------- | |
| droRho1 | scf7180000758580:948-1005 + | TTTT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCAGGCCCACGA--AAAGACGCACAAATCGCC-GCGGTACGAGTGTGCGGATTGCGG | |
| droBia1 | scf7180000302261:1482377-1482433 - | TTT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCAGGCTCACGA--AAAGACGCATAAGTCCCC-GCGTTACGAGTGTGCGGATTGCGG | |
| droTak1 | scf7180000415560:72526-72653 + | GTTTTACGCATGGGTGCAG--GG-----------------------CAGCTGATTGCCCAGCAGCTACCAGAT-------------------AT-----TAAACGTTAGAGA----------GAGAGGGCAGTCGGCTGAG---------------------------------------------CAAGCGAGAAGCA---------------------------CTCACAAAATGGCCACGGAGAAAAAGAACA-----------------------------------------------------------------------GT---------------------------------------------------------------------------------------------------------------------------A-------------------------------- | |
| droEug1 | scf7180000409231:12703-12799 + | GTTTTACGCATGGGTGCAG--GG-----------------------C-------------GCAGCTACCAGAC-----------------------------ACGTTAGAAA----------GAGAGGGCACGCAGACGAG---------------------------------------------CGAAAGAGAGCCA---------------------------TTCACAAAATGGCCTCAGA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------G | |
| dm3 | chr2L:15112060-15112142 + | TTATCTTATTTGTCT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATT----ATGTTCTTATTTCAGGCTCATGA--AAAGACGCACAAATCGCC-GCGTTACGAGTGTGCGGATTGTGG | |
| droSim2 | 2l:14663659-14663714 + | TT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCAGGCTCATGA--AAAGACGCACAAATCACC-GCGTTACGAGTGTGCGGATTGCGG | |
| droSec2 | scaffold_3:1373909-1373989 + | ATCTTATTTGTCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATT----ATGTTCTTATTTCAGGCTCATGA--AAAAACGCACAAATCGCC-GCGTTACGAGTGTGCGGATTGTGG | |
| droYak3 | 2L:20221150-20221273 - | GTTTTACGCATGGGTGCAG---------------------------------------------CTACAAGAT-----------------------------ACGTTAGAAA----------GAGCA----AGCCGTCGAG---------------------------------------------CAAGCGAGAGGTA---------------------------TTCACAAAATGGCCCCTTTGAAAAAGAACA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCAAAT-------TGAGTGGCTATCTGAATAATTG | |
| droEre2 | scaffold_4929:6852342-6852458 + | GTTTTACGCATGGGTGCAG---------------------------------------------CTACGAGAT-----------------------------ACGTTAGAAA----------GAGCA----AGCCGCCGAG---------------------------------------------CGAGCGAGAGGTA---------------------------TTCACAAAATGGCCCCTTGGAGAAAGAACA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAACTCAAAT-------------------TGAATAATTG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:31 PM