
ID:dvi_10393 |
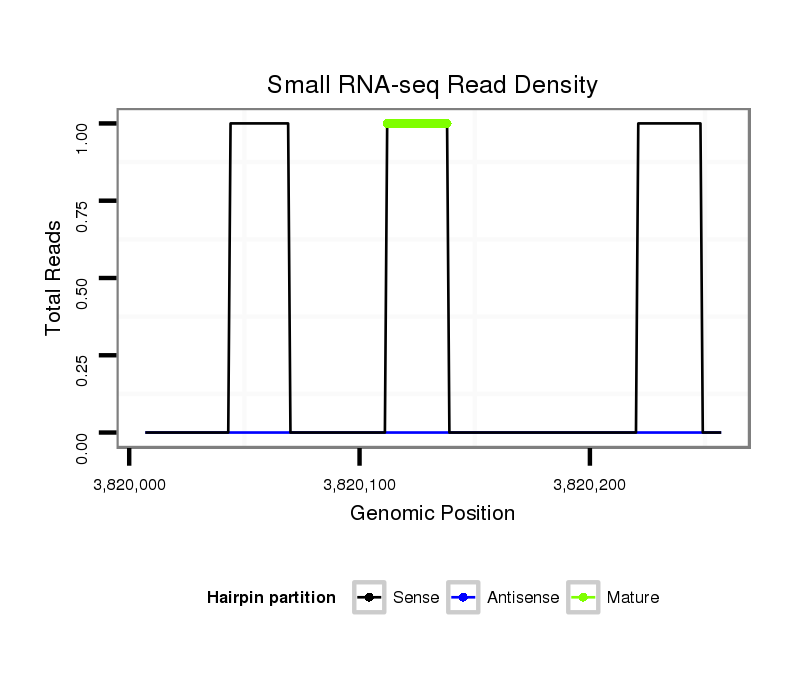
Coordinate:scaffold_12963:3820057-3820207 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
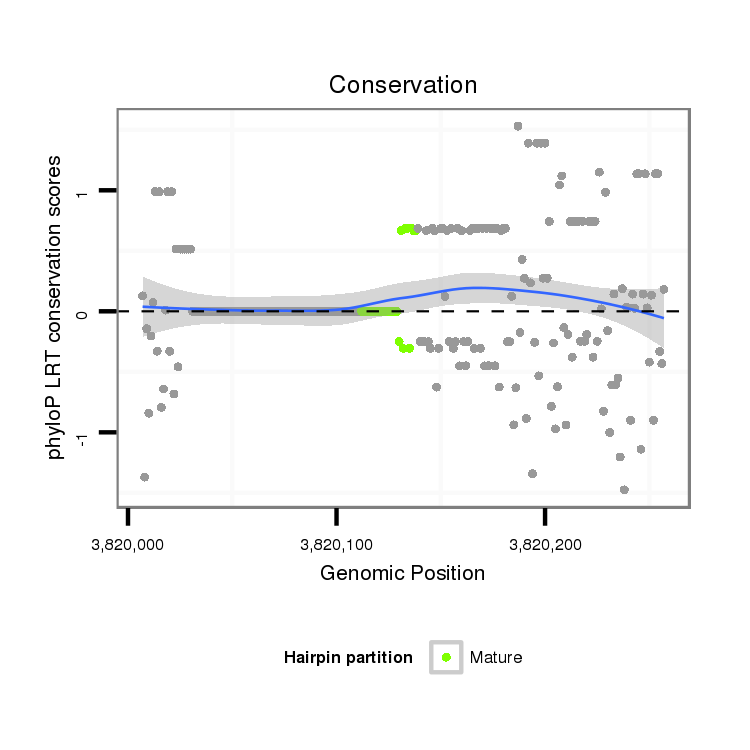
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -25.7 | -25.5 | -25.5 |
 |
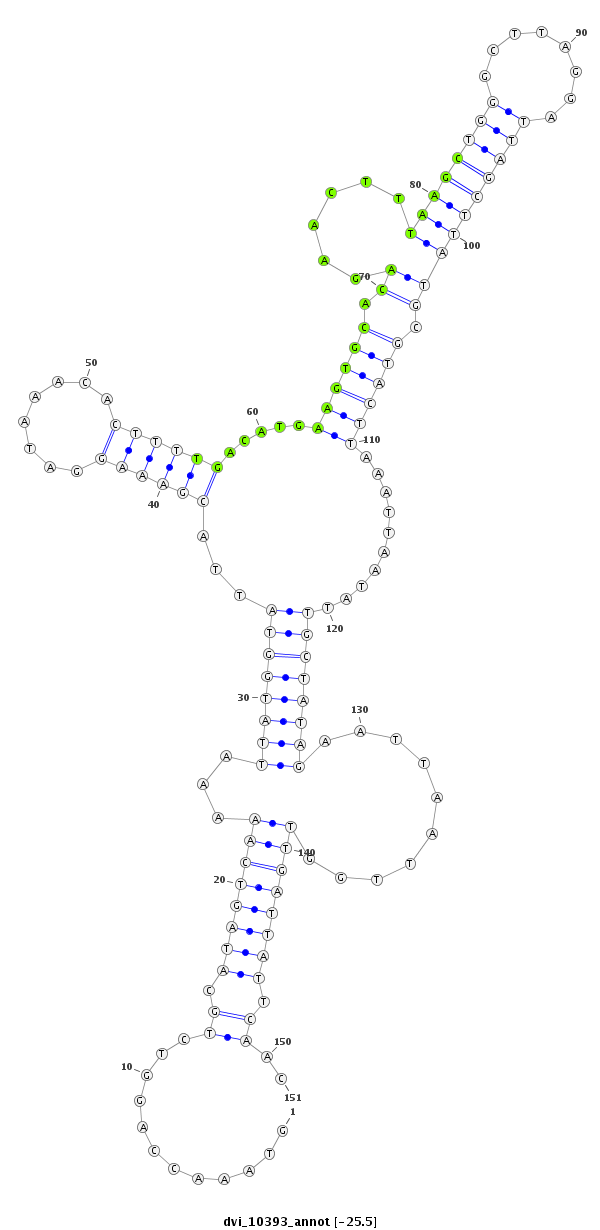 |
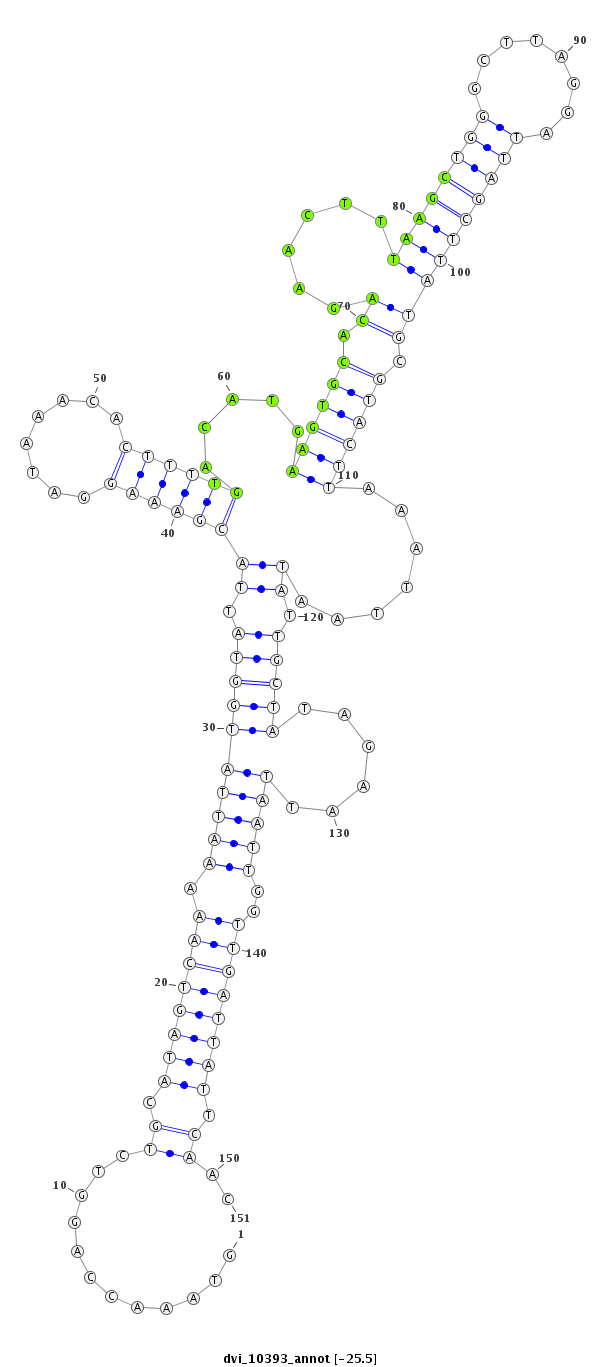 |
exon [dvir_GLEANR_1609:1]; CDS [Dvir\GJ15647-cds]; intron [Dvir\GJ15647-in]
No Repeatable elements found
| -----------------------------------------#########--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ATTTGTTCTGTTTGTTAGAAAATTCGACTCTGCGCCCTACAATGGATCAGGTAAACCAGGTCTGCATAGTCAAAAATTATGGTATTACGAAAGGATAAACACTTTTGACATGAAGTGCACAGAACTTTAAGCTGGGCTTAGGATTAGCTTATGCGTACTTAAATTAATATTGCTATAGAATTAATTGGTTGATTATTCAACTCATTAAATATACTTCTATATAGTCTCACACAACAATTCTTTCACTTTGA **************************************************............((.((((((((...((((((((.((((((((........)))))).....((((((.((......((((((((........)))))))))).)))))).......)).))))))))..........)))))))).))..************************************************** |
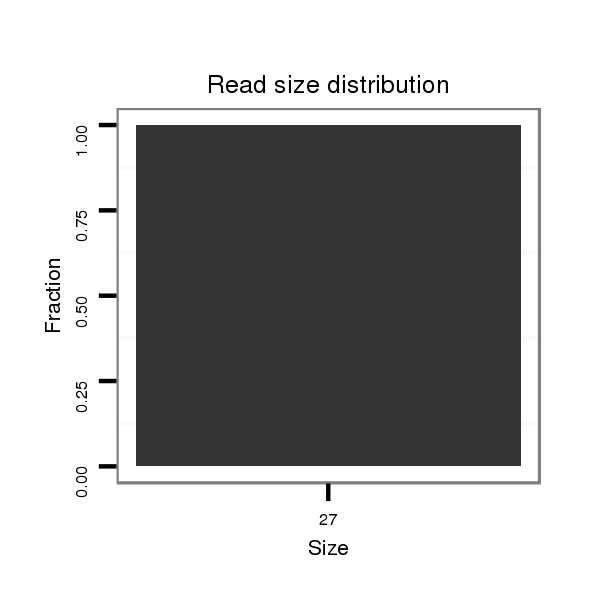
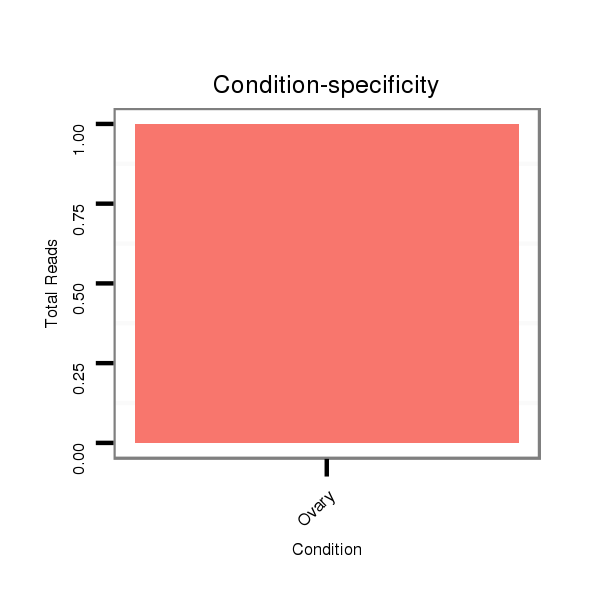
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060674 9x140_ovaries_total |
SRR060676 9xArg_ovaries_total |
SRR060677 Argx9_ovaries_total |
V047 embryo |
SRR060682 9x140_0-2h_embryos_total |
M047 female body |
M027 male body |
SRR060684 140x9_0-2h_embryos_total |
SRR060669 160x9_females_carcasses_total |
SRR060658 140_ovaries_total |
SRR060689 160x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................................................................................TTCTATATAGTCTCACACAACAATTCTT......... | 28 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................TGACATGAAGTGCACAGAACTTTAAGC....................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................TACAATGGATCAGGTAAACCAGGTCT............................................................................................................................................................................................ | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................GACATGCACTGCACAGAAC.............................................................................................................................. | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................TAGTACGAAAGGACAAACA...................................................................................................................................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................GCACAGAACTTCAAGCAGGTC.................................................................................................................. | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........TGATTGGTAGAAGATTCGAC............................................................................................................................................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...........................CTGTGCGCCCGACAATGGAAC........................................................................................................................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................CAATGGACCAGGCAACCCAG................................................................................................................................................................................................ | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........TGATTGGTAGAAGATTCGA................................................................................................................................................................................................................................ | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 |
| ..........................................................................................................................................................................................GGTTGCTTTTTCAACTCA............................................... | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................CGCAAATTCGACGCTGCGCC....................................................................................................................................................................................................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................AGGGAAACCAAGTCTGAAT........................................................................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................AATTCAACTCAGCGCCCTT.................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........TTGGTAGAGCATTCGACTCT............................................................................................................................................................................................................................ | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TAAACAAGACAAACAATCTTTTAAGCTGAGACGCGGGATGTTACCTAGTCCATTTGGTCCAGACGTATCAGTTTTTAATACCATAATGCTTTCCTATTTGTGAAAACTGTACTTCACGTGTCTTGAAATTCGACCCGAATCCTAATCGAATACGCATGAATTTAATTATAACGATATCTTAATTAACCAACTAATAAGTTGAGTAATTTATATGAAGATATATCAGAGTGTGTTGTTAAGAAAGTGAAACT
**************************************************............((.((((((((...((((((((.((((((((........)))))).....((((((.((......((((((((........)))))))))).)))))).......)).))))))))..........)))))))).))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060684 140x9_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
V116 male body |
M027 male body |
|---|---|---|---|---|---|---|---|---|---|
| .....................................................................................................................................................ATACGCATGAATTCAAATATA................................................................................. | 21 | 2 | 1 | 2.00 | 2 | 1 | 1 | 0 | 0 |
| ......................................................................................................................................................TACGCATGAATTCAAATATA................................................................................. | 20 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 |
| .................................................................................................................TCACGTGTCTAGCAATTCGG...................................................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 |
| ...........................................................................................................................................................................................CATCTAAAAAGTTGAGTAA............................................. | 19 | 2 | 12 | 0.08 | 1 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:3820007-3820257 + | dvi_10393 | A-------TTTGTTCTGTTTGTTAGAAAATTCGACTCTGCGCCCTACAATGGATCAGGTAAACCAGGTCTGCATAGTCAAAAATTATGGTATTACGAAAGGATAAACACTTTTGACATGAAGTGCACAGAACTTTAAGCTGGGCTTAGGATTAGCTTATGCGTACTTAAATTAATATTGCTATAGAATTAATTGGTTGATTATTCAACTCATTAAATATACTTCTATATAG---------------------------------------------------------------------------------------------------------------------------------------------------------------TCTCACACAACAAT------------------------------------------TCTTTCACTTTGA |
| droMoj3 | scaffold_6328:152147-152235 + | A-------CTTAA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAAACTTAATTAAACAATTAATTAA-----------------TTAATTA---------------------TAATATTTATAAC------------------------------------ATATGCTT----------------------------------CTATTCCAAACAAATT------------------------------------------TGTTTCAATTCGA | |
| droGri2 | scaffold_15126:7129983-7130227 - | A-------ATAATTTTCGTTATGAAAAAATT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGCTGC---------------------------------TTCAATCAGTGGAAAAAATCAGGTATTATTTATAAGTCTCTTTCTTTCGCTCTTTCCACCAGGAAAGTGGGAAGAAATTAAGTAATTTATGCATTTAATTTTAATTTGAGCTGCCTGGTTTTAAATGCATCTTTTTTTATTTATTTATTATTTTATTATTGTTATTCTTGTTGTTATTATTATTAAAGTTTAAAGTATTAAGTTTTAAATTTTAG | |
| droWil2 | scf2_1100000004902:3285462-3285538 - | A-------CAGA----------------------------------------------------------------------------------------------------------------------GCATTTAGCTAAACCAAGCTTTAGTTAGTTCAGGCTAAATTGACTAGTCCTACGGTTTTAATTAATTGATTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302817:395312-395358 + | TTTCTATA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAATAAAATGTCTAATTA-----------------------------------------------------------------------------------------------TTCTATGTAATAA------------------------------------ATATACTT--------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491017:348858-348941 + | TTAATATT------------------------------------------------------------------------------------------------------------------------------------------------A--------------------------------ATTAATGTGCCTTAATT-GCTGCTGC--------------------------------------------------------------------------TTCCGTGGCTCCTTCAACTCATTAA------------------------------------AAACACATCAGCTTGG----------------------------CCTCACAC------------------------------------------------------------A | |
| droBia1 | scf7180000302025:38345-38422 + | T-------ATA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGATTAATTGATTGATTATTTCACTGTCTCAATTTTTTTATTTAT---------------------------------------------------------------------------------------------------------------------------------------------------------------ATATCAATAAAATGTT------------------------------------------TTCTTTTTTTCTA | |
| dm3 | chr3R:13389604-13389643 + | TAAATATA------------------------------------------------------------------------------------------------------------------------------------------------T--------------------------------TTTAG------------------------------------TAAAGTCTAATACACTAATTCATTCA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 2L:6732361-6732375 - | A-------TGTGTTTTGTATAT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/16/2015 at 10:10 PM