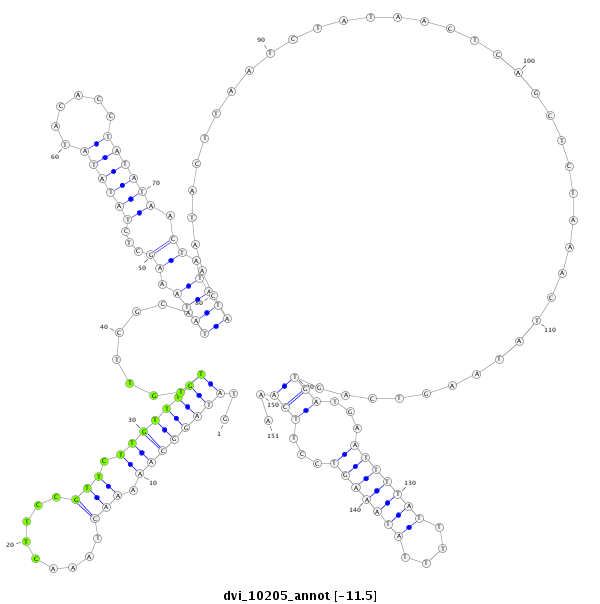
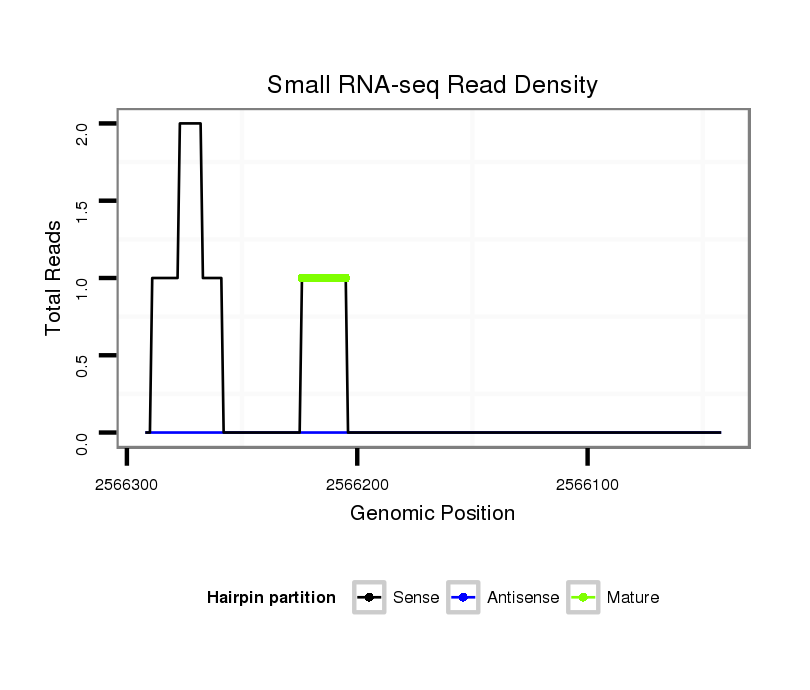
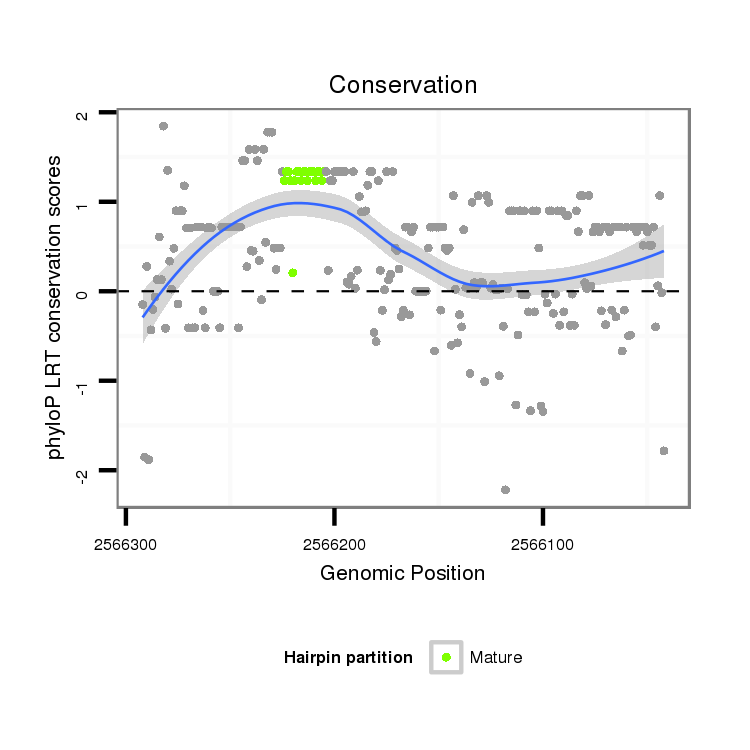
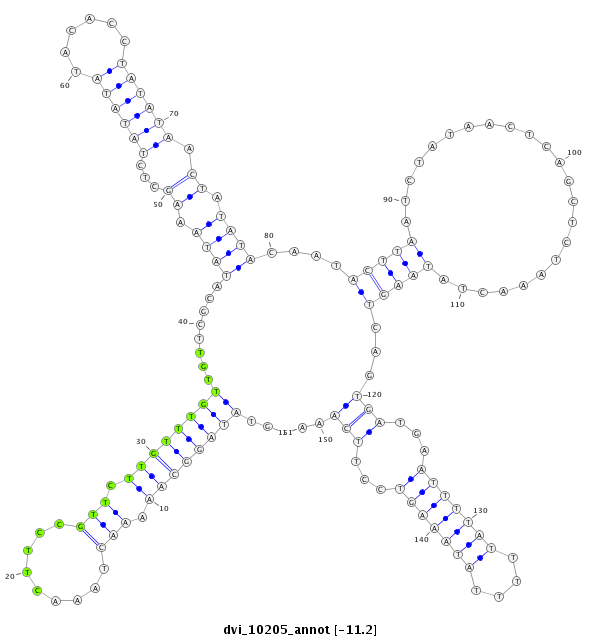
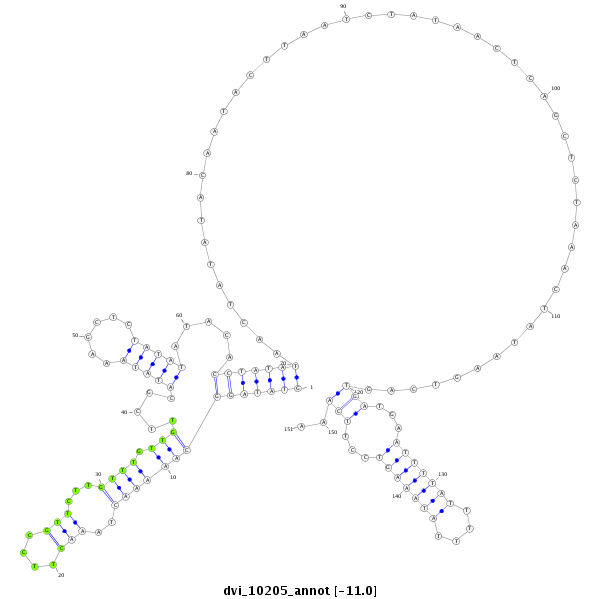
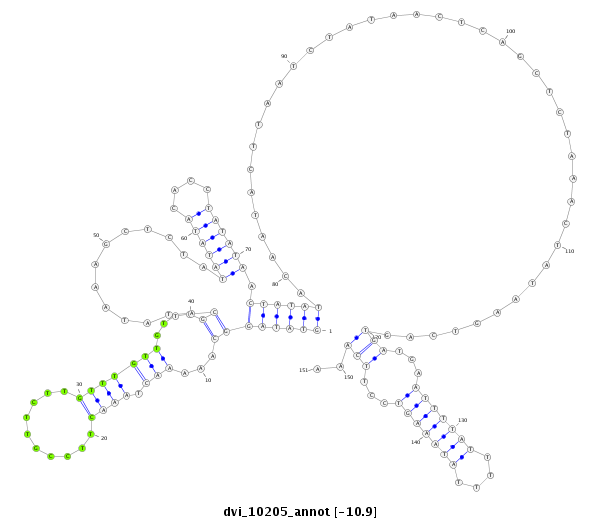
ID:dvi_10205 |
Coordinate:scaffold_12963:2566092-2566242 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -11.2 | -11.0 | -10.9 |
 |
 |
 |
exon [dvir_GLEANR_1290:1]; CDS [Dvir\GJ12970-cds]; intron [Dvir\GJ12970-in]
No Repeatable elements found
| ----------------------------------------------####--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TGTTTTAAAAATGATATAAAACCGTAAAAGAGACATAAAATTAAAAATGGGTATAGGCAAAAACTAAACTTCCGTTCTTGTTTGTTGTTCGCATATAAAGCTCTATATATACACCTATATAACTATATACAATACTTAATCTATAACTCAGCTCTAAACTATAAGTCAGTGATGAATTTTATTTTTATAAAGTCCTTCAAATTCCTAATCTTTTTTGTAGTTCTGTTAAACTAAATTCATTACTTTCAAAT **************************************************..((((((((.(((.........))).))))))))........((((.((...((((((......)))))).)).))))........................................(((...(((((((....)))))))...)))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060660 Argentina_ovaries_total |
SRR060683 160_testes_total |
GSM1528803 follicle cells |
V116 male body |
M027 male body |
SRR060684 140x9_0-2h_embryos_total |
SRR060677 Argx9_ovaries_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060654 160x9_ovaries_total |
SRR060667 160_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................CTTCCGTTCTTGTTTGTTGT................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...TTTAAAAATGATATAAAACCGT.................................................................................................................................................................................................................................. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............ATAAAACCGTAAAAGAGAC......................................................................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................TTATTTTTATAAAGTCCA....................................................... | 18 | 1 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................AAATTTATTTTTATAAATTCC........................................................ | 21 | 2 | 12 | 0.17 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................AACAACTTAACCTCCGTTCT............................................................................................................................................................................. | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................ATTCTTGTTCGTTGTCCGCA.............................................................................................................................................................. | 20 | 3 | 18 | 0.11 | 2 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................AACTTAACCTCCGTTCTGG........................................................................................................................................................................... | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................TTAAAAATGGGTATAAGAAAAA............................................................................................................................................................................................. | 22 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................AGCTATATATATACAACTATAT................................................................................................................................... | 22 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................TGCATGAACTAAACTTCCG................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...............................................TGAGTCTAGGCAAAAGCTAAA....................................................................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........AATGATTTAAAACCGAAAA............................................................................................................................................................................................................................... | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
ACAAAATTTTTACTATATTTTGGCATTTTCTCTGTATTTTAATTTTTACCCATATCCGTTTTTGATTTGAAGGCAAGAACAAACAACAAGCGTATATTTCGAGATATATATGTGGATATATTGATATATGTTATGAATTAGATATTGAGTCGAGATTTGATATTCAGTCACTACTTAAAATAAAAATATTTCAGGAAGTTTAAGGATTAGAAAAAACATCAAGACAATTTGATTTAAGTAATGAAAGTTTA
**************************************************..((((((((.(((.........))).))))))))........((((.((...((((((......)))))).)).))))........................................(((...(((((((....)))))))...)))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR1106729 mixed whole adult body |
V047 embryo |
SRR060663 160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................................AAAACATCAAGACAA........................ | 15 | 0 | 3 | 0.33 | 1 | 1 | 0 | 0 |
| ...........................................................................................................GTATGTGGATATATTGGTTTAT.......................................................................................................................... | 22 | 3 | 12 | 0.17 | 2 | 0 | 2 | 0 |
| ..................................................................................................................................................................................................................AAAAAACATCAACACAAT....................... | 18 | 1 | 9 | 0.11 | 1 | 0 | 1 | 0 |
| ..........................................................................................................................................................................................................TGGCTTAGAAAAAACATC............................... | 18 | 2 | 13 | 0.08 | 1 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:2566042-2566292 - | dvi_10205 | TGTTTTAAAAATG----ATA---TAAAA-----------------------------------CCGTAAAAGAGACATAAAATTAAAAATGGGTATAGGCAAAA----ACTAAACTTCCGTTCTTGTTTGTTGTTCGCATATAAAGCTCTATATATACACCTA-----------------TATAACTA---------------TATACAATACTTAATCTATAACTCAGC-------------TCTAAACTATAAGTCAGTGATGAA---------------------------------TTTTATTTT---TATAAAGTCCTTCAAATTCCTAATC------------------------------------------------------------TTTTTTGTAGTTCTGTTAAACTAA-ATTCAT-TACTT--T-CAA--------------AT |
| droMoj3 | scaffold_6500:12754422-12754708 - | TGTATGGAAAAAGTTCGATA--------AAAAGTTAATTAAATGAGAAGAT--CAAAT--------------------------------GGGTATAGGCAAAA----ACGAAACTTCCGTTCTTGTTTGTTGTTCGCATATAAAGCTCTATATATACTCCTA-----------------TATAACTGCATATTAACCATATATATTCA------AATATATAACTGAA-----------------GATCTATAACTCAGTGCTATATATTTTCTTCCTTCCTGTTCTATTTTCTGTTACTTTTCTTTT---CAT-AAGTCTTTCAATTCCCAATT------------------------------------------------------------GTTTTTCGTAGTTTTGTTGAAATACTCTTCAT--AC---GTT-TA--------------AC | |
| droGri2 | scaffold_15252:2177717-2177972 - | TATTTGGAAAA-------TTATGTGAAAAAGAG---------AGAGAAGA-GACAAAT--------------------------------GGGTATAGGAAAAAAAAGACT--ACTTCAGTTCTTGTTTGTTGTTTGCATATAAAGCTCTA----TATCCTATATATAT-----TCTCTATATA--TGTATAT---ATATATACATACA-------ATATACA----AA-----------------GGCCTTTAACTCAGTGCTATGTGTTCTCT--------------------------------------------------------------------------------------------TTATATACTATATACTCCTAAATCCTAACGTTTCTTGTAGATTTGCTGAAACAA-CTTCATATACTTGGTT-AA--------------AT | |
| droWil2 | scf2_1100000004521:6864017-6864131 - | TGTGAAA----AG----TCAAA-----AAATAA---------TAAAAAAA-AAAACAT--------------------------------GGGTTTAGGCAAAA----ACT--ACTTCCGTTCTTGTTTGTTGTTCGCATATAATACTAT----ATAATCCATATATAA-----GCTCTATGTA--C------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dp5 | 4_group5:1825689-1825956 - | ACTTAAC-AAAAG----ATAAAGTGAAAGAAAG---------AAAG---TGCAAATAT--------------------------------GGGTATAGGCAAAA----ACT--ACTTCCGTTCTTGTTTGTTGTTCGCATATAATAATA----TATAATCCTTATAAACGTTATACTCTATGGA--T---------------------------------------CAAGGGAATACTCACTGAATGAACCATGGATCCATTGGGGA---------------------------------TTTTATATATCATGTGA-TTTCTTGGATTTCCAGTTCATTTTTGGGAGCTTCCCCTTGATTCCTTC------TTCTATAAAACTATAATCCTTAATATTCTTT-----------------------------------AAA--------------GA | |
| droPer2 | scaffold_5:6231198-6231487 - | TTAACAAAAGA-------TAAAGTGAAAGAAAG---------AAAG---TGCAAATAT--------------------------------GGGTATAGGCAAAA----ACT--ACTTCCGTTCTTGTTTGTTGTTCGCATATAATAATACATATATAATCCTTATAAACGTTATGCTCTATGGA--T---------------------------------------CAAGGGAATACTCACTGAATGAACCATGGATCCAATGGGAA---------------------------------TTTCATATATCATATGA-GATCTTGGATTTCCAGTTCATTTTTGGGAGCTTCCCTTTGATTCCTTTTTTCCTTTCTATAAAATTATAATCCTTAATATTCTTT-----------------------------------AAAGAAAAGTTTCATTTAT | |
| droBip1 | scf7180000395493:11876-11886 - | TATATCAAAAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302808:33051-33111 + | AATTTAATAAA------------------------------------------TAAATTGGTACAGGCAAAACGAC---CAATTAAAACTGGCTAAAGTAACAA----AC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000490466:6491-6502 - | GGTAGAAATTAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000412962:10692-10712 - | TACATAAGTAAAG----ACA---------------------------------------------------------------------------------AAA----AA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
Generated: 05/16/2015 at 08:08 PM