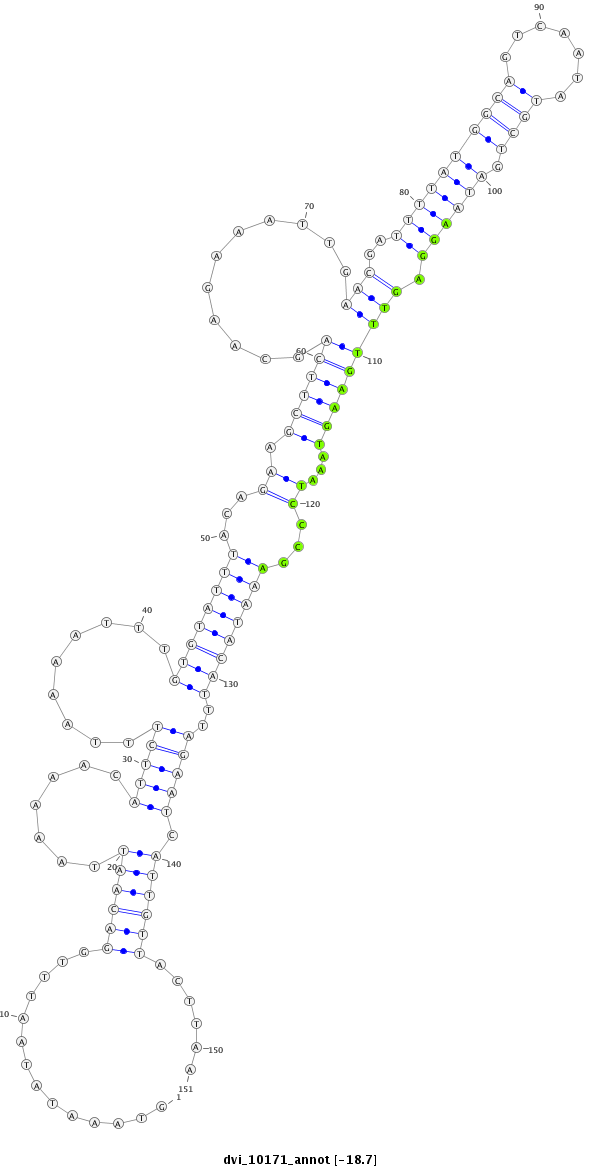

ID:dvi_10171 |
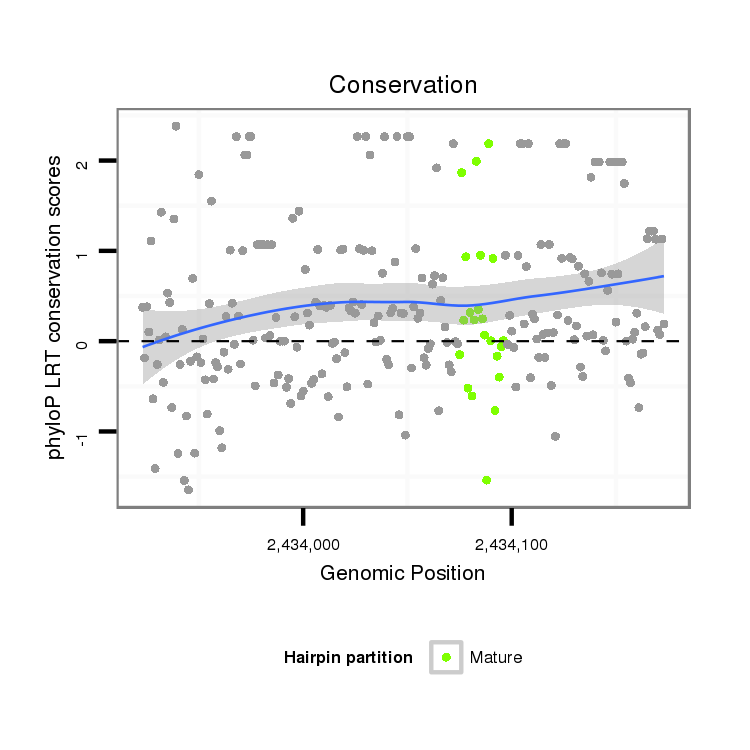
Coordinate:scaffold_12963:2433973-2434123 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -18.8 | -18.8 | -18.4 |
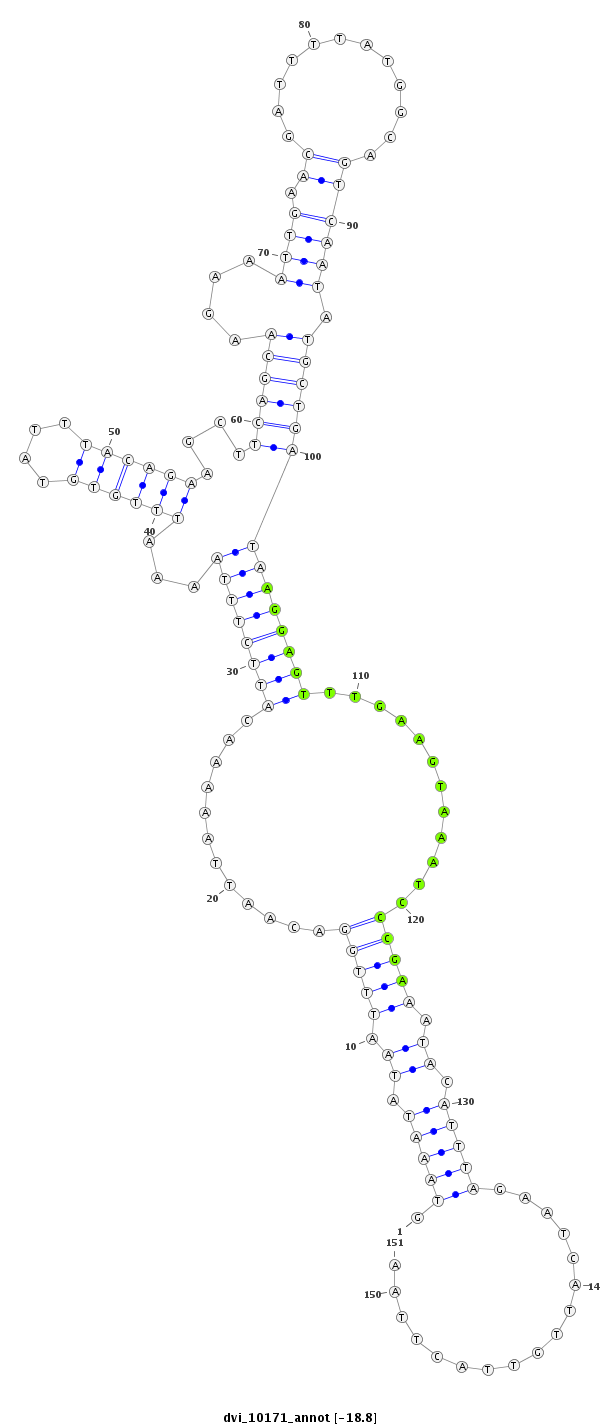 |
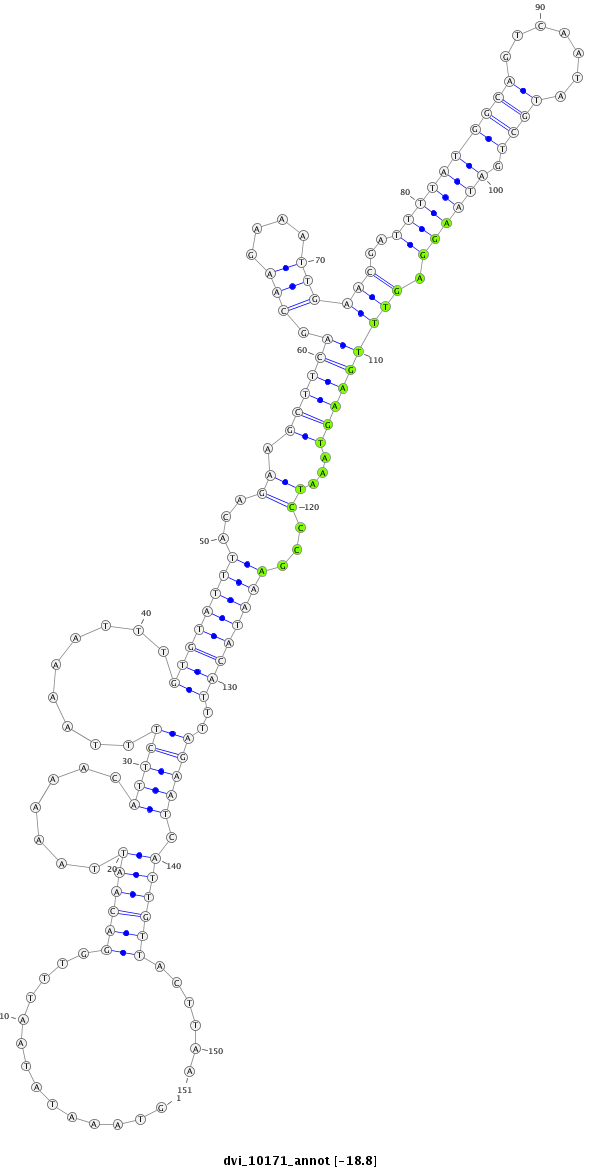 |
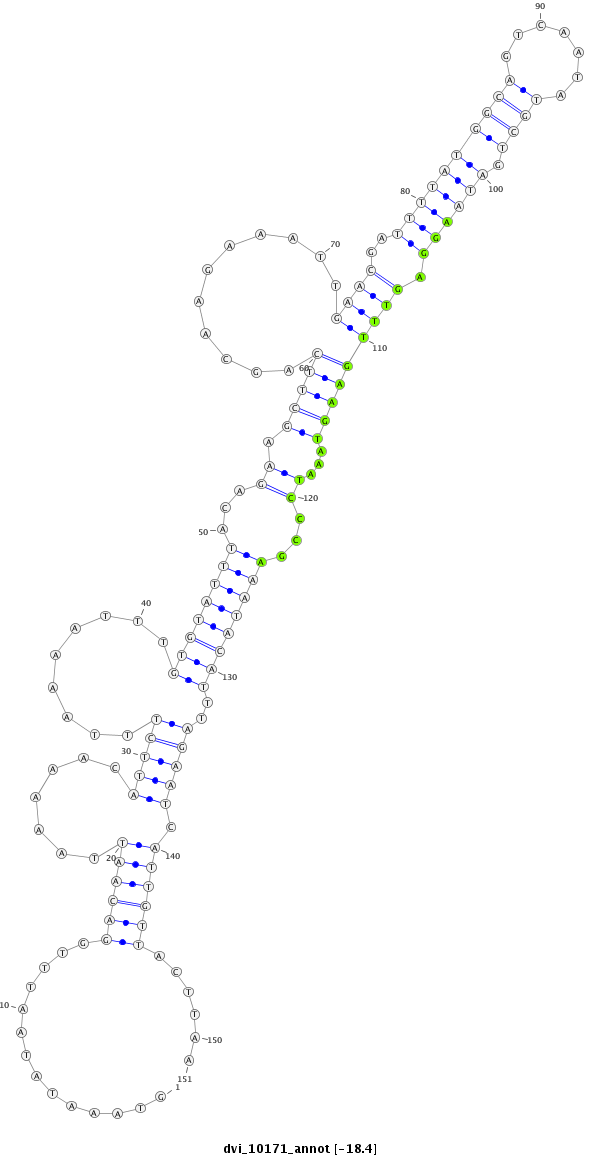 |
exon [dvir_GLEANR_1535:1]; CDS [Dvir\GJ15046-cds]; intron [Dvir\GJ15046-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CTGAATATGATCATACAGCGCCGGATGAAGCTGCTCGGCTAAAAGTGATGGTAAATATAATTTGGACAATTAAAAACATTCTTTAAAATTTGTGTATTTACAGAAGCTTCAGCAAGAAATTGAACGATTTTATGGCAGTCAATATGCTGATAAGGAGTTTGAAGTAAATCCCGAAATACATTTAGAATCATTGTTACTTAAATATAATACACTACGTAATACTGTTTTTTAAATATTCTAGTCTTTTAAAT **************************************************..............((((((.......(((((.........((((((((...((.((((((...........(((..((((((((((.......)))).)))))).)))))))))...))...))))))))..))))).))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060665 9_females_carcasses_total |
SRR060679 140x9_testes_total |
V116 male body |
V047 embryo |
M047 female body |
SRR060689 160x9_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060669 160x9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................................................................AGGAGTTTGAAGTAAATCCCGA............................................................................. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................AATACATTTAGAATCATTGTTA....................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................TAATACTGTCCTTTAAATATTTT............ | 23 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................ATGGAAGGATTTTATGGCA.................................................................................................................. | 19 | 2 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................TTGAAGGACATCGCGAAATA......................................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................ATGCTCGGCGAAAAGGGAT.......................................................................................................................................................................................................... | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................TAATACTGTTATTTAAATATTTC............ | 23 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................................................................................................................................................................ATACTGTTTTTTAAGTGTCC............. | 20 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................AGATAAAAGTGAGGGTAAAT................................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..................CGCGGGATGGAGCAGCTCG...................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
|
GACTTATACTAGTATGTCGCGGCCTACTTCGACGAGCCGATTTTCACTACCATTTATATTAAACCTGTTAATTTTTGTAAGAAATTTTAAACACATAAATGTCTTCGAAGTCGTTCTTTAACTTGCTAAAATACCGTCAGTTATACGACTATTCCTCAAACTTCATTTAGGGCTTTATGTAAATCTTAGTAACAATGAATTTATATTATGTGATGCATTATGACAAAAAATTTATAAGATCAGAAAATTTA
**************************************************..............((((((.......(((((.........((((((((...((.((((((...........(((..((((((((((.......)))).)))))).)))))))))...))...))))))))..))))).))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060668 160x9_males_carcasses_total |
V116 male body |
SRR060679 140x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060681 Argx9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ............................................................................................................................................TTGCACGACTATTCCTCAA............................................................................................ | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................TGGAAATGTTAGTCACAATGAA.................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................AGTAATAATGAATTTATAT............................................. | 19 | 1 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................TGGGGCTTTACGTAAATCGT................................................................ | 20 | 3 | 18 | 0.22 | 4 | 0 | 0 | 0 | 0 | 4 | 0 |
| ...................................................................................................................................................................................................TGAATTAATATTATGGGCTGC................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................................................................................................................................................GAATTAAAATTATGTGATGAA.................................. | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................................................................................................CGTTGTGTAACTTGGTAAAA........................................................................................................................ | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:2433923-2434173 + | dvi_10171 | CTGAATATG-------ATCAT----------ACAGCGCCGGATGAAGC---TGCTCGGCTAAAAGTGATGGTAAA---TATAATTTGGACAATTAAAA--A--------------------CATTCT-------T-------------------TAAAAT--------TTG-TGTATTTACAGAAGCTTCAGCAAGAAATTGAACGATTTTATGGCAGTCAATATGCTGATAA---GGAGTTTGAAGTAAATCCCGAAATACATTTAGAATCATTGTTACTTAAATATAATACACTACGTAATACTGTTTTTTAAATATTCTAGTC--TTTTAAAT |
| droMoj3 | scaffold_6500:12592630-12592887 + | CAGAAAATG-------AGCCT----------TCAACAGGAGACGAAGC---GGCGCATTTAAAAATTTTGGTAAA---TATAATATGAA---TTCAAA--A-T----ATCCA-----TTCACTTCAA-------G-------------------TAAAAA--------ATT-ATTTTTCACAGAAATTACAACATGAAATTGATCGGTTTTATGGCGCTCAAAATACTGAAGA---GGAATTTGAAGTGAATGCCCAAATACATTTAGAGTCATTGTTACTAAAATATAATACAATACGTAATACTGTTTTTTAAATTCACCAGAC--TTTTAAAT | |
| droGri2 | scaffold_15126:2577604-2577862 - | CAAAATTTGAGCAAGT----T---GC-------ACCAGCGGAGGAAGC------AAAGCAAAAACTTTTGGTAAG---TATAATTTTAA---AAAATT--A-T----ATCCA-----AAAAAATTTT-------C-------------------TAAAAT--------AT--AGATTTTGCAGAAATTACAACATGAAATAGAGCGATTTTATGGCACTCAATGTGATGATGAG---CAGTCTGAAGCAAATGTGGAAATGCATTTAGAATATCTGCTATTAAAATATAACGCTTTGCGAAATACTATTTTTTAAATGTACATGTCTGTTTTGAAT | |
| droWil2 | scf2_1100000004516:3736771-3737016 - | CTGAATGGAT--------CACTAAAGATTCTTCAAA------------------AACTACTGAGATTATGGTAATTAC--------------------------TAGT----TTAT----TCCTTAT-------CCAATATCAAAGACGGTGACTTATAGTAATCTTTGTT-TCCTTTTCTAGAAATTACAATGTCAAACGGAGCGTTTATATGGCACATTAAGTGATAATGGCATGGATATTGAGAGCAATGTGAGAATGCGTTTAGAGGCACTACAAACGCAATATAAAGCCATAAGGAATACAATTTATTA---------------------- | |
| dp5 | 4_group4:6470038-6470279 - | CTGAGAGAG-------ATTAT----------ACAAT------------------ATTAAATCAAGTTATGGTAATTTCTATACTTTAAT---TTCAAT--A-T----ATTTCTTATATTC----------------ACTTTAATACACTTTGGCTAATAT--------TTAAACATTCAACAGAAATTTCAATGTGAAACTGAACGTTTATATGGTACGCAAGGCGGAGATGGGTCGGACTGTGGAACTAATATTGGATTGCGATTGGAATC---------GCAATATACTGCCATTAGAAATACAATTTTTTAACCATT---------------- | |
| droPer2 | scaffold_16:390000-390241 + | CTGAGAGAG-------ATTAT----------ACAAT------------------ATTAAATCAAGTTATGGTAATTTCTATACTTTAAT---TTCAAT--A-T----ATTTCTTATATTC----------------ACTTTAATACACTTTGGCTAATAT--------TTAAACATTCAACAGAAATTTCAATGTGAAACTGAACGTTTATATGGTACGCAAGGCGGAGATGGGTCGGACTGTGGAACTAATATTGGATTGCGATTGGAATC---------GCAATATACTGCCATTAGAAATACAATTTTTTAACCATT---------------- | |
| droAna3 | scaffold_13258:396043-396288 + | TCGAATCTAG------ACCACAAAGC-------ATTATTAAAAGATCGTTTAGCTAATCTGCAAATAAAGGTAAA------------AT---TTAAAA--A-T----ATGTTTTCT----TAAGTAT-------TTAATTTT------------CATCAT--------GCAAATTTTCAACAGAAATTCCTGTGTGAATCTAAGCTTTTTGACA---TT---------TATAGTCTGGAAGGCGAATCTACCCTAAAAATGGCTTTGGAGGA---------CAAGTACAATGCAGTTAGAAATAGTATCTTTTAAGATGTCCAATC--TTTTAA-A | |
| droBip1 | scf7180000393981:44791-45029 + | CTAGAC------------CATAAAGC-------ATTATTCGATAATCGTTTAGCTTATCTGCAAATAAAGGTAGG-------------T---TA---ATAACATTAAT----TTCC----TTAGTAT-------TCAATATT------------AAACAT--------ACACATGTTCTACAGAAATTGCTGTGTGAAACTAAGCTGTTTGACG---TT---------CACGATCTGGAAGGCGAAACGACTGTAAAAATGGCTTTGGAATC---------AAAATACTATGCAACTAGAAATAATATTTTCTAAGGTGTCCATTC--TTT-AGAA | |
| droFic1 | scf7180000452363:37957-37976 + | CTGAACTCAAAG----ATTAT----------GCA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000490112:152-357 - | CTGAATTTAC--CTTC----T---GC-------AACACTAGAAAACGCTTCAACTTTAAATAAAATATTGGTATG-------------A---GT---GTA--T---------TAAT----TTTTTAACTAACATT-------------------TAATGT--------TTA-ATTTTCAACAGAAATTTCAGTGCGAGTCTGAGAGATTGTACGGAGCACAAAGTAAGGATGTCTTGGACTACGAAACGATTTCTGAAGGATGTTTGAAATC---------AAAATACAATGC------------------------------------------- | |
| droBia1 | scf7180000297823:2620-2754 - | CTGGATATAG--AAGA----T---TC-------ACTAAAGTATAATGATTTTACTTTTGATAAAATAATGGTATG-------------G---AT---AAA--ATTAGT----TCTA----GTTTTAT-------T-------------------TAATAT--------TTA-TTTTGTAACACAAATTTCAGACTGAAACTGAGCTGTTGTACGG------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000411738:621-828 + | CTGAATTTAC--CCCC----T---GC-------AACACTAGGAAACGCTTCAACTTTAAATAAAATATTGGTATG-------------A---GT---ATA--TT--------TAAT----TTTTTAACTAACATT-------------------TAATGT--------TTAATTTTTTAACAGAAATTTCAGTGCGAGTCTGAAAGATTGTACGGAGCACAAAGTAAGGATGTCTTGGACTACGAAACGATTTCTGAAGGATGTTTGAAATC---------GAAATACAATGC------------------------------------------- | |
| droSec2 | scaffold_42:24222-24442 + | CTGGAACC------------T---GC-------ATCCAAAGAATATAACATGACCTTTCATTCAATAATGGTAAG-------------T---AA---ATA--AT--------TTAT----CTATAAC-------C-------------------TAACAT--------TG--ATCTTACAAGAAGATTTCAGAGCGACGTTGACCGATTTCGGTACCGACAATGTAACGACATTTTGGCCTGTGGAACAAATTTTGCC---AATTTAAAGTC---------AAAATATAAAATCATTAGAAATGACATTTATTAAGAATTTTGG------------ | |
| droEre2 | scaffold_4845:1828609-1828827 - | TCGAATCTG----------------C-------ATCAAAGAAATATGACTTAACCTTTAATACTATAATGGTAAG-------------T---AC---ATA--AA--------TTAT----GAATAAC-------C-------------------TATGAT--------TT--ATCTTACAACAAGTTTTCAGAGCGATGTTGACCGATTTCGGTACCGACAATGTAACAACATCCTGACCTGCGGAACAAATTTTGCAATAATTTTAAAGTC---------AAAATATAATATCATTAGAAATGACATTTATTAAGAATT---------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 04:02 PM