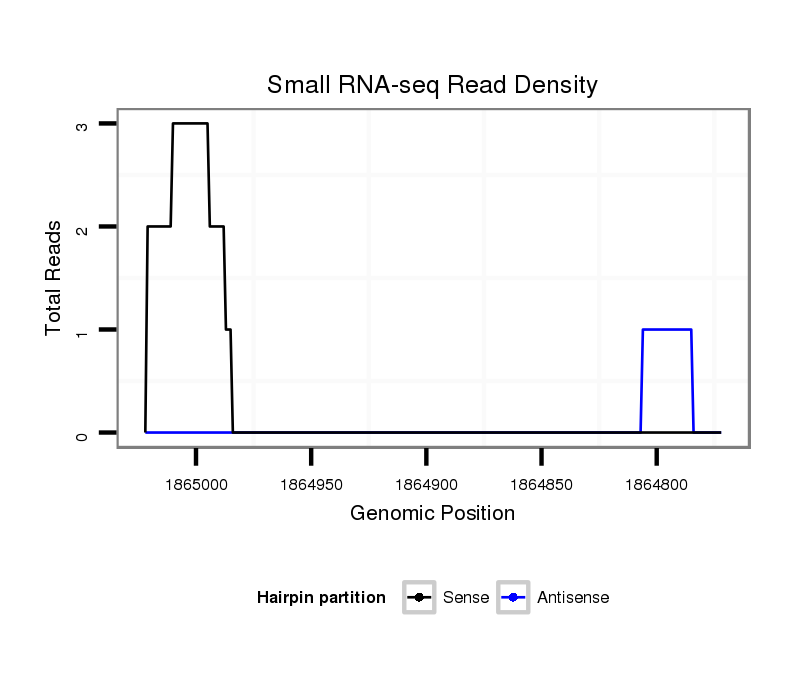
ID:dvi_10077 |
Coordinate:scaffold_12963:1864822-1864972 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ13469-cds]; exon [dvir_GLEANR_1336:1]; intron [Dvir\GJ13469-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TGCAGCTCATGAGCGGGCAAATCGATCCGGTGGATGGCCTCAAAGTAACGGTAAGTTAGTAAGTTAGTTAGTTGGCCAACGATTACCAAGACAACAAGTATTTGCCAATCGGCTATTGGGTAATGTTTTTATTGAAAGAGTTGAAAGTGGCCAGCCGTGCGTATACTTGATATTTTATATTTATCCCATTGTTTTTAGACAAACATACTTGCCCCGGGCCGGGAGTCATAAATTTAGTTACATTTATTAAA **************************************************(((((.((.(((((..((..((((((((.(((((..((((((.....)).))))..)))))(((.((.(((((.....))))).))..)))......))))))))...))....))))).)).))))).......................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060667 160_females_carcasses_total |
SRR1106730 embryo_16-30h |
V047 embryo |
V116 male body |
SRR060659 Argentina_testes_total |
M047 female body |
SRR060676 9xArg_ovaries_total |
M027 male body |
V053 head |
SRR060683 160_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................GATTACTAAGACACCAAGTAGT..................................................................................................................................................... | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............GCGGGCAAATCGATCCGGTGGAT........................................................................................................................................................................................................................ | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .GCAGCTCATGAGCGGGCAAATCGATCCGGTGGATGGC..................................................................................................................................................................................................................... | 37 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .GCAGCTCATGAGCGGGCAAATCGATCC............................................................................................................................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................ATTGAGCGAGATGAAAGTGGC.................................................................................................... | 21 | 3 | 4 | 0.50 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................AACTAGTATTTCCCAAGCGGC.......................................................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..............GGCCATATTGATCCGGTG........................................................................................................................................................................................................................... | 18 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 |
| ...............................................................................................................................TGTATTGAAAGAGTTAAAA......................................................................................................... | 19 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .............................................................................................................CGGCTGTTTGGTAATGTT............................................................................................................................ | 18 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................................................................................................................................TTATATTTCTCCCATTGTT.......................................................... | 19 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................TATCCGCTGGATGACCTC.................................................................................................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ACGTCGAGTACTCGCCCGTTTAGCTAGGCCACCTACCGGAGTTTCATTGCCATTCAATCATTCAATCAATCAACCGGTTGCTAATGGTTCTGTTGTTCATAAACGGTTAGCCGATAACCCATTACAAAAATAACTTTCTCAACTTTCACCGGTCGGCACGCATATGAACTATAAAATATAAATAGGGTAACAAAAATCTGTTTGTATGAACGGGGCCCGGCCCTCAGTATTTAAATCAATGTAAATAATTT
**************************************************(((((.((.(((((..((..((((((((.(((((..((((((.....)).))))..)))))(((.((.(((((.....))))).))..)))......))))))))...))....))))).)).))))).......................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060674 9x140_ovaries_total |
V047 embryo |
V116 male body |
SRR060670 9_testes_total |
SRR1106729 mixed whole adult body |
SRR060657 140_testes_total |
M061 embryo |
SRR060685 9xArg_0-2h_embryos_total |
M047 female body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................................................................................................................................CCGGCCCTCAGTATTTAAATCA............. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................CCGTTGGTATGAACGGGGCC.................................. | 20 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................GTTTGTATGAAGGGGGCC.................................. | 18 | 1 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................GTATGAAGGGGGCCTGGC.............................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................AGTTGGTATGAACGGGGC................................... | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................................................CTAATGGTTCTGGAGATC......................................................................................................................................................... | 18 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 |
| ....................................................................................................................................................CCAGTCGGCACGCATTT...................................................................................... | 17 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................................CGATAACCCATCACAGAAAC........................................................................................................................ | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................CTAATGGTTCTGGAGTGC......................................................................................................................................................... | 18 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........................................................................................................................................................................................................GTATGAAGGGGGCCCGAA.............................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:1864772-1865022 - | dvi_10077 | TGCAGCTCA-TGAGC-------------GGGCAAATCGATCCGGTGGATGGCCTCAAAG------TAACGGTAAGTT------------------------------------------------AGTAAGTTAGTTAGTTGGCCA-------ACGATTACCAAGACAACAAGTATTTGCCAATCGGCTATTG-GGTAATGT----TTTTATTGAAAGA---------GTTG--AAAGTGGCCAGCCGTGCGTATACTTGATATTTTATATTTATCCCATTGTTTTTAGACAAACATACTTGCCCCGGGCCGGGAGTCATAAATTTAGTTACATTTATTAAA----------------------------- |
| droMoj3 | scaffold_6500:28830693-28830926 - | TGCTGCTGC-TGAGC-------------GGACAAATCGAACCGGTGGATGGCCTCAAAG------TAACGGTAAGTTGTTAGTTAGCCTTAGT----------TGG-------------------------------AGTTGGCCAACAA----------------------GT-CTTGCCAATAAGCAATTG-CCCAAGCCTCGCATTAAGTGCAAGAAAACACCAAGCCAAACAGGTAGCCGAGCGTGCGTATACTTAATATTTTATAATTTT----------------------------------------------------TTAAATTTTATTATATTCACTTGCTACACATTCATTTTCACTTA | |
| droGri2 | scaffold_14978:456439-456606 + | TGCAATTAC-TAAGC-------------GGACACATCGAACCCGTGGATGGCCACAAAG------TGACGGTAAGTCAGTTACCAGTTGCCTGTTACCAGTTA-CCATTTAC----------------CAGTTAAC-AGTTAGCCAACAACAACAGATTGCCAATAT-AATTGGC-------------CAGTGGGGTAATAT----CAATCT----------------------------------------------------------------------------------------------------------------------TAATTTAAAAGT----------------------------- | |
| droWil2 | scf2_1100000004510:3964966-3964976 - | TGCTGCTCC-TG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | 4_group5:51407-51596 - | TGCTGCTGA----GC-------------GGACAGATCGAGTCGGTGGATGGACACAAAG------TGACGGTAAGCGAATGTGCGGTGTTCGGTGTTCGGTGTTCGATGTGCAACCAAAGTGGTC---AAGCTGGAAGATTGG--------CAAAGATTGCCCAGAT-ACAGGTA-------------CATTG-GGTA-----------------------ACGCTAATTAGACAATTTGGCCTGCATTGCGTATACT--------------------------------TGA------------------------------------------------T----------------------------- | |
| droPer2 | scaffold_5:4552034-4552216 - | TGCTGCTGA----GC-------------GGACAGATCGAGTCGGTGGATGGACACAAAG------TGACGGTAAGCGAATGTGCGGTGTTCGG-------TGTTCGATGTGCAACCAAAGTGGTC---AAGCTGGAAGATTGG--------CAAAGATTGACCAGAT-ACAGGTA-------------CATTG-GGTA-----------------------ACGCTAATTAGACAATTTGGCCTGCATTGCGTATACT--------------------------------TGA------------------------------------------------T----------------------------- | |
| droAna3 | scaffold_13340:10627323-10627416 - | GGCAGCAACATCAGCGGGGGTCTCAGTCGCAG--------------------------------------------------------------------TCATCGAA--GCGGCCGGAGTAGTCGG-------------------------------------------------------------------------------------------------CAAATCAGACAGTC-AACGGCCGTCGGCAAGACA--------------------------------ACA------------------------------------------------A----------------------------- | |
| droBip1 | scf7180000396741:1869037-1869081 + | GCGTATACT-T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATATTGCGTATACGCCCCATTGCCTTTGTACGA------------------------------------------------A----------------------------- | |
| droKik1 | scf7180000302277:981944-981950 + | TGCTGCT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454073:3595073-3595139 + | TCCTGATAA-TGGCC-------------A--CAAAATGCGGCAGTGGCTCGACCCAGAACCAACATCACGGTAAGTG------------------------------------------------GGCA------------------------------------------------------------------------------------------------------------------------------------------------------------------G-------------------------------------------------G----------------------------- | |
| droEle1 | scf7180000491249:3881363-3881390 + | AGTCATAAA-T--------------------TTA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTACTTTTATTAAA----------------------------- | |
| droRho1 | scf7180000779985:92747-92784 + | TGTTGCTGG-C--------------------CAGGGCGGCGCACCAGGCGGCCCCGGAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBia1 | scf7180000302529:943568-943573 - | TGCCGC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415158:355439-355469 + | GGCAACTTG-TTGGC------------TGGGTAAAAGGATCC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------G-------------------------------------------------G----------------------------- | |
| droEug1 | scf7180000409787:828692-828756 + | TCCTGATAA-TGGCC-------------A--CAAAATGCGGCAGTGGCTCGACCCAGAATCAGCATCACGGTAAGTG------------------------------------------------GGC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A----------------------------- | |
| dm3 | chr3R:24100567-24100620 - | TGTTGCCTT-TGAGT------------TTCGCACGCCTATTTGGCGAGTGCACACAGAG------AACCCGT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A----------------------------- | |
| droSim2 | 3l:20496206-20496223 + | CGCAGCCAA-T--------------------CAAATC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------G-------------------------------------------------A----------------------------- | |
| droSec2 | scaffold_11:832236-832253 + | CGCAGCCAA-T--------------------CAAATC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------G-------------------------------------------------A----------------------------- | |
| droYak3 | 2L:2414889-2414905 - | TGCTGCAGC-----------------------AACTC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GA------------------------------------------------T----------------------------- | |
| droEre2 | scaffold_4929:3383314-3383334 - | TGCTCCTCG-CAAAT-------------TGGCAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A----------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 07:45 PM