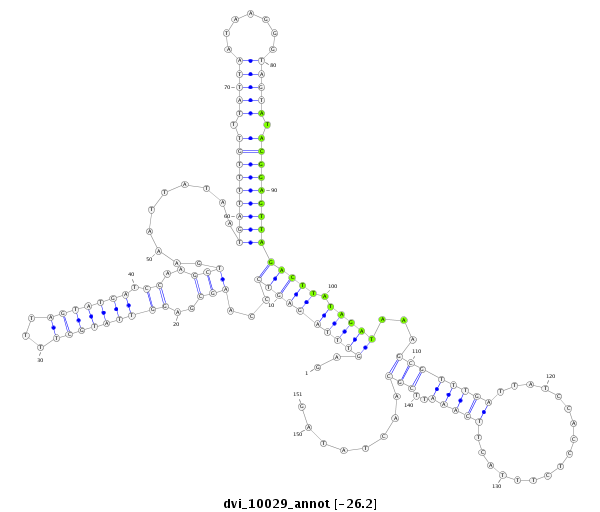
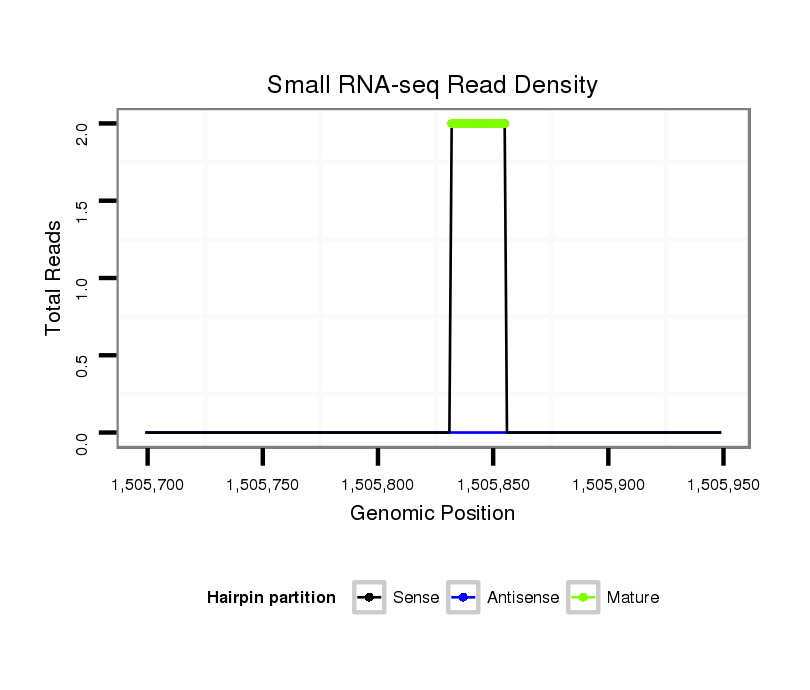
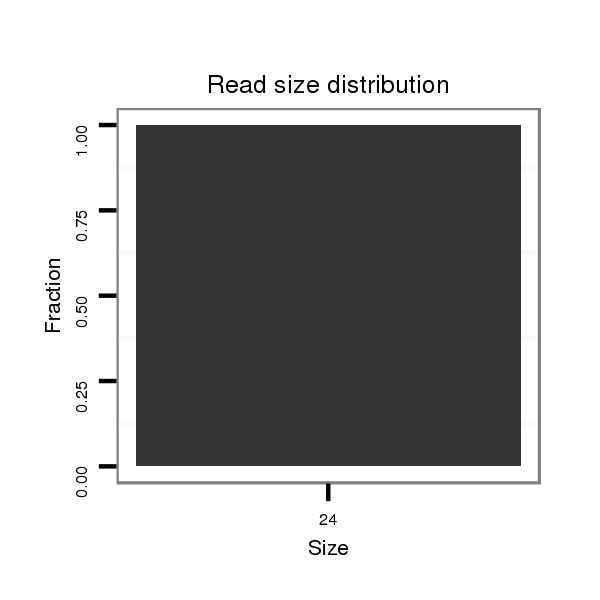
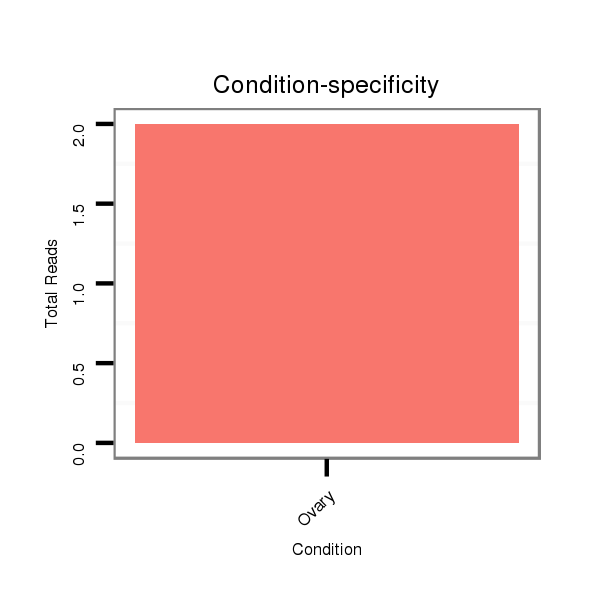
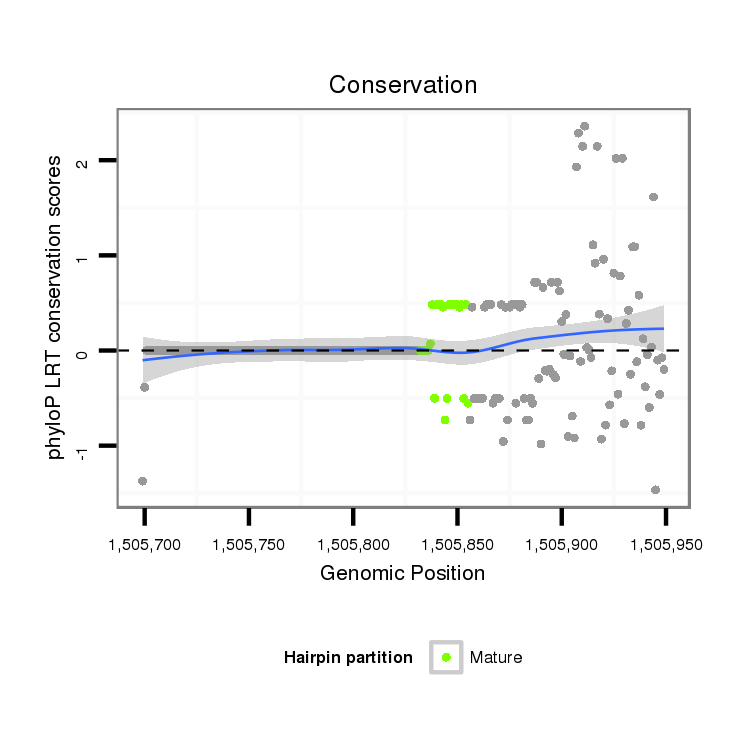
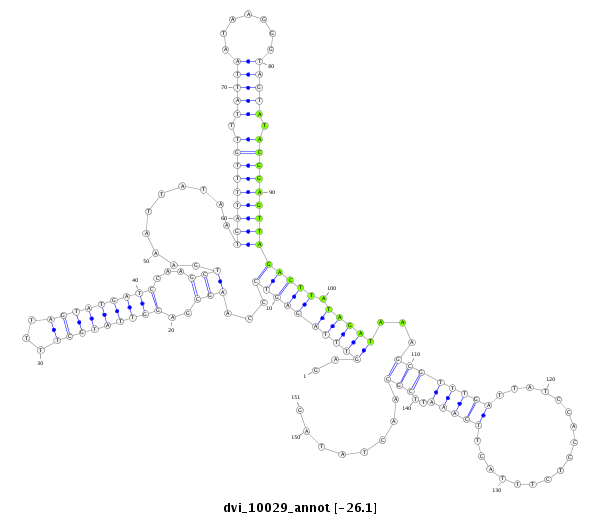
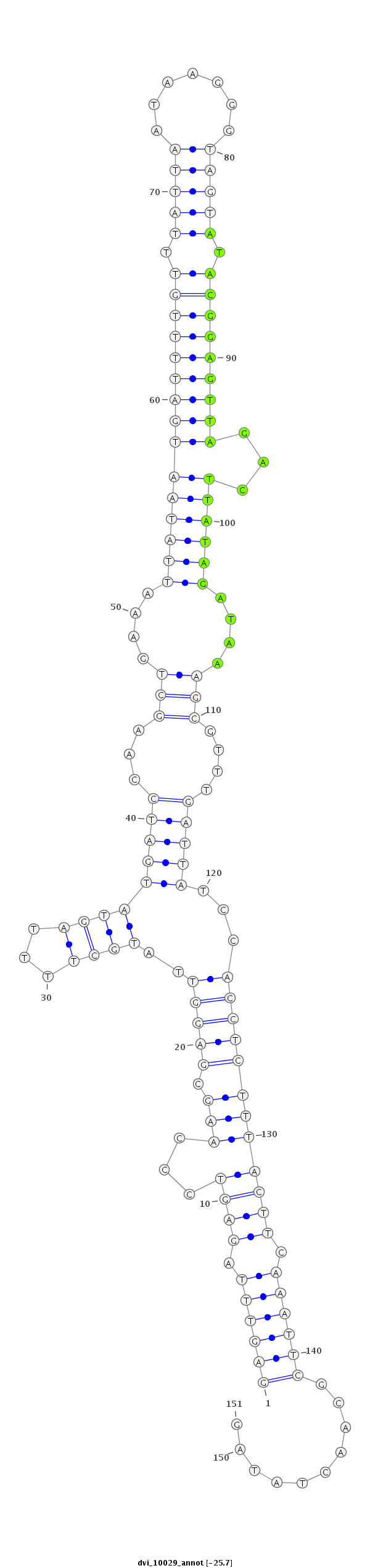
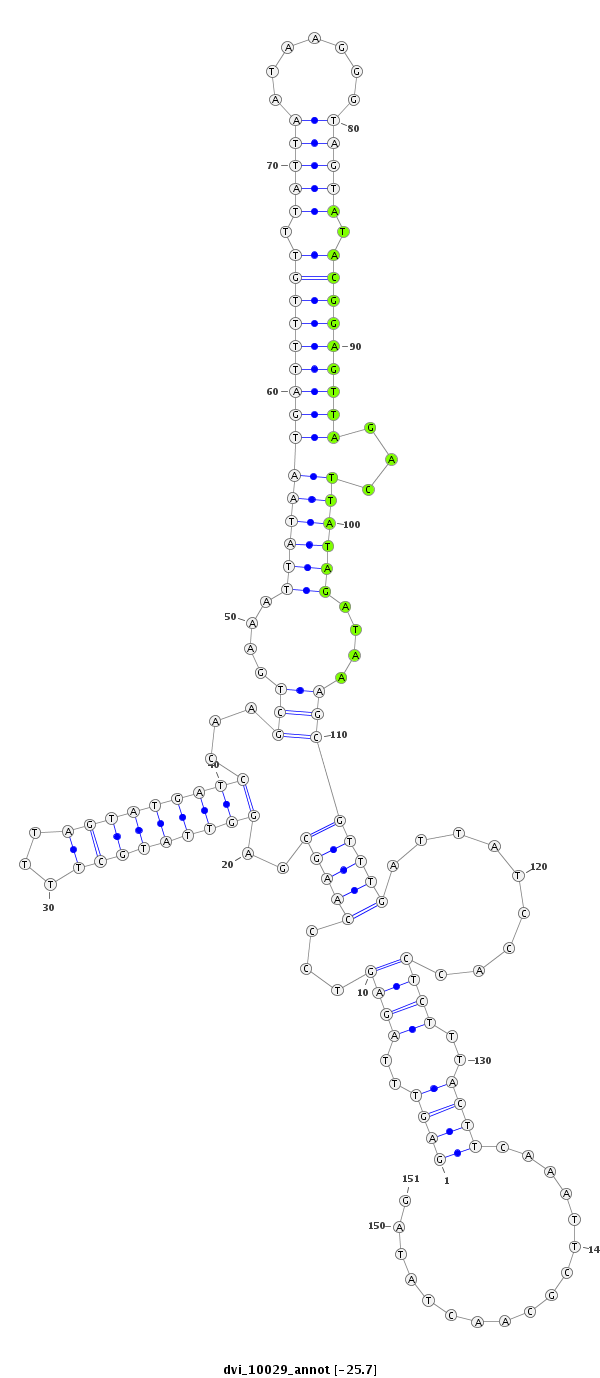
ID:dvi_10029 |
Coordinate:scaffold_12963:1505749-1505899 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -26.1 | -25.7 | -25.7 |
 |
 |
 |
CDS [Dvir\GJ14677-cds]; exon [dvir_GLEANR_1485:2]; intron [Dvir\GJ14677-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TTATTGAAAGGTTTTTTAAAGATCGAGTCGTTAGGAGAATTAAAGAGCGAGAGTTTAGAGTCCCAAGCGAGGTTATGCTTTTAGTATGATCCAAGCTGAAATTATAATGATTTTGTTTATTAATAAGGGTAGTATACGGAGTTAGACTTATAGATAAAGCGTTTGATTATCCACCTCTTTACTTCAAATTCGCAACTATAGTGCACATCATCAGCAACGGGCAGCTGCCGCCCACTATGCCCATTCGAGCT **************************************************..((((((((((...(((..(((((((((...))))))).))..)))..........(((((((((.(((((.......))))).)))))))))))))).)))))...((((((((.................)))))..)))........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060677 Argx9_ovaries_total |
M028 head |
SRR060684 140x9_0-2h_embryos_total |
V047 embryo |
GSM1528803 follicle cells |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................................................................................................ATACGGAGTTAGACTTATAGATAA.............................................................................................. | 24 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ........................................TAAAGAGCGACAGGTTAGACT.............................................................................................................................................................................................. | 21 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................GGGTCGTATACTGAGTTAG.......................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| .............................................................................................................................................................AGCGTTAGATTATTCACCCC.......................................................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................................................................................................................................................GGACATCATCAGCACCGGT.............................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ................................................................................................................................................GACTTATAGACAAAACGGT........................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
|
AATAACTTTCCAAAAAATTTCTAGCTCAGCAATCCTCTTAATTTCTCGCTCTCAAATCTCAGGGTTCGCTCCAATACGAAAATCATACTAGGTTCGACTTTAATATTACTAAAACAAATAATTATTCCCATCATATGCCTCAATCTGAATATCTATTTCGCAAACTAATAGGTGGAGAAATGAAGTTTAAGCGTTGATATCACGTGTAGTAGTCGTTGCCCGTCGACGGCGGGTGATACGGGTAAGCTCGA
**************************************************..((((((((((...(((..(((((((((...))))))).))..)))..........(((((((((.(((((.......))))).)))))))))))))).)))))...((((((((.................)))))..)))........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
M047 female body |
SRR060667 160_females_carcasses_total |
V047 embryo |
M027 male body |
V116 male body |
SRR060668 160x9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....ACTCTGCAAAAAATCTCTAG................................................................................................................................................................................................................................... | 20 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................GCAAGCTAACACGTGGAGAA........................................................................ | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................CCACGGCGGGTGCTACGGTTA....... | 21 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................TCCCGTGTAGATGTCGTTGC................................ | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................TCCCAGGGTTCGGTCCTAT................................................................................................................................................................................ | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................................................................................................................................................GCCAACTAACACGTGGAGAA........................................................................ | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................................................................................................................................CACCTGTAGAAGTGGTTGC................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:1505699-1505949 + | dvi_10029 | TTATTGAAAGGTTTTTTAAAGATCGAGTCGTTAGGAGAATTAAAGAGCGAGAGTTTAGAGTCCCAAGCGAGGTTATGCTTTTAGTATGATCCAAGCTGAAATTATAATGATTTTGTTTATTAATAAGGGTAGTATACG-GAGTTAGACTTATAGATAAAGCGTTTGATT----ATCCACCTCTTTACTTCAAATTCGCAACTATAGTGCACATCATCAGCAACGGG--CAGCTG---------CCGCCCACTATGCCCATTCGAGCT |
| droMoj3 | scaffold_6500:28406210-28406324 + | G------------------------------------------------------------------------------------------------------------------------------------------AAATTAGCTTTATAGACATCGTACCCGATTTAGTTCTTAGCGCTTAACTCACGTTTCACAACTGCAGTGC---ACATCAGCAAAGGT--CAGCAG---------CCGCCCACTACGGCCACTCGAGCT | |
| droGri2 | scaffold_8427:958-1019 + | A------------------------------------------------------------------------------------------------------------------------------------------A-----------------------------------------------------TTATCGTTTACAGTAC---ACACCAGCAACGGA--CACAAG---------CATCACACTATGCCCATGCCAGTT | |
| droWil2 | scf2_1100000004521:4021113-4021167 + | C------------------------------------------------------------------------------------------------------------------------------------------A------------------------------------------------------------------------TCATCATCAACGTC--CAGCCGGCGGCAACTCATCGCACTATGGCAATTCAGCTA | |
| dp5 | 4_group5:96841-96883 - | C---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATCATTCACGTG--CTGGAG---------CCTCTCATTACGGCAGTTCTTCCG | |
| droAna3 | scaffold_12916:9041508-9041553 + | TG----------------------------------------------------------------------------------------------------------------------------------------TA-----------------------------------------------------------------GTGGAAGTCATCATCCACGTT--CGGGAT---------CCTCTGCATAC---------TCCT | |
| droBip1 | scf7180000396535:1595637-1595680 + | T------------------------------------------------------------------------------------------------------------------------------------------A-----------------------------------------------------------------GTGGTAACCATCATCAACGTT--CTGGAT---------CCTCTGCGTAC---------TCCT | |
| droKik1 | scf7180000302684:1314364-1314391 + | T------------------------------------------------------------------------------------------------------------------------------------------G-----------------------------------------------------------------T---TGGCCACCATTCACGTT--CGAAT-------------------------------GCT | |
| droFic1 | scf7180000454082:244986-244997 - | C------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCATTA---------TAGCT | |
| droEle1 | scf7180000491046:3217631-3217674 + | T------------------------------------------------------------------------------------------------------------------------------------------A-----------------------------------------------------------------GTTCTAGTCACCATTCACGTC--CGGGAG---------CTTCCCACTAC---------AGCG | |
| droBia1 | scf7180000302408:656546-656594 - | T------------------------------------------------------------------------------------------------------------------------------------------A-----------------------------------------------------------------GGTCAAACCATCATTCACGTC--CTGGAG---------CTTCCCACTACAGCG----GAGAT | |
| droTak1 | scf7180000415873:295347-295397 - | C------------------------------------------------------------------------------------------------------------------------------------------A-----------------------------------------------------------------G----------CAGCGGCGGCTCTGGCGG-CC--GCCTCCTCGGCCAGCAGTCCGTCGGACT | |
| droEug1 | scf7180000409752:739211-739254 - | T------------------------------------------------------------------------------------------------------------------------------------------A-----------------------------------------------------------------GGTCAAACCACCAATCACGTC--CTGGAG---------CTTCCCACTAT---------AGTG | |
| droSec2 | scaffold_17:1138602-1138616 + | A------------------------------------------------------------------------------------------------------------------------------------------T------------------------------------------------------------------------------------------------------------------ACTCACCCAAACG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
Generated: 05/16/2015 at 07:37 PM