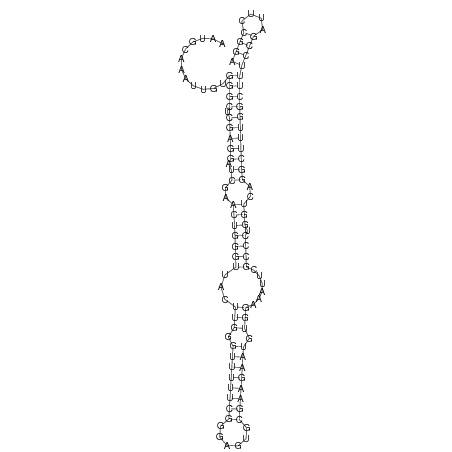
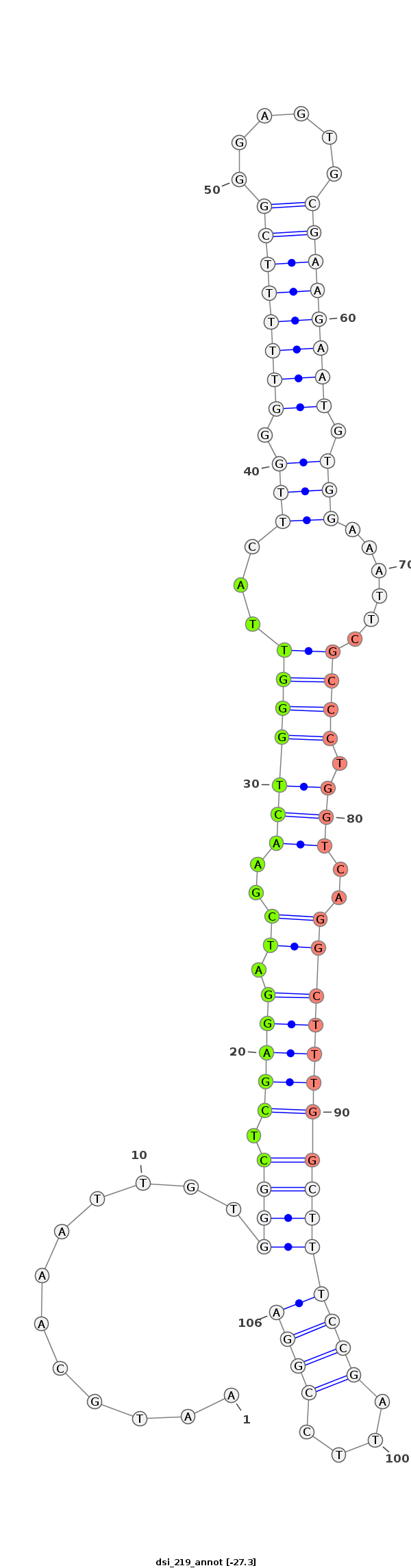
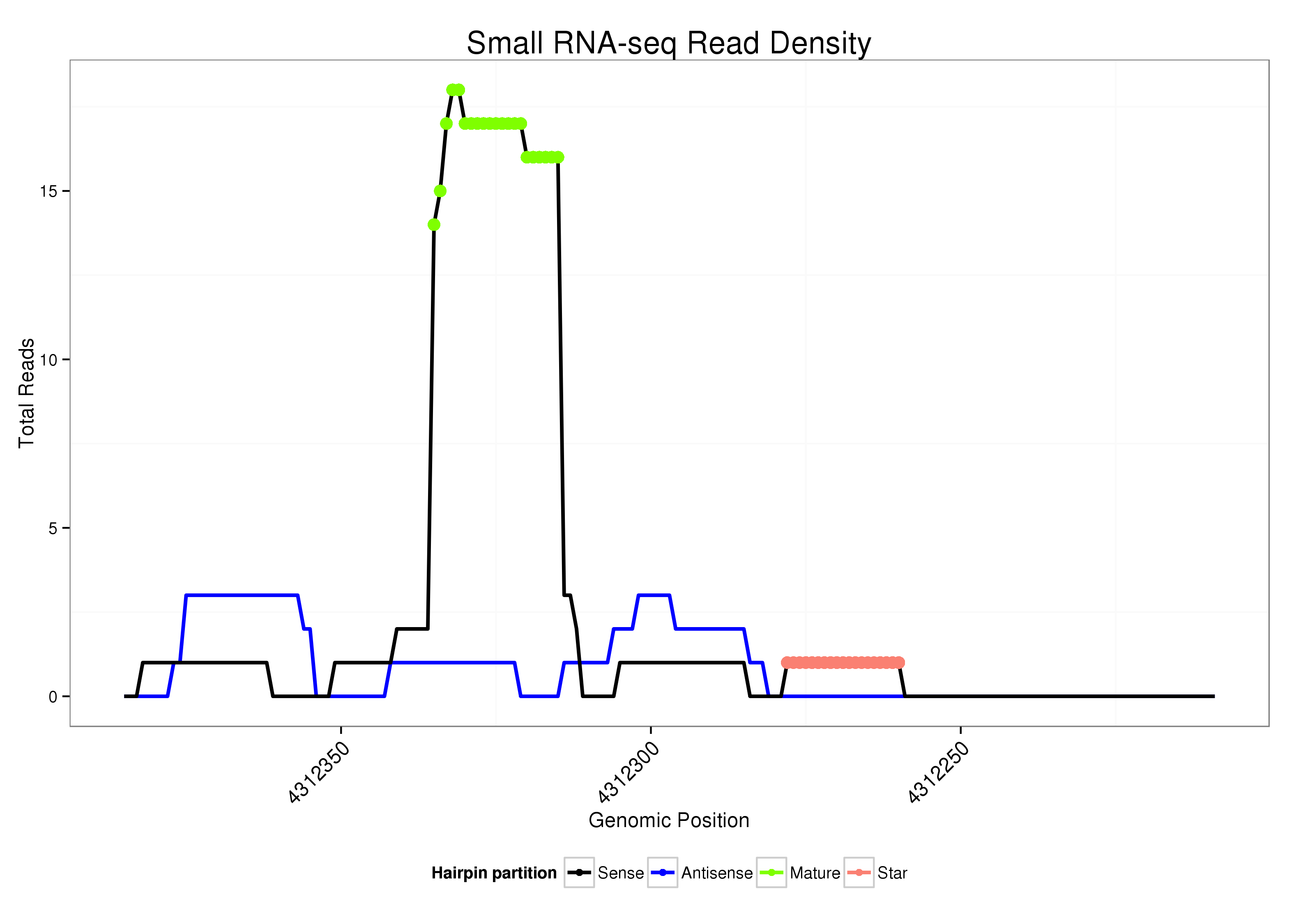
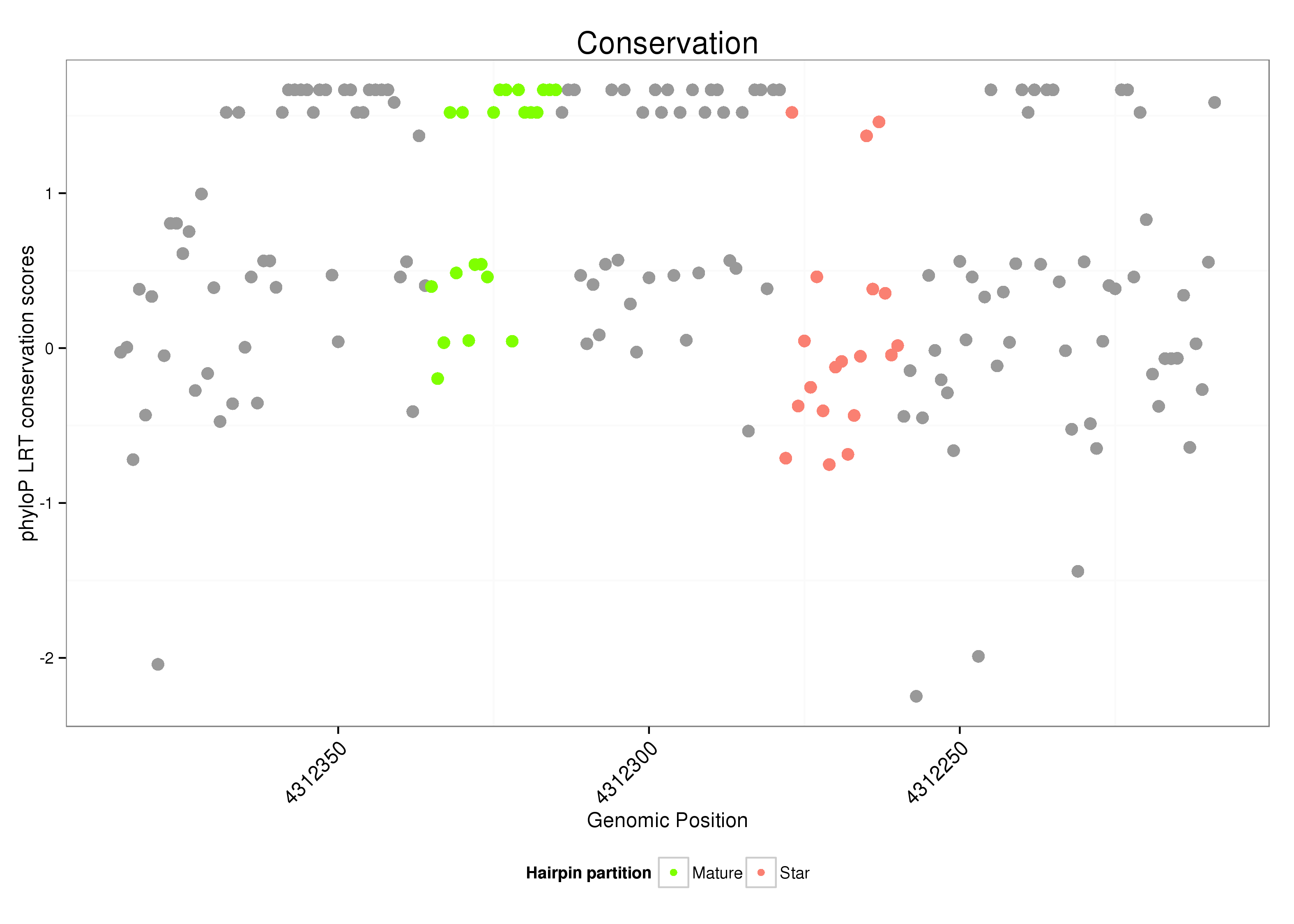
ID:dsi_219 |
Coordinate:2L:4312259-4312335 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
| -27.2 | -27.2 | -26.9 | -26.5 |
 |
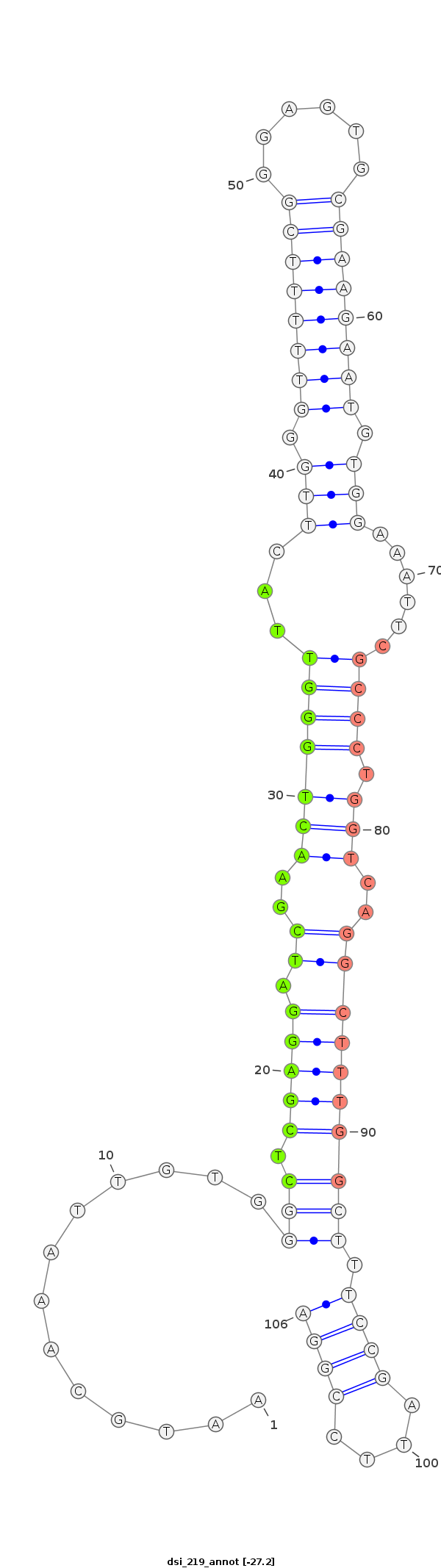 |
 |
 |
Antisense to intron [Dsim\GD23282-in]
No Repeatable elements found
|
GGGGGCCTTTGGTCGGTCGGGCGGGGCTTATCTTTAATGCAAATTGTGGGCTCGAGGATCGAACTGGGTTACTTGGGTTTTTCGGGAGTGCGAAGAATGTGGAAATTCGCCCTGGTCAGGCTTTGGCTTTCCGATTCCGGAGAATTGATAAGCGACGAGCTTACGACTCGATCGATT
***********************************............((((.(((((.((..(((((((...(((.((((((((......)))))))).)))......)))).)))..)))))))))))((((....))))************************************ |
Read size | # Mismatch | Hit Count | Total Norm | Total | M023 head |
M024 male body |
M053 Female-body |
SRR618934 Ovary |
|---|---|---|---|---|---|---|---|---|---|
| ..................................................CTCGAGGATCGAACTGGGTTA.......................................................................................................... | 21 | 0 | 1 | 12.00 | 12 | 7 | 5 | 0 | 0 |
| ...........GTCGGTCGGGCTGGG....................................................................................................................................................... | 15 | 1 | 6 | 1.50 | 9 | 0 | 0 | 0 | 0 |
| ...........................................................................................................CGCCCTGGTCAGGCTTTGG................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 |
| ...GGCCTTTGGTCGGTCGGGCGG......................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| ....................................................CGAGGATCGAACTGGGTTACT........................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| ..........GGTCGGTCGGGCTGGG....................................................................................................................................................... | 16 | 1 | 3 | 1.00 | 3 | 0 | 0 | 0 | 0 |
| ..................................TAATGCAAATTGTGGGCTCGA.......................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ....................................................CGAGGATCGAACTGGGTTACTT....................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ................................................................................TTCGGGAGTGCGAAGAATGTG............................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 |
| ............................................TGTGGGCTCGAGGATCGAACT................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .....................................................GAGGATCGAACTGGGTTACTT....................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ...............................CTTTAATGCAAATTGTGGGCG............................................................................................................................. | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ...................................................TCGAGGATCGAACTGGGTTA.......................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ....................................................CGAGGATCGAACTGGGTTACC........................................................................................................ | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................ATTACGGAGAACTGATAAGGG....................... | 21 | 3 | 4 | 0.75 | 3 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................TTACGGAGAACTGATAAGGGA...................... | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 |
| .....................................TGCAAATTGTGGGCT............................................................................................................................. | 15 | 0 | 4 | 0.50 | 2 | 0 | 0 | 0 | 2 |
| .........................................................................................................................................................GAAGAGCTTACGACTC........ | 16 | 1 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 |
| ........................................................................................................................................CCGGAGAACTGATAAG......................... | 16 | 1 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................TGACGGAGAACTGATAAGCGA...................... | 21 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 |
| .....................................TGCAAATTGTGGGCTT............................................................................................................................ | 16 | 1 | 13 | 0.08 | 1 | 0 | 0 | 0 | 1 |
| ............................................................................GTTTATTGGGAGTGCGA.................................................................................... | 17 | 2 | 18 | 0.06 | 1 | 0 | 0 | 0 | 1 |
| .GGGGGCTTTGGACGCTCGG............................................................................................................................................................. | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 1 | 0 |
| ...........GTCGGTCGTGAGGGCCTT.................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 |
| ...............................................................................................................................................CTTGATAACCGACGAG.................. | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................GCTTACGACTCGTCCG... | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 |
|
AATCGATCGAGTCGTAAGCTCGTCGCTTATCAATTCTCCGGAATCGGAAAGCCAAAGCCTGACCAGGGCGAATTTCCACATTCTTCGCACTCCCGAAAAACCCAAGTAACCCAGTTCGATCCTCGAGCCCACAATTTGCATTAAAGATAAGCCCCGCCCGACCGACCAAAGGCCCCC
************************************((((....))))(((((((((((..(((.((((......(((.((((((((......)))))))).)))...)))))))..)).))))).))))............*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M023 head |
M024 male body |
O001 Testis |
O002 Head |
|---|---|---|---|---|---|---|---|---|---|
| ..........GTCGTAAGCTCGTCGCTTATC.................................................................................................................................................. | 21 | 0 | 1 | 2.00 | 2 | 0 | 2 | 0 | 0 |
| ...........................................TCGGAAAGCCAAAGCCTGACC................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ........GAGTCGTAAGCTCGTCGCTTA.................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ...............................................................................ATTCTTCGCACTCCCGAAAAAC............................................................................ | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 |
| ...................................................................................TTCGCACTCCCGAAAAACCCA......................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 |
| .......................................................................ATTTCCACATTCTTCGCA........................................................................................ | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 |
| .........................................................CCTGAGCAGGGCGAA......................................................................................................... | 15 | 1 | 5 | 0.60 | 3 | 0 | 0 | 0 | 0 |
| .................................................AGCCAAAGCCTGACCAGGGT............................................................................................................ | 20 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 |
| ...............................................AATGACAAAGCCGGACCAGGG............................................................................................................. | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 |
| ............CGTAAGCCCGTCGCTA..................................................................................................................................................... | 16 | 2 | 18 | 0.17 | 3 | 0 | 0 | 0 | 0 |
| .........................................AATCGGAAAGACAAACC....................................................................................................................... | 17 | 2 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 |
| ................................................................................................................AGTTCGACCCTCGGGACCA.............................................. | 19 | 3 | 17 | 0.12 | 2 | 0 | 0 | 0 | 0 |
| .................................................................................TCTTCGCAGACCCGAAAA.............................................................................. | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................................CCCAAGTAGCTCAGTTGGAT......................................................... | 20 | 3 | 13 | 0.08 | 1 | 0 | 1 | 0 | 0 |
| .......................................................TGCCTGAGCAGGGCGAA......................................................................................................... | 17 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................AATGTGCATTAAAGA.............................. | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim3 | 2L:4312209-4312385 - | dsi_219 | GGGGGCCTTTGGT----------------------CG------GTCGGGCGGGGCTTATCT----TTAATGCAAATTGTGGGCTCGAGGATCGAACTGGGTTACTTGG-G----------TTTTTCGGGAGTGCGAAGAATGTGGAAATTCGCCCTGGTCAGGCTTTGGCTTTCCGATTCCGGAGAATTGATAAGCGACGAGCTTACG-------------------------ACTCGATC----------GATT |
| droSec2 | scaffold_5:2487449-2487626 - | GGGGGCCTTTGGT----------------------CG------ATCGGGCGGGGCTTATCTT---TTAATGCAAATTGTGGGGTCGAGGATCGAACTGGGTTACTTGG-G----------TTTTTCGGGAGTGCGAAGAACGTGGAAATTCGCCCTGGTCAGGCTTTGGCTTTCCGATTCCGGAGAATTGATAAGCGACGAGCTTACG-------------------------ACTCGATC----------GATT | |
| dm3 | chr2L:4389206-4389383 - | GGGGGCCTTTGGT----------------------CG------GTCGGGCGGGGCTTATCT----TTAATGCAAATTGTGGGCTCGAGGATCGAACTGGGTTACTTGGAG----------TTTTTCGGGAATGCGGGGAACGTGGAAATTCGCCCTGGTCAAGCTTTGGCTTCCCGATTCCGGAGAATTGATAAGCGATGAGCTTACG-------------------------ACTCGATC----------GATT | |
| droEre2 | scaffold_4929:4471465-4471633 - | GGGGCC-TTT--------------------------G------GGCGGGCG--GCTTATCT----TCAATGCAAATTGTCGGCACGAGGATCGAACTGGGTTACTTGGTG----------TTTCTCTGCAGTGCAAAGAACATGGAAATTCGGACTGGTCAAGCT--GACTGTCCGATTCCGGAGAATTGATAAGCACCGAGCTTACG-------------------------ACTCGATC----------GATT | |
| droYak3 | 2L:4422519-4422683 - | TT----------------------------------G------GTCGGGCG--GCTTATCT----TCAATGCAAATTGTGGGCTCGAGAATCGAACTGGGTTACTTGTGG----------TTTTTCTGGAGTGCGAAGAACATGGAAATTTGGCCTGGTCAAGCTTTCGCTGTCCAATTCCAGAGAATTGATAAGCGCCGAGCTTACG-------------------------ACCCGATC----------GATT | |
| droEug1 | scf7180000409005:261816-261992 - | GGTGGC-----G----------------------GCG------GTCAGGCG--GCTTATCT----TCAATGCAAATTGTGGGCTCGAGGATCGAACTGGGTTACTTATGG----------ATTTTCGGGAGTACGAAGAACATGAAAATTTGCCCTAACCAATCTTTGGCTCTTCACGTCCGGAGAATTGATAAGCG---TGCTTACG-------------AA---ATCAT---------CGAA-CATTTCGTAT | |
| droBia1 | scf7180000302188:921986-922157 - | GGTGGC------------------------------T------GTCGGGCG--GCTTATCT----TCAATGCAAAT-GTGGGCCCGAGGATCGAACTGGGTTACTTGGGG----------ATTTTCGGGAGTGCGAAGAACATGGAAATTGGACTTTTTCGCGCTTTGGGCCCCCAATCCCAGAGAGTTGATAAGCG---CGCTTACG-------------AA---ACCAT---------CGGA-AATTTCGTA- | |
| droTak1 | scf7180000415264:445341-445517 - | GGTGGCG---------------------------GCT------GTCGGGCG--GCTTATCT----TCAATGCAAATTGTAGGCTCGAGGATCGAACTGGGTTACTTGGGG----------ATTTTCGGGAGTGCGAAGAACATGAAAATTTGCCCTGATCGAGCTTTGGGCCCCCAATTCCGGAGAGGTGATAAGGG---TGCTTACG-------------AA---ATCAT---------AGGA-AATTTCTAAT | |
| droEle1 | scf7180000490767:68840-69026 - | GGGGTCATTT----------ATTGGGTGGCGTTGGCT------GTCAGGCG--GCTTATCT----TCTATGCAAATTGC--TCTAGAGGATCGAACTGGGTTACTTGGGG----------ATTTTCGGGAGTGCGAAGAACATGGAAATTTGCCTTGG-----------C-TCCCAAATACGGACAATTGATAAGCCGCCTACTTACG-------------ACAGCATCAT---------CGAATAATGCCGTAT | |
| droRho1 | scf7180000766469:36927-37112 + | GGGGGCTTTTGGAGCTATTTATTGGGAGGCG---GCTGT----GACAGACT--GCTTATCT----TCTATGCAAATTGT----TCGAGGATCGAATTGGGTTACTTGGGG----------ATTTTCGGGAGTGCAAAGAACATGGAAATTTGCTC-------------GGTGCCCGATTTCCGACACTTGATAAGCC---TGCTTACG-------------AA---ACCAT---------TAGA-AATGTGTTAT | |
| droFic1 | scf7180000448917:1035-1216 + | GGTGGCGATTGGA-------------------------------CCTGCCT--GCTTATCTATCGTCTATGCAAATTGTGGGCTCGAGGATCGAACTGGGTTACTTGG-G----------ATTTTCGGGAGTGCGAAGAACATGAAAATTCGCCCTCGTTCGGCTTTAGCTCACCAGCTCCCGAGAATTGATAAGCG---TGCTTACG-------------AA---ACGAT---------CGGA-AATTTCGAAT | |
| droKik1 | scf7180000302473:582768-582985 + | CTGTTG--------------CTTGGGTGGCG---GCGGCAACGGCCAGGCGGGGCTTATCT----TCTATGCAAATTGTGGGCTCGAGGATCGAACTGGGTTACTTGGGGATTGGATTCGATTTTTGGGAGTGCGAAGAACACGAAAGTTTGTCCAGGCCGAGCCTAGGCAGCCTAATTCCCGAATATTGATAAGCA---GGCCTACGTATTGTGGAATGGGA---ATCAT---------GGAA-AAGTTCGTAT | |
| dp5 | 4_group3:9370059-9370232 + | GGTGGC------------------------------G------GCCAGGTT--ACTTATCT----TCAATGCAAATTATAGGCCTGCGATCTGAATTGGGTTACTTGGAT----------TCTTTTGGGAGTGCGACGAACATGAAAGTTTGCCTTGACAAAACTTTGAAAGTCGACCTCTTCAATATTGACAAGAA---AAACTATG-------------GA---ACTTTTGAATTAT----------TCGTAT | |
| droPer2 | scaffold_8:560711-560884 + | GGTGGC------------------------------G------GCCAGGTT--GCTTATCT----TCAATGCAAATTATAGGCCTGCGATCTGAATTGGGTTACTTGGAG----------TCTTTTTGGAGTGCGACGAACATGAAAGTTTGCCTTGACAAAACTTTGAAAGTCGACCTCTTCAATATTGACAATAA---AAACTATG-------------GA---ACTTTTGAATTAT----------TCGTAT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
Generated: 12/06/2013 at 02:35 PM