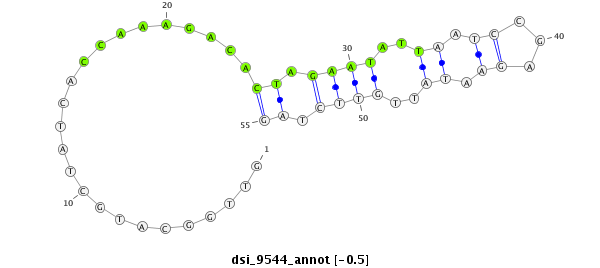
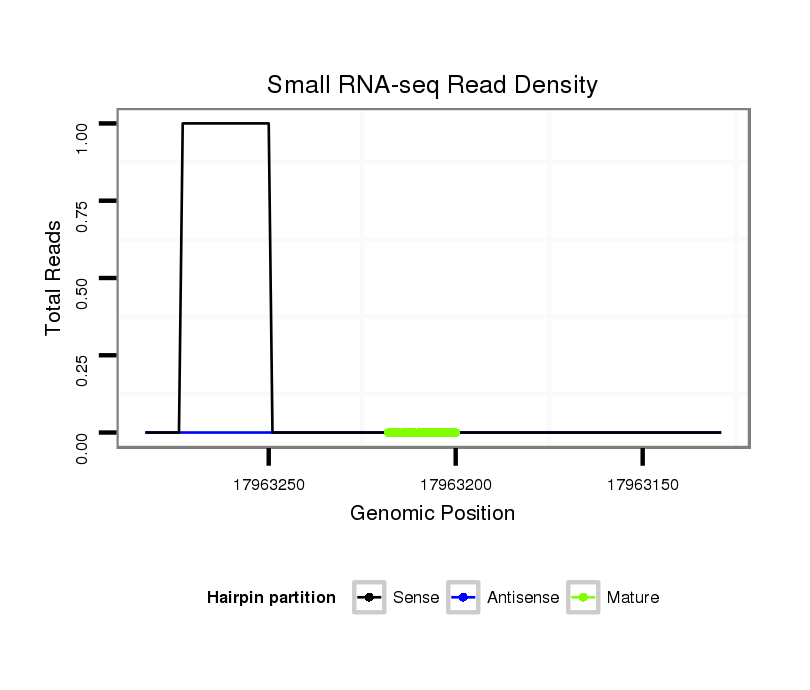
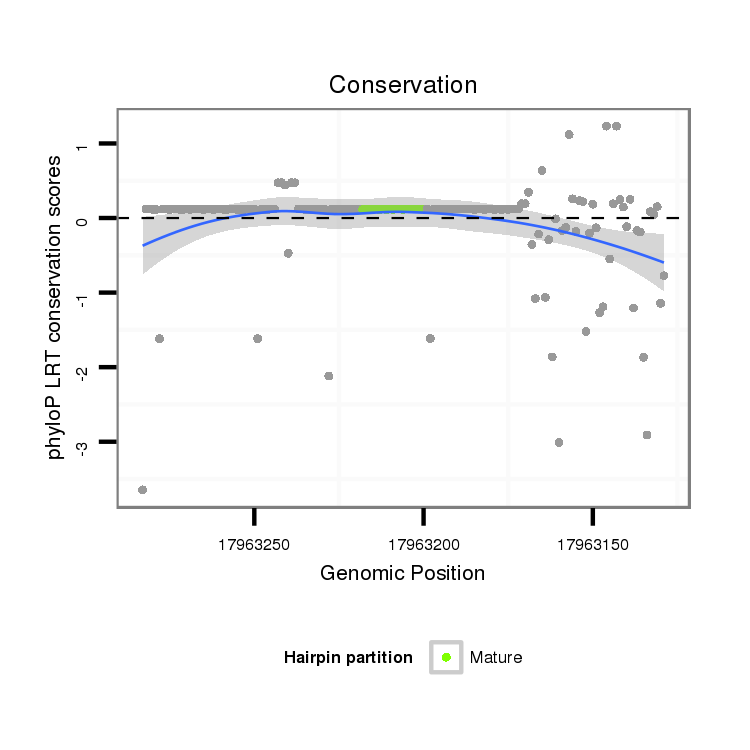
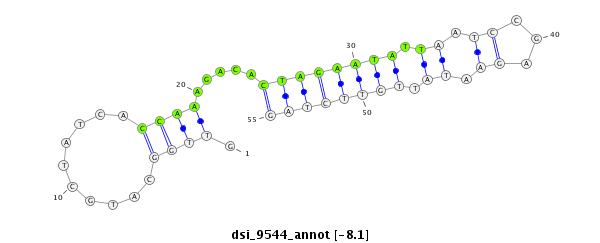
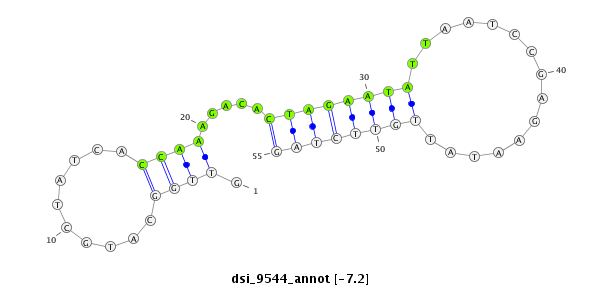
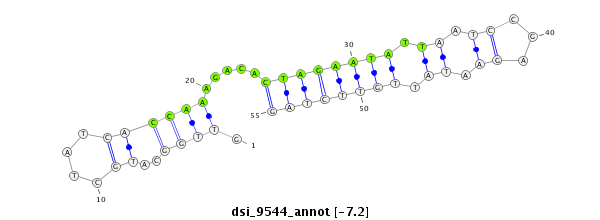
ID:dsi_9544 |
Coordinate:2r:17963179-17963233 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -8.1 | -7.2 | -7.2 |
 |
 |
 |
intergenic
No Repeatable elements found
|
GTTGGCATGATCCAGATACAACAAGCAACCGAAACGCCAAATGGAAAACGGTTGGCATGCTATCACCAAAGACACTAGAATATTAATCCGAGAATATTGTTCTAGTGTCTTTGCTATCACCGAGCAGTCTTCGCGCAAACATGAAAGTGTTCAAT
**************************************************........................((.((((..((.((...)).))..)))).))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M023 head |
SRR618934 dsim w501 ovaries |
SRR553487 NRT_0-2 hours eggs |
SRR553486 Makindu_3 day-old ovaries |
SRR553485 Chicharo_3 day-old ovaries |
SRR553488 RT_0-2 hours eggs |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ..........TCCAGATACAACAAGCAACCGAAA......................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........TCCAGATACAACAAGCACCCGAAA......................................................................................................................... | 24 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............ACACAACAAGCAACCGAA.......................................................................................................................... | 18 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................GAGCATCTTGTTCTAGTGT............................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................TCCAAAGACACTAGAATATAAAT.................................................................... | 23 | 2 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................GCAAACATGAAAGTGG..... | 16 | 1 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................TGGAAGACAGTAGGCATGC............................................................................................... | 19 | 3 | 14 | 0.14 | 2 | 0 | 0 | 0 | 1 | 1 | 0 |
| ................................................CAGTAGGCATGCTATGACC........................................................................................ | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ............................................................TATTACCAAAGACACTAGAACACTA...................................................................... | 25 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................................................CCAAAGACACTAGAATATT....................................................................... | 19 | 0 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
|
CAACCGTACTAGGTCTATGTTGTTCGTTGGCTTTGCGGTTTACCTTTTGCCAACCGTACGATAGTGGTTTCTGTGATCTTATAATTAGGCTCTTATAACAAGATCACAGAAACGATAGTGGCTCGTCAGAAGCGCGTTTGTACTTTCACAAGTTA
**************************************************........................((.((((..((.((...)).))..)))).))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR618934 dsim w501 ovaries |
SRR553486 Makindu_3 day-old ovaries |
SRR553487 NRT_0-2 hours eggs |
SRR553485 Chicharo_3 day-old ovaries |
M023 head |
GSM343915 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................ATCTCATAATTAGGCTCAT............................................................. | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................GTCATCCGTACGATACTGGT....................................................................................... | 20 | 3 | 5 | 0.40 | 2 | 0 | 0 | 2 | 0 | 0 | 0 |
| ............................................................................................TTATATCAAGATCACAGAAACCAT....................................... | 24 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................................TCCGTGCGATAGTGGTT...................................................................................... | 17 | 2 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................AGTGGTTTCTGTGATCTTA.......................................................................... | 19 | 0 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................TCACAAGCGCATTGGTACT........... | 19 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| .AACCGGACTAGGTCTA.......................................................................................................................................... | 16 | 1 | 7 | 0.14 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................TTTATATCAAGATCACAGAAACCA........................................ | 24 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................GGATAGTGGCTCGGCAAAAG....................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................GGTTAGTACTTTCACAAGGT. | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| .........................................................................GGATCTCATAATTAGG.................................................................. | 16 | 2 | 20 | 0.10 | 2 | 0 | 0 | 2 | 0 | 0 | 0 |
| ...............................................................GTGGTTTCTGTGATCTTA.......................................................................... | 18 | 0 | 20 | 0.10 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................GTGGTTTCTGTGATCTTAT......................................................................... | 19 | 0 | 20 | 0.10 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................GGTTTGTACTTTCAC...... | 15 | 1 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................AGGGTTTGTACTTTCAC...... | 17 | 2 | 19 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................TAATTGCGCTCTTCTAACA....................................................... | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................GATCTCATAATTAGGTT................................................................ | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | 2r:17963129-17963283 - | dsi_9544 | GTTGGCATGATCCAGATACAACAAGCAACCGAAACGCCAAATGGAAAACGGTTGGCATGCTATCACCAAAGACACTAGAATATTAATCCGAGAATATTGTTCTAGTGTCTTTGCTA-----------TCACCGAGCAGTCTTCGCGCAAACATGAAAGTGTTCAAT |
| droSec2 | scaffold_9:737698-737863 - | GTTGGTATGATCCAGATACAACAAGCAACCGAAATGCCAAATGGAAAACGGTTGGGATGCTATCACCAAAGACACTAGAATATTATTCCGAGAATATTGTTCTAGTGTCTTTGCTATCACCGAGCCACCACCTAGCAGTCTTCGCGCAAACATGAATGTGTTCAAT | |
| dm3 | chr2R:17409324-17409367 - | T---------------------------------------------------------------------------------------------------------------GCTA-----------TCACCGATCAGTCTTCGCGCAACCATGGAAGTGATCAAT | |
| droEre2 | scaffold_4845:11553207-11553245 - | T-------------------------------------------------------------------------------------------------------------------------------CACCAAGTAGTCTTCGCAAAATCATGAAAGTAGTCAGT | |
| droYak3 | 2R:11770608-11770649 + | C-----------------------------------------------------------------------------------------------------------------TG-----------TTAGCAACCAGACTTTGCAAAAATATGGCAATGGTCAAG | |
| droEug1 | scf7180000409474:455780-455810 - | ---------------------------------------------------------------------------------------------------------------------------------------CAGTCTGCTCAGTAACATGAACGCCTTCGTG | |
| droBia1 | scf7180000301754:3677796-3677836 + | T------------------------------------------------------------------------------------------------------------------A-----------CCATAGCTCAGTCGTCGCAAGAACATGAAAGAACTCACA | |
| droTak1 | scf7180000415138:406439-406473 - | A---------------------------------------ATGAAA---------------------------------------------------------------------------------------AATGGTTTTGATATTATCACCAAAA-----ACA | |
| droEle1 | scf7180000491201:1739689-1739723 + | C-----------------------------------------------------------------------------------------------------------------------------------AAACAGTTTTGGCAAAAACATGAAGGTCTATATA | |
| droRho1 | scf7180000780066:520675-520705 + | ---------------------------------------------------------------------------------------------------------------------------------------CAGTCTTTGCAATAACATGAAAGTCGTCATA | |
| droFic1 | scf7180000453201:172448-172473 + | G--------------------------------------------------------------------------------------------------------------------------------------CGGTCTTCGCTTAAACATGAAGGTT------ |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
Generated: 05/18/2015 at 07:15 PM