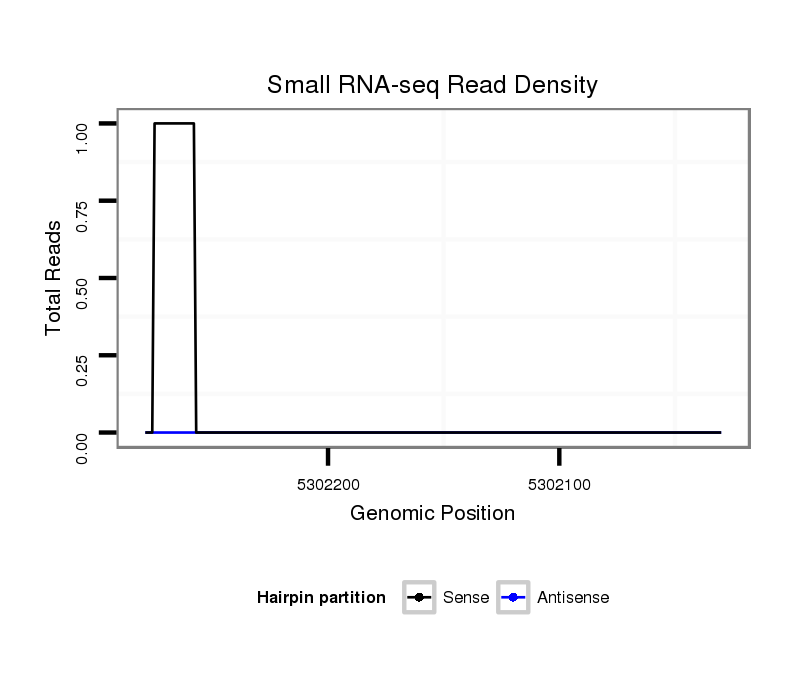
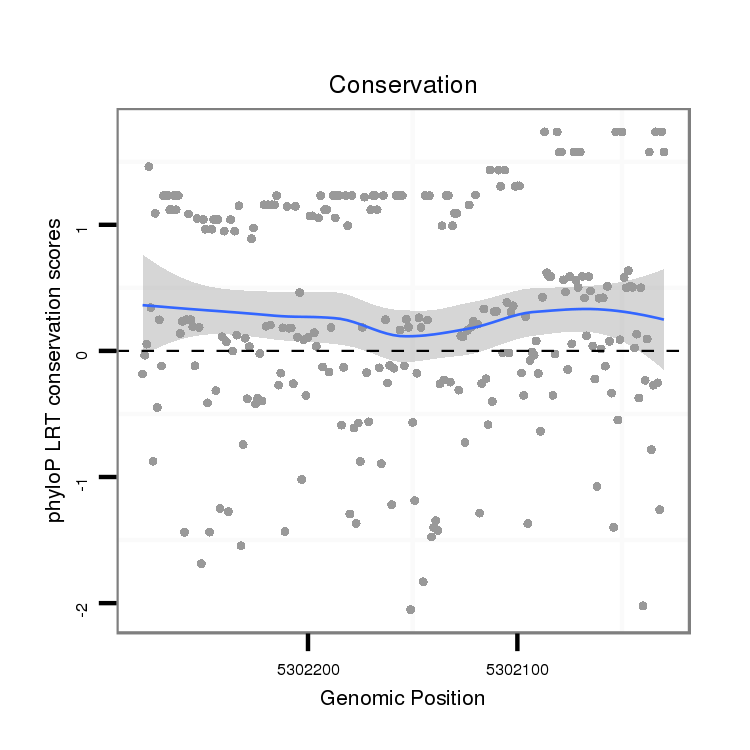
ID:dsi_5717 |
Coordinate:x:5302080-5302229 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [x_5301654_5302050_-]; five_prime_UTR [x_5302051_5302079_-]; exon [x_5301654_5302079_-]; intron [x_5302080_5304673_-]
No Repeatable elements found
| --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## ACAAAATGAAAATCGTGACACTCATTGTTTTCGTATTGAGCCACGTTAGAAGCTATCTAATATTTGGCTAGTTAGGGGGTTTTCCAAGGCCAGTTTTTCCTCTAGCACTGATCGCAAGTTAATTAATGAAGCCGCTTTCGGTTGATAACATCGTGCTGATGGCTTTTCGCTGTTTACTGATAACCGAATCACTTTTCCAGGTCGATCTGCTGATAGCAATAACGCGACGATGACGCAAAAAACCCTGTTG **************************************************(((((.........))))).(((((((((..................)))))))))..((((((..(((.(((....(((((((...(((((((((...)))).)))))))))))).....))).)))......)).)))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M025 embryo |
SRR553488 RT_0-2 hours eggs |
SRR618934 dsim w501 ovaries |
SRR553485 Chicharo_3 day-old ovaries |
GSM343915 embryo |
|---|---|---|---|---|---|---|---|---|---|---|
| ....AATGAAAATCGTGACACT.................................................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................AGCACTGATCGCAAC.................................................................................................................................... | 15 | 1 | 9 | 0.11 | 1 | 0 | 0 | 1 | 0 | 0 |
| ................................................................................................................................................................................................TTTTCCAGGTCCATC........................................... | 15 | 1 | 20 | 0.10 | 2 | 0 | 2 | 0 | 0 | 0 |
| ......................................................................................................TGGCACTGATCGCAA..................................................................................................................................... | 15 | 1 | 14 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 |
| ............................................................................................................................................................................TGTACTGATAACCGCAGCA........................................................... | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................TGATTTAATGAAGCCGCATT................................................................................................................ | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
|
TGTTTTACTTTTAGCACTGTGAGTAACAAAAGCATAACTCGGTGCAATCTTCGATAGATTATAAACCGATCAATCCCCCAAAAGGTTCCGGTCAAAAAGGAGATCGTGACTAGCGTTCAATTAATTACTTCGGCGAAAGCCAACTATTGTAGCACGACTACCGAAAAGCGACAAATGACTATTGGCTTAGTGAAAAGGTCCAGCTAGACGACTATCGTTATTGCGCTGCTACTGCGTTTTTTGGGACAAC
**************************************************(((((.........))))).(((((((((..................)))))))))..((((((..(((.(((....(((((((...(((((((((...)))).)))))))))))).....))).)))......)).)))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR553488 RT_0-2 hours eggs |
SRR618934 dsim w501 ovaries |
SRR902008 ovaries |
SRR553486 Makindu_3 day-old ovaries |
M024 male body |
|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................................AAGGAGATCGTGTCTA.......................................................................................................................................... | 16 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................AAGGTCCAGCAAGACG........................................ | 16 | 1 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................CAGAACTCGGTGCAATGT........................................................................................................................................................................................................ | 18 | 2 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................AAAAGGACCTGCTAGAAGACT..................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................CCCTCAAAGGTACCGGTCAAA.......................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..............................TGCAGAACTCGGTGCAATG......................................................................................................................................................................................................... | 19 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..............................TGCAGAACTCGGTGCAA........................................................................................................................................................................................................... | 17 | 2 | 7 | 0.14 | 1 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................AAAGGACCTGCTAGAAGACT..................................... | 20 | 3 | 17 | 0.12 | 2 | 0 | 0 | 2 | 0 | 0 |
| .......................................................................................................................................AAAGCCACCTATTGT.................................................................................................... | 15 | 1 | 11 | 0.09 | 1 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................CGACAGATGACTAGTGGCC............................................................... | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 |
| ..................................................................................................................................................................................................AAGGTCTAGCAAGACGATTA.................................... | 20 | 3 | 13 | 0.08 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................................................ACCGATCTAGCCCCCAAAG....................................................................................................................................................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................................................................................................................AAAGGTCTAGCAAGACGA....................................... | 18 | 2 | 19 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................CGAACAGCGGCAAATG......................................................................... | 16 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | x:5302030-5302279 - | dsi_5717 | ACAAAATGAAAATCGTGACACTCATTGTTT-TCGT---ATT-GAGCCACGTTAGAAGCTATCTA-----ATATTTGG-CTAGTTA---GGGG-GT---TTTCCAAGGCCAGTTTTTCCTCTAGCA---CTGATCGCAAGTTAATTAATGAAGCCGCTTTC---GG------TTGATAACATCGTGCT-------GATGGCTTTTCGCTGTTTACTGATA------------ACC--GAATCACTTT-TCCAGGTCGATCTGCTGATAGC------A---ATAAC---GCGA---CGATGACGCAAAAAACC------CTGTTG |
| droSec2 | scaffold_4:1102039-1102287 + | ACAAAATGAAAATCGTGACA--CATTGTGT-TCGT---ATT-GAGCCACGTTAGAAGCTATCTA-----ATATTTGGGCCAGTTA---GGGG-GT---TTTCCAAGGTCAGTTTTTCTTCTAGC----CTGATCGCAAGTAAATTAATGTAACTGCTTTC---GG------TTGATAACATCGTGCT-------GATGGCTTTTCGCTGTTTACTGATA------------ACC--GAATCACTTTTTCCAGGTCAATCTGCTGATAGC------A---ACAAC---GCGA---CGATGGCGCAAACAACC------CTGTTG | |
| dm3 | chrX:5661707-5661960 - | ACAAAATGAAAATCGTGACACTCATTGTTT-TCGT---ATT-GAGACACGTTAGAAGCTATCTA-----ATATTTGG-CCAGTAACT-G-GG-GT---TTTCCAAGGCCAGTTTTTCCTTTAGCACTACTGATCGCAAGTTAATTAATGAAGCTGTTTTC---GG------TTGATAACATCGTGCT-------GATGGCTTTTCGCTGTTTACTGATT------------ACC--GAATCACTTT-TCCAGGTCAATCTGCTGATAGC------A---ACAAC---GCGA---CAATGACGCAAAAAACT------ATGTTG | |
| droEre2 | scaffold_4690:3010470-3010721 - | ACAATATGAAAATCGTGACACT-ATAGTGT-TCGT---ATT-AAGCTACGCTAGA-------TA-----ATATTTGT-CCAATAACT-G-CG-GT---TTTCCAAGGCCAGTTTTTCCTTTACCA---ATGATCAGAAGCTAAT-AATGCAGCTGTTTTT---GGGTGA--TTGTTATCATCGTGCT-------GATGACTTTTCGCTGTTTACTGATT------------GCC--GCGTCACTTT-TCCAGGTCAACCTGCTGATAGC------A---ACAAC---GCGA---AGATGACGCAGAAAACTCAACTGCTGATG | |
| droYak3 | X:2811539-2811813 + | ACAATATGAAAATCGTGACACACATTGTAT-TCATCCTATTAAAGCCACGTTAGATGCTAACTA-----ATGTTTGA-CCAATAACT-G-TGGGTTGGTTACCAAGGCCAGTTTCTCCTTTAGCA---CTGATCAGAAGTTAATTAATGAAACTGCTTTTTACGC------GTGATAACATTGTGCT-------GATGGCTTTTCGCTGTTTACTGATTACCACCG-----ACC--GAATCACTTT-TCCAGGTCAACCTGCTGATAGC------A---ACAAA---GCGA---AAATGACGCAAAAAACTCAACTGCTGATG | |
| droEug1 | scf7180000409061:33664-33927 + | CAAATTTCTGAATCGTGACACTCATTGTTTTTTAT---ATT-AAGCCACGTTCAAAACTTTCGATTTTTATATTTCG-CTAAAGACT-T-TA-AT---TTTCCATGGCCAGTTTTTCCTTTGGTA---CTGATCAGAAGTTAATTAGTGTGACTATTTTT---TT------GAGATAACATATCGCT-------GATTGCTT-TTGTTGATTACTGATT------------ACGGGTGATTACTTT-TCCAGGTCAACCTGCTGATAGA------C---ACAAC---GCAA---AGATGACGCAAAACACTAATTTGCTAGTG | |
| droBia1 | scf7180000298389:773089-773326 - | ACGATCTGAAAATCGTGACAATCATTGT-----------------------TGGAGGCTTTCGATTTGTTTATTTGG-C-----------------------CAAGGCCAGTTCTTCCTCCGACA---CCGATCGCAGCCCCATTAATGGAGTTGCTTTC---GG-------TGATAACATCTCTCTATTAGCAAATTATTGATTGCTGATTGCTGATA------------AGC--GAATTGCTTT-TCCAGGTCAACCTGCTGATAGA------G---ACAGCG-AGCCAGAAAGATGACGGAAGCCACTCGACTGCTGTTG | |
| droTak1 | scf7180000415769:847407-847660 + | ACAATATGAAAATCGTGACATTCGTT---------------------------AAAGCTTTCGATTTGTATATTTGG-CCAAAGACT-G-GG-GT---TTTCCATGGCTAGTTTTTCCTTGAACA---CAGATCAGAAGTTAATTAATGATGCTGCTTTT---AAGTGGAGCTGATAACATCTCTC-----GCTGATTTCTGATTGCTGATTACTGATA------------GCG--GAATTACTTT-TCCAGGTCAACCTGCTGATAGA------AGCCACAACGCCGCAA---AGATGACGCAAAGAACCCAACTGCTGATG | |
| droEle1 | scf7180000491356:101812-102043 + | ACAATATAAAAATCGTGATCGTCAATGTTT-TCAT---AAT-AAGCCAAGTTTA--------------------TGG-TAAAAGAATAGGGG-TT---TTTGCAAGGTCAGTTATTCATTCAACA---CTGATCAGAAGTTAATAAATGGAATTGTTTTG---TG------TGGTTACCATCTCA-----------------TTAGCTGATTACTGATT------------ACG--GAATT--TTT-TTCAGGTCAACCTGCTGATAGA------C---ACAAC---GAGA---TGATTACGCAAAAAACCCATCTGCTGATG | |
| droRho1 | scf7180000779467:528028-528275 - | ACAATGTAAAAATCGTGATATTCAAAGCTT--------ATTACTGCAAAGTTCATAGCTCAGAATTTGTATATTTTG-CTAAAGACT-G--------------AAGGCCA-TTGGTCTTAAAGCA---CTGATCAGAAGTCAATAAATGGAGCTGCTCTT---AG-------TGATAACATCTCGAT-------GTTTGGTGATTGCTGATTACTGATT------------ACG--GAATTGCTTT-TTCAGGTCGACCTGCTGATAGA------C---ACAAC---AAAA---AGATGACGCAAAGAACTCATCTGGTGCTG | |
| droFic1 | scf7180000453927:1306792-1307029 - | ACG-----AGAATCGTGACACTCATTGTTTTTTCT---AAT-TAGCGAAGTTTC---------ATTTGTATATTTGG-CCAAAGACT-G-GG-GG---TTTCCAAGGCCAGTTTTT-GTTTGGCA---TCGATCACAATTCTATTAGCAGGGCTGTTTTC---GA----------TA-----------------------TGGTTGCTGATTGTGG-------TCGAGATTACA--GAACTACTCT-TCCAGGTCAACCTGCTGATAGCCTGCT-G---ATATC---GCCA---AGATGATGCACAGAACCCAACTGCTGATG | |
| droKik1 | scf7180000299277:261813-261908 - | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACT-------GATTACCAATCACTGTTCACTGATT------------GCC--AACCATCTTAACAGAGGTCAGCCTGCTCTCGCA--------------C---GTAA---CGATGACGCG---CCCCCATCTCCTGATG | |
| droAna3 | scaffold_13117:1096513-1096586 - | CAAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGGCTTT-TCCAGGTATACCTACAGCTGAT---AAGA---GCA-C---ACGA---TGACGACAATGACAGCCCACCGCCTTCTG | |
| droBip1 | scf7180000396425:1019176-1019249 + | CAAAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGTTTT-TATAGGAATATCGACAGCTGAT---AAGA---ACC-C---AAGA---TGACGACAATAACAGCCCAACAGTTCCTG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
Generated: 05/19/2015 at 02:04 AM