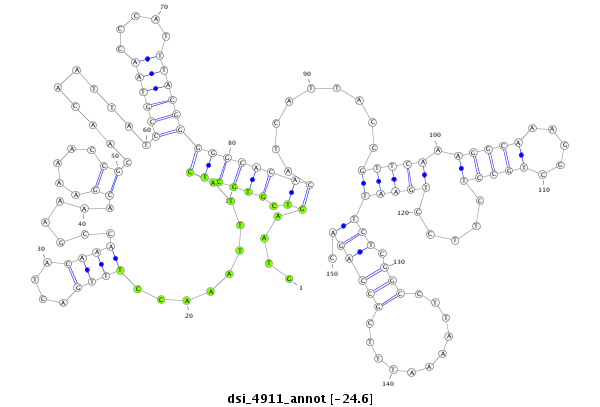
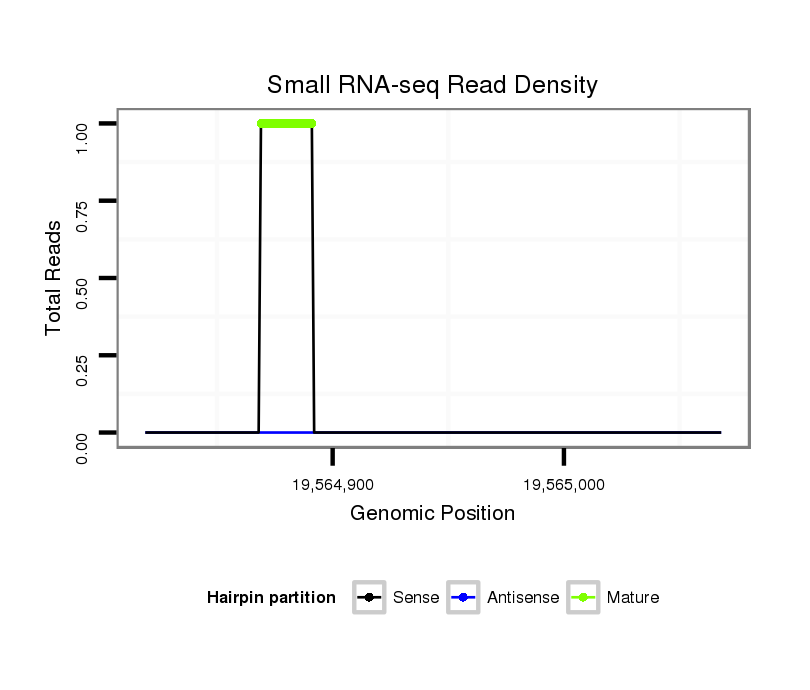
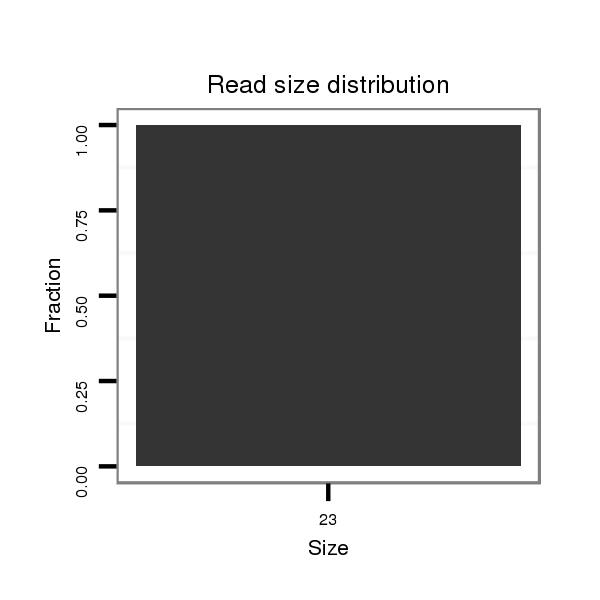
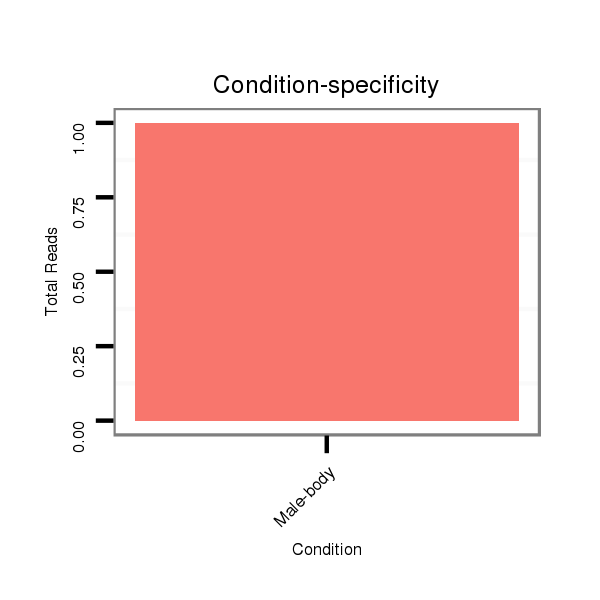
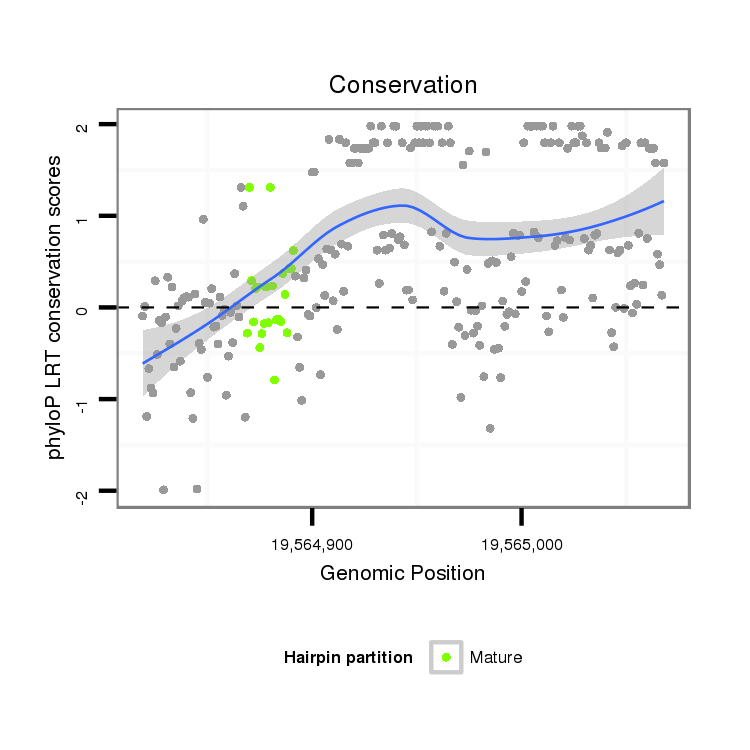
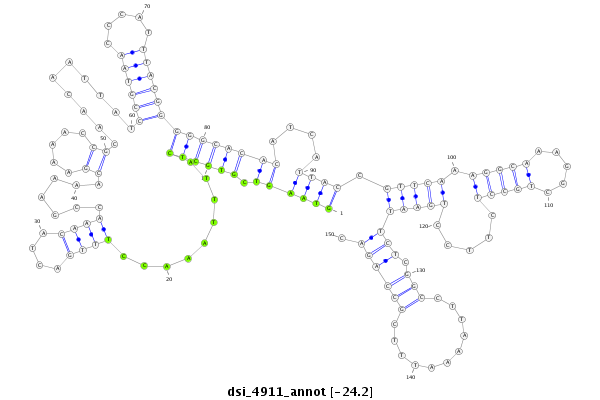
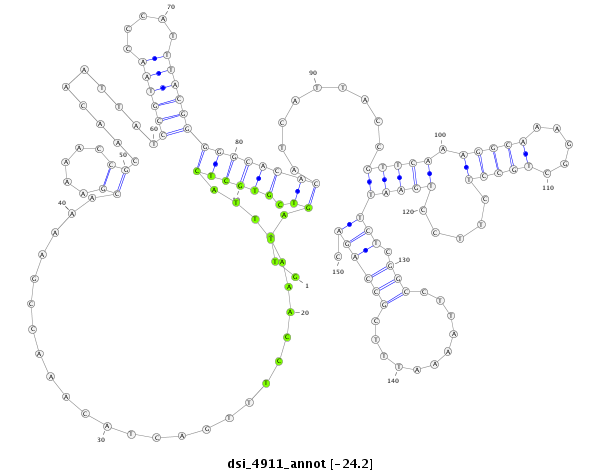
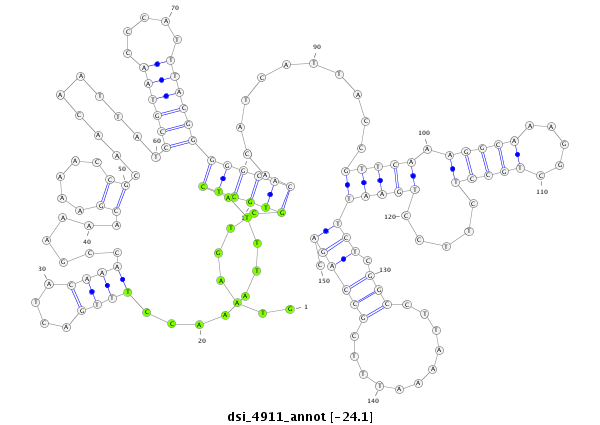
ID:dsi_4911 |
Coordinate:2r:19564869-19565018 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -24.2 | -24.2 | -24.1 |
 |
 |
 |
intergenic
No Repeatable elements found
|
AAGACGAGTCATTCAGCCCTAACTGTCCAAAGTTCAGGAGTCCTTTTTCGGTAAGTCGTGCTCATTTAAACCTTTGACTACAAACCGAAAACGAAAACCGCAACAATTATCCGTAACCCATTTACGGGGGCACACATCATTACCGTTCAAAGGCAAAGGCTGCCTCTTCCTGAATTCTCGGCCTTAAAATTTCGCCAGACCGAAATCGATTTGCCGGAGGCCTCTGCAGAGCCTTTATCACATAACTCAG
**************************************************....((.((((((.........((((....)))).......((.....))..........((((((.....)))))))))))))).........(((((.(((((.....))))).....)))))(((.(((...........)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M024 male body |
SRR553487 NRT_0-2 hours eggs |
GSM343915 embryo |
SRR902009 testis |
SRR553488 RT_0-2 hours eggs |
SRR553485 Chicharo_3 day-old ovaries |
SRR553486 Makindu_3 day-old ovaries |
O001 Testis |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................GTAAGTCGTGCTCATTTAAACCT................................................................................................................................................................................. | 23 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................AAGGCATAGGCTGTCTCT................................................................................... | 18 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................AAGTCGTGCTTCTGTAAACCT................................................................................................................................................................................. | 21 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................TTTTAACCTTTGCCTACAACC..................................................................................................................................................................... | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................CTTCTCGGCCTTAAAA............................................................. | 16 | 1 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...........................................TTTTTCGGTAAGACGGGC............................................................................................................................................................................................. | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................AATTGACGGGGGCACACA.................................................................................................................. | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| CGGACTAGTCATTCAGCC........................................................................................................................................................................................................................................ | 18 | 3 | 20 | 0.15 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................CGGTCCTAAGTTCAGGCG.................................................................................................................................................................................................................. | 18 | 3 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 |
| .........................................................................................................................................................................................AAAATTACGTCAGGCCGAAA............................................. | 20 | 3 | 7 | 0.14 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................CACCTGAATTCGCGGCCTT................................................................. | 19 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................AAAATTTCTCCAGACTGGAA............................................. | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................ATTGACGGGGGCTCAC................................................................................................................... | 16 | 2 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
|
TTCTGCTCAGTAAGTCGGGATTGACAGGTTTCAAGTCCTCAGGAAAAAGCCATTCAGCACGAGTAAATTTGGAAACTGATGTTTGGCTTTTGCTTTTGGCGTTGTTAATAGGCATTGGGTAAATGCCCCCGTGTGTAGTAATGGCAAGTTTCCGTTTCCGACGGAGAAGGACTTAAGAGCCGGAATTTTAAAGCGGTCTGGCTTTAGCTAAACGGCCTCCGGAGACGTCTCGGAAATAGTGTATTGAGTC
**************************************************....((.((((((.........((((....)))).......((.....))..........((((((.....)))))))))))))).........(((((.(((((.....))))).....)))))(((.(((...........)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR902009 testis |
M024 male body |
SRR553486 Makindu_3 day-old ovaries |
SRR618934 dsim w501 ovaries |
SRR902008 ovaries |
|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................TTTAGGCGTTGTTGATGGGCAT....................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................TTACTGTGGTCTGGCTTTAG........................................... | 20 | 3 | 13 | 0.46 | 6 | 3 | 0 | 0 | 0 | 3 |
| .............................................................................................................................................................................TAAGAGCCGGAAGTTTCAA.......................................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...........AAGGCGGGATTGACATGTT............................................................................................................................................................................................................................ | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 |
| .................................AGTCAACAGGAAAAAGCTATT.................................................................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................................................................................AAGTTGCAGTGTCCGACGGA..................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 | 0 |
| ............................................AACAGGCATTCAGCAGGAG........................................................................................................................................................................................... | 19 | 3 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | 2r:19564819-19565068 + | dsi_4911 | AAGACGAGTCATTC--------------------------------------------------------------------------AGCCCTAACTGTCCAAAGT--------------------T----C---AGGAGTCCTTTTTCGGTAAGTCGTGCTCATTT---AAACCTTTG-ACTACA-------AACCG-AAAACGAAAACCGCAACAATTATCCGTAA--CCCATTTACGGGG-GCACACATCATTACCGTTCAAAGG-------CAAAGGCT-GC-------CTCTTC----CT-------GAA------TTCTCGGCC-TTAAAATTTCGC-CAGACCGAAATCGATTTGCCGG-AGGCCTCTGC--AGAGCCTTTATCACATAACTCAG |
| droSec2 | scaffold_9:2335858-2336107 + | AAGACGAATCATTC--------------------------------------------------------------------------AGCCCTAACGATCCAAAGT--------------------T----C---AGGAGTCCTTTTTCGGTAAGTCGTGCTCATTT---AAACCTTTG-ACTACA-------AACCG-AAAACGAAAACCGCAACAATTATCCGTAA--CCCATTTACGGGG-GCACACATCTTTACCGTTCAAAGG-------CAAAGGCT-GC-------CTCTTC----CT-------GAA------TTCTCGGCC-CTAAAATTTCGC-CAGACCGAAATCGATTTGCCGG-AGGCCTCTGC--AGAGCCTTTATCACATAACTCAG | |
| dm3 | chr2R:19041415-19041668 + | AAGACGAATCTCTC--------------------------------------------------------------------------AACCCCAACGGATCAAAGT--------------------T----C---AGGAGGCCTTTTTCGGTAAGCCGTGCTCATTT---AAACCATCG-ACTACA-------AACCG-AAAACGAAAACCGCAACAATTATCCGTAA--CCCATTTACGGGG-GCACACATCATTACCGTTCAAAGG-------CAAAGGCT-GGCT---GTCTCTTC----CT-------GAA------TTCTCGGCC-TTAAGATTTCGC-CAGACCGAAATCGATTTGCCGG-AGGCCTCGGC--AGAGCCTTTATCACATAACTCTG | |
| droEre2 | scaffold_4845:20408446-20408694 + | AAA--------TTC-------------------------------------------------------------------------CAGCCTTAACGGTCCAAAGT--------------------T----C---AGAGGTC-CTTTTCGGTAAGCCGTGCTCATTT---AAGCC-CCGAACAGAAAACAGAAAGCCGAAAAACGAAAACCGCAACAATTATCCGTAA--CCCATTTACGGGG-GCACACATCATTACCGTTCAAAGG-------CAAAGGCT-GC-------CTGTTC----CG-------GAA------TTCTCGGGC-TTAAAATTTCGC-CAGGCCGAATTCGAT-TGCCGG-AGGCCTCGGC--AAAGCCTTTATCACATAACTCAG | |
| droYak3 | 2R:19004931-19005187 + | AATATGAATTTTCC--------------------------------------------------------------------------AGCCTTAACGGTCCCAAGT--------------------T----C---GGAAGTC-CTTTTCGGTAAGCCGTGCTCATTT---AAACCTCCG-ACAGAAAACCGAAAACCG-AAAACGAAAACCGCAACAATTATCCGTAA--CCCATTTACGGGGAGCACACATCATTACCGTTCAAAGG-------CAAAGGCC-GC-------CTCTTC----CG-------GAA------TTCTCGGGC-TTAAAATTTCGC-CAGGCCGAATTCGATTTGCCGG-AGGCCTCGAC--AAAGCCTTTATCACATAACTCAG | |
| droEug1 | scf7180000409672:7080488-7080746 + | AGGGATAATAATTT-------------------------------------------------------------------------AAGCCTTCAT-TTATAAACT--------------------T----TATAAAGAGTAACTTTTCCGTTCGCTGCGTTCCACC---AATTTTCCG-GCAACA------AAACTG-AAAACGAAAACCGCAACAATTATCCGTAA--CCCGTTTACGGTG-GCACACATCATTACCGTTCACCAC-------CAAAGAGCTCC-T---TCTTCTTC----TC-------AAA------GTCTCAACCTTTAAAATTTCGC-CAGACCGAAATCGATTTGCCGA-GGGCCTCGAC--AAAGCCTTTATCACATAACTCAG | |
| droBia1 | scf7180000301754:4372955-4373173 + | AGG----------------------------------------------------------------------------------------------------------------------------------------AGCAACTTTTCCGTAAGCTCTGCTC----TGCAGTTTTCCC-ACAAAAAACC---AGCAG-AAAACGAAAACCGCAACAATTATCCGTAA--CCCATTTACAGTG-GCACACATCATTACCGCTCACCGC-------CGAAAGTT-CC-------CTCATC----CT-------GGG------TTCCCAACCTTTAAAATTTCGC-CAGACCAAAATCGATTCGCCGA-AGGCCTCGAC--AAAGCCTTTGCCACATAACTCAG | |
| droTak1 | scf7180000415672:256478-256713 + | TGCTGTAA-CCTTT--------------------------------------------------------------------------CATTCTACTAATTTAAAGC--------------------TCTTCTATAAGGAGCAACTT---------------------------TTTCCG-GCAACA-------AACAG-AGAACGAAAACCGCAACAATTATCCGTAA--CCCATTTACGTTG-GCACACATCATTACCGTTCACCAC-------CAAAAGTT-CC-------CTCTTC----CT-------GGC------TGCTCAACCTTTAAAATTTCACCCAGACTGAAATCGATTTGCCGA-AGGCCTCGATCGAAAGCCTTTGTCGCATAACTCAG | |
| droEle1 | scf7180000490976:192634-192919 + | AAGGCATTACATT---TTTCTTTTCCA------------------------------------TTTATAAATAAACTACCATA-----------AAT-TTGT--------------AAACTTTAAACACCCTTATAGAGAACGAATTAT-AATAGTCCCCATTCGACC---CATTTTCCC-CAAGCA-------AACTG-AAAACGTAAGCCGCAACAATTATCCGTAA--CCCATTTACGGTG-GCACACATCATTAACATTCGCCAC-------CGAAATCT-CC-------CTCTTT----CT-------CAA------TTCTCAACCTTTAAAATTTCGC-CAGACCGAAATCGATTTGCCGA-AGGCCTCGAC--AAAGCCCTTATCACATAACTCAG | |
| droRho1 | scf7180000770245:21675-21838 + | A---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACGTAGGCAGCAACAATTATCCGTAA--CCCATTTACGATG-GCACACATCATTACCATTCACCAT-------CGAAATCT-GCC------TTCATC----CT-------TAA------TTCTCAACCTTTAAAATTTCGT-CAGACAGAAATCGATTTGCCGA-AGGCCTCGAC--AAAGCCTTTATCACATAACTCAG | |
| droFic1 | scf7180000454066:3087980-3088298 + | AAAGCAAAACTTTCATTTTCTATTCGATAAATGCCATAACAATCCATTATCACAAAAACCCAATTTAAAAACAAAATACCATCATTTAAGTCTTAAT-TTGCCAGCTTAAAAGGTTA-------------------------AACTT-----------------TTCTTCCAATTTTCTG-GCAACA-------AATCG-AAAACAAAAGCCGCAACAATTATCCGTAA--CCCATTTACGGTG-CCACACATCATTACCGTTCCCCAA-------CGAAATCT-GC-------CTCCTG----CTAAACTCTGAA------CTCTGAACCTTTAAAATTTCGT-CAGACCGAAATCGATTTGCCGA-AGGCCTCGAC--AAAGCCTTTATCACATAACTCAG | |
| droKik1 | scf7180000302366:688420-688600 - | AA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCCGCAACAATTATCCGTAA--CCCATTTACGGTG-GCACACATCATTACCATTCACCATTCACCGCCGGAATCA-CCCCGCT--TTCCTGGATCCAACCCTCTCAACCTCTTTCCTGAACCTTTAAAATTTCGT-CAGACCAAAATCGATTTGCCCA-AGGCCTCCT-------CTTCCATCACATAACTCTG | |
| droAna3 | scaffold_13266:11745516-11745706 - | AAGACTAT-CAGTT--------------------------------------------------------------------------AAC------------------------------------T----C---AAGTAACTTTGCTCCGTAAGC--TGCTTATTTTCCA--TTT-------------------CCG-GAATCGCAAGCCGCAACAATTATCCCCAAAGCCCATTTTCGGC--CCACACATCATTACCATTCT------------------------------------------------GGC-----TCCCTCAACCTTTAAAACTTCTC-CAGACCGGAATCGATT-GCCAAAAG-------------GCCTGTGTCGCGTAACCAAG | |
| droBip1 | scf7180000396547:725558-725733 + | C----------------------------------------------------------------------------------------------------------------------------------------------TTCACTCTCTAAGC--TGCTTATTTTCCA--TTT-------------------CCG-GAATAGAAAGCCGCAACAATTATCCGAAAA-GCTATTTTCGG-G-CCACACATCATTACCATTCT------------------------------------------------GGC-----TTCCTCAACCTTTAAAACTTCTC-CAGACCAGAATCGAT-TGCCAA-AGGCCTCCAC--AAATCCCCTGTCGCATAACCAAG | |
| dp5 | 3:3259969-3260125 - | C-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCTT-ACAACA-------AACTT-TAAATGTAATCC----------TCCGTAT--CCCATTGACGGTG-GCACACATCATTACCATTCCCTTTGCG----CAGACACT-CA-------GTCT------------------------TTGTGAATCTCTAAAATTTCAT-CAAACCGA-ATCGATTTGATGA-AGGCCTTTGG--TAACCCTTCGTCACA-------- | |
| droPer2 | scaffold_2:3452906-3453062 - | C-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCTT-ACAACA-------AACTT-TAAATGTAATCC----------TCCGTAT--CCCATTGACGGTG-GCACACATCATTACCATTCCCTTTGCG----CAGACACT------------CAGC----C----------------TTTGTGAATCTCTAAAATTTCAT-CAAACCGA-ATCGATTTGATGA-AGGCCTTTGG--TAACCCTTCGTCACA-------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
Generated: 05/18/2015 at 12:11 PM