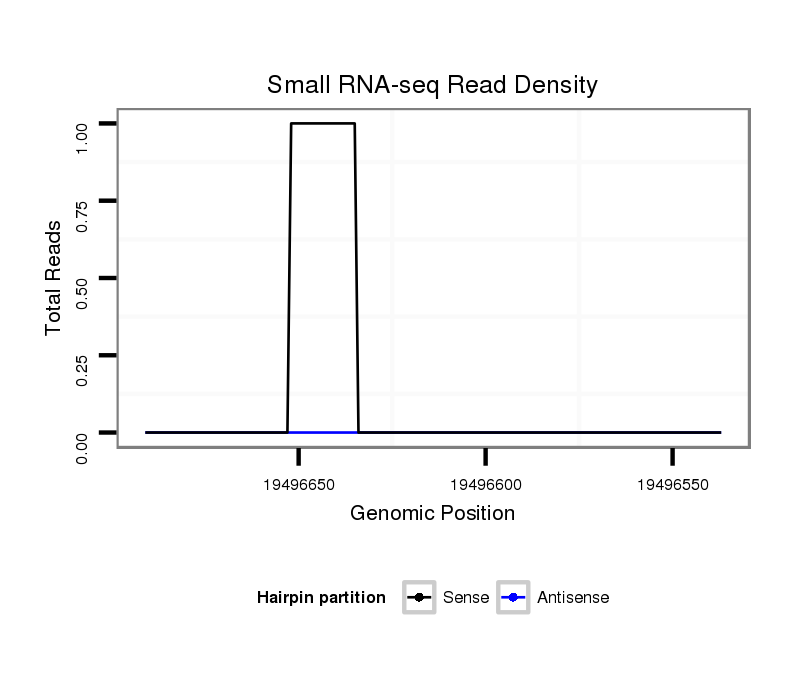
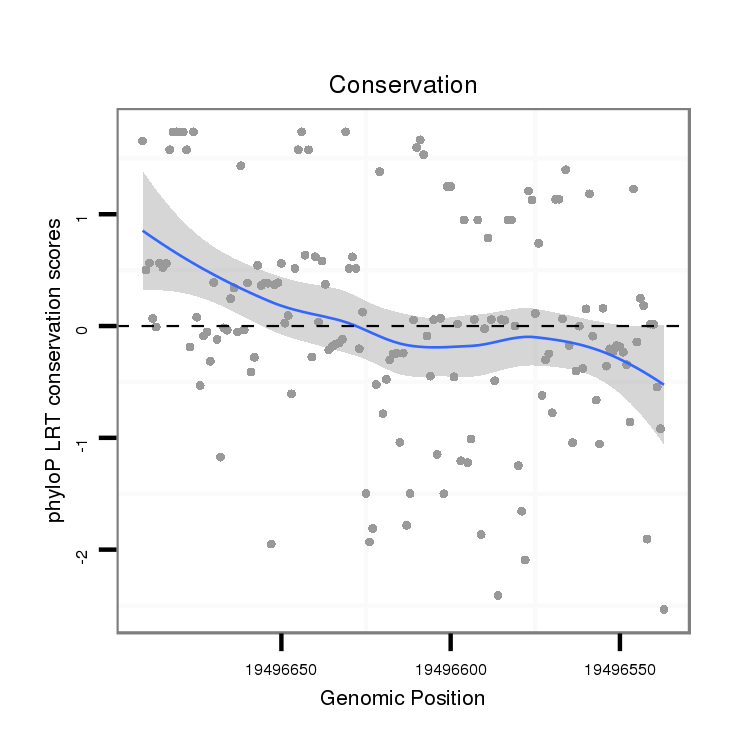
ID:dsi_4411 |
Coordinate:2r:19496587-19496641 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
intergenic
No Repeatable elements found
|
AATTGTTTGTTTACAAAAAACTGTTGGACAAACGTTTAGCATAAGTCAAGGTTAATTGGTTGACTTAGTCAGTCTTCTGTAAATAAACATATCGCACTTACGCAGGTTTTTTACAACTCCACAAATGGATTATTATACTCATCTTAATTGAACAC
**************************************************(((.(((((..((((.....))))..))...))).))).................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | O001 Testis |
SRR553485 Chicharo_3 day-old ovaries |
SRR902009 testis |
M023 head |
SRR553487 NRT_0-2 hours eggs |
SRR553486 Makindu_3 day-old ovaries |
SRR618934 dsim w501 ovaries |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................GTTGGTTGACTTGGTCACTCT................................................................................ | 21 | 3 | 3 | 1.33 | 4 | 0 | 3 | 0 | 0 | 1 | 0 | 0 |
| .......................................CATAAGTCAAGGTTAATT.................................................................................................. | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......TTGTTTACAATAGAGTGTTGGA............................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................AGTGAGTCTTCTCTAAAAAAA.................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................AAGGTTAAGTGGATGAGTTAG....................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................TACAACGCCTCAAATGGA.......................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................ACTGTTGGACAAATGT........................................................................................................................ | 16 | 1 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ........................................................TGGTTGACTTGGTCACTCTG............................................................................... | 20 | 3 | 13 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................TGAGTCTTCTGTAAAAA...................................................................... | 17 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................CTAAGTCATGGTTAAGTGG................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TTAACAAACAAATGTTTTTTGACAACCTGTTTGCAAATCGTATTCAGTTCCAATTAACCAACTGAATCAGTCAGAAGACATTTATTTGTATAGCGTGAATGCGTCCAAAAAATGTTGAGGTGTTTACCTAATAATATGAGTAGAATTAACTTGTG
**************************************************(((.(((((..((((.....))))..))...))).))).................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR553487 NRT_0-2 hours eggs |
SRR553486 Makindu_3 day-old ovaries |
SRR553488 RT_0-2 hours eggs |
SRR618934 dsim w501 ovaries |
SRR902009 testis |
M024 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................ATCAATCAGAAGTCATTTA....................................................................... | 19 | 2 | 3 | 1.33 | 4 | 4 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................TCAATCAGAAGGCATTTA....................................................................... | 18 | 2 | 9 | 0.33 | 3 | 1 | 2 | 0 | 0 | 0 | 0 |
| .................................................................................................................TTTGAGGTGTTTAGTTAATAAT.................... | 22 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................................TCAATCAGAAGTCATTTA....................................................................... | 18 | 2 | 10 | 0.20 | 2 | 1 | 0 | 1 | 0 | 0 | 0 |
| .................................................................ATCAATCAGAAGGCATT......................................................................... | 17 | 2 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................TCAGTCAGAAGGCATT......................................................................... | 16 | 1 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................TCAATCAGAAGGCATTT........................................................................ | 17 | 2 | 18 | 0.11 | 2 | 0 | 1 | 1 | 0 | 0 | 0 |
| .................................................................ATCAATCAGAAGTCATTT........................................................................ | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................................................AAAAAATGTTGAGGAGTGT.............................. | 19 | 2 | 11 | 0.09 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................................AAAAAAATGTTGAGGAGTGTA............................. | 21 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................................ATCAATCAGAAGGCAT.......................................................................... | 16 | 2 | 16 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..................................................................TCAATCAGAAGGCATTTAG...................................................................... | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................AAAAATGTTGAGGTTTAT.............................. | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................CAACTAGTTTGCCAATCGT.................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | 2r:19496537-19496691 - | dsi_4411 | AATT---GT---------------TTGTTTACAAAAAACTGTTGGACAAACGTTTA------------------------------------GCATAAGTCAAGGTTAATTGGTTGACTTAGTCAG------------T-C------------------------TTCTGTAAATAAACATATCGCA-CTTACGCAGGTTTTTTACA--A------------------------CTCCACAAATGG-------------------ATT---------------------------------------ATTATACTCATCTTAATTG-----AACAC |
| droSec2 | scaffold_9:2268101-2268254 - | AATT---GT---------------TTGTTTACAAAAAACTGTTGGACAAACGTTTA------------------------------------GCATAAGTCAAGGTTAATTGGATGACTTAGTCAT------------T-C------------------------TTCTGTAAATAAACATATCGCA-CTTATGCAGGTTTT-TACA--A------------------------CTCCACAAATGG-------------------ATT---------------------------------------ATTATACTCATCTTAATTG-----AACAC | |
| dm3 | chr2R:18974465-18974618 - | AATT---GT---------------TTGTTTACAAAAAACTGTTGGACAAACGTTTA------------------------------------TCATAAGTCAGGGTTAAATTGTTGACTTAGTCAT------------T-C------------------------TTCTGTAAATAAACATATCGCA-TTTACACAGGTTTT-TGCA--A------------------------CTCCACAAATGG-------------------ATT---------------------------------------ATTTTACTCATCTTAATTA-----AACAC | |
| droEre2 | scaffold_4845:20338643-20338783 - | AATT---GT---------------TTGTTTACTAAAAACTGATGGACAAACGTTTA------------------------------------TCATAAGCCAAGGTTAAATTATTGACTTAGTCAT------------G-C------------------------TTCTGTAAATAAACGCATCGCA-CTTACGCAGCTTTT-CG------------------------------------------------------------------------------------------------------ATTAAACTCATCACAAATATAAAAAGCAC | |
| droYak3 | 2R:18937786-18937936 - | AATT---GT---------------TTGTTTACAAAAAACTGTTGGACAGATGTTTA------------------------------------GCATAAGCCAAGCTTAAATTAATGACTTTGTCAT------------T-C------------------------TTCTGCAAATAAACATATCGCA-CTTACGCAGATTTT-TGTA--A------------------------CTCTATAAATGG-------------------ATT------------------------------------------AAACCCATCACAATTG-----TACAT | |
| droEug1 | scf7180000409672:6990908-6990988 - | AATT---GT---------------TTGTTTACAAAA-ATT--ATCATAAACATGTA------------------------------------CTATAAACCAAGCC-AATAGATTATTTCAAGCAG------------T-T------------------------GTTCAAAAA----------------------------------------------------------------------------------------------------------------------------------------------------------------TA | |
| droBia1 | scf7180000301754:4284558-4284729 - | -------------------------TGTTTACGAA--TCTTTATGACAAACATTTA------------------------------------TTATAAACCAAGGTCAGGTTGTTGACTTGGTAAAATATTAC-AAA--GCTTTATAGAACAGCATTTAGTATTAGTCAGGAAATGAAAAAATAAAA-TAAATGCTGTATTT-ATTAT-A------------------------CTTTTAAAAT-G-------------------TTT------------------------------------------ACCTACGGCATAATT---------AT | |
| droTak1 | scf7180000415672:156272-156432 - | AACAAAAGA---------------TTGTTTACAAAA-ACCTCATGACAAACATTTA------------------------------------CTATAATCCAAGGTCAGGTTATTGACTTGACCAAG----A------C-C--------------------------TCTTGAATATTTAAATTGTA-ATTAA--AACTTTT-AC-A-----------------------------TCAA---TAC-------------------ATG-------------ATTTAACAAATCGTCATAAAGGGAATATAT----------AAT-------AAAAT | |
| droEle1 | scf7180000490976:95559-95740 - | AATC---AT---------------GTGTTTACAAA--ATTTTATGACAAACATTTA------------------------------------TTTCCAACCAAGATTAGGTTATTGACATAGCCAGA-----------T-C------------------------TATT----ATAAAAATATCGAA-AGTACGCAGGTTTT-CTGA-AAAACAGTGAACAACATGTTTAACAACTACAAAA------------------CACGGGTTATTTCAATTGATA----------------------------TATATTTATTTTATTA---------GA | |
| droRho1 | scf7180000776932:39575-39692 - | AATC---AT---------------GTGTTTACAAAT--TTCGATGACAAAGATTTA------------------------------------TTATCGTCCAAGGTCAGGTTATTGACATCAACAGC-----------T-C------------------------TAGTAAAAATATATACATCGCAAAATATGCATGTATT-CTCA--A------------------------T---------------------------------------------------------------------------------------------------TA | |
| droFic1 | scf7180000454066:3008632-3008767 - | AATT---GT---------------TTGTTTACAAC--ATTTTATGACAAACTTTTG------------------------------------TTATAAACCAAGGTCAGGTCGATGACCTATCT--------------C-C------------------------TCCTTAAAATA-------------------------------------------------------------------TGTATAATTACCCAAAACAAAAGAAAAATTCATTTGTA----------------------------TTTA-TTATTTAAATT---------GC | |
| droKik1 | scf7180000302366:773866-774003 + | AATT-GTGT---------------TTGTTTACAAAGGTTTTCTAGCTACGGATTCAGCGTAGAGTCCCAGCAGAGAAATTTCCTGATCAGTTCTATAAACCAAGGTCAGGTTATTGACACT------------------------------------------------CAAAATT-------------------------------------------------------------------TAT-------------------AAT---------------------------------------GTTACACT----TAAGTTT-----AATTC | |
| droAna3 | scaffold_13266:11815062-11815147 + | AATT---GT---------------TTGTTTACGAGC--C----------------------------------------------------------CTCCAAGGTCGGGCTATTGACTTTCCGAGA----GC-CAA--CTTC------------------------TT----------------------------------TGGA--A------------------------C--CTCAAGTGC-------------------ATA---------------------------------------ATC------GCC---------------TT | |
| droBip1 | scf7180000396547:657640-657738 - | ATTT---ATTGAGGAAACCATTTTTGGTTTACAAA---------------CCC------------------------------------------TCATCCAAGGTCGGGGTATTGACTTAAA--------GCTCAAATCATC------------------------TTTGGAAT------AT--------------------------------------------------------CAAGTGC-------------------ATA---------------------------------------AT-------------------------TG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
Generated: 05/18/2015 at 11:57 AM