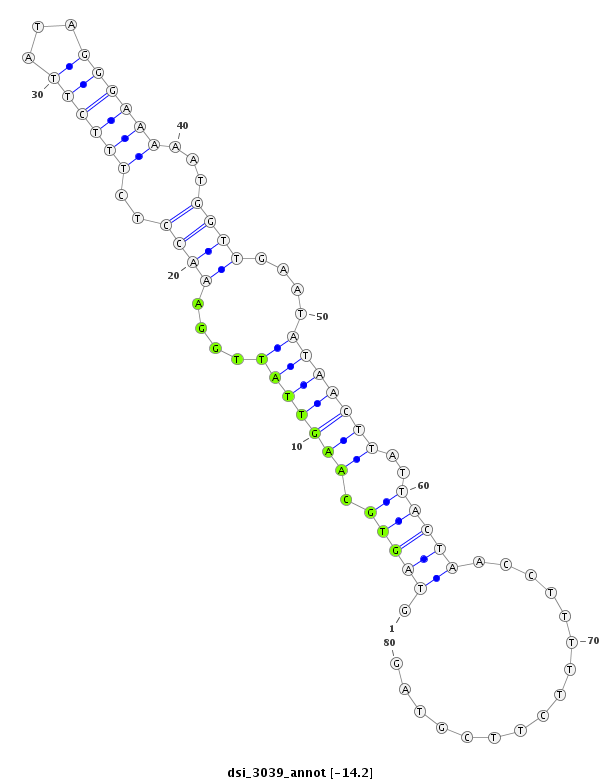




ID:dsi_3039 |
Coordinate:3r:5617368-5617447 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
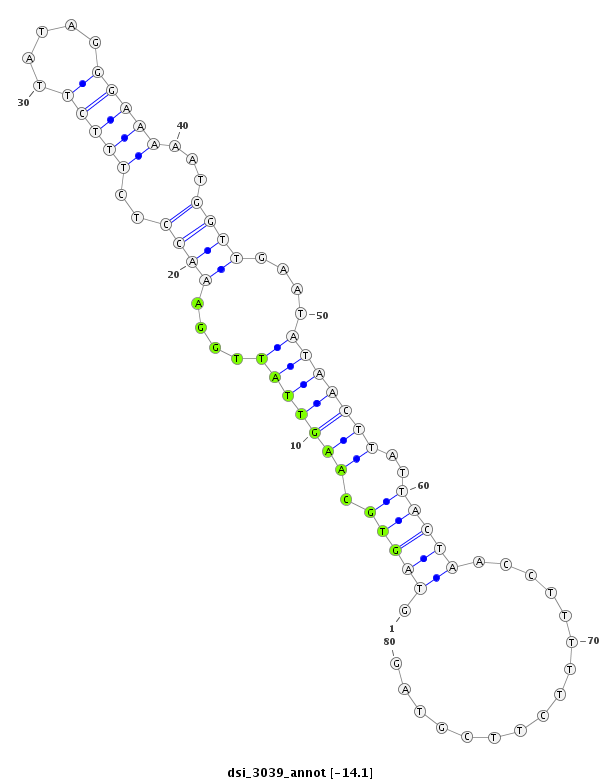
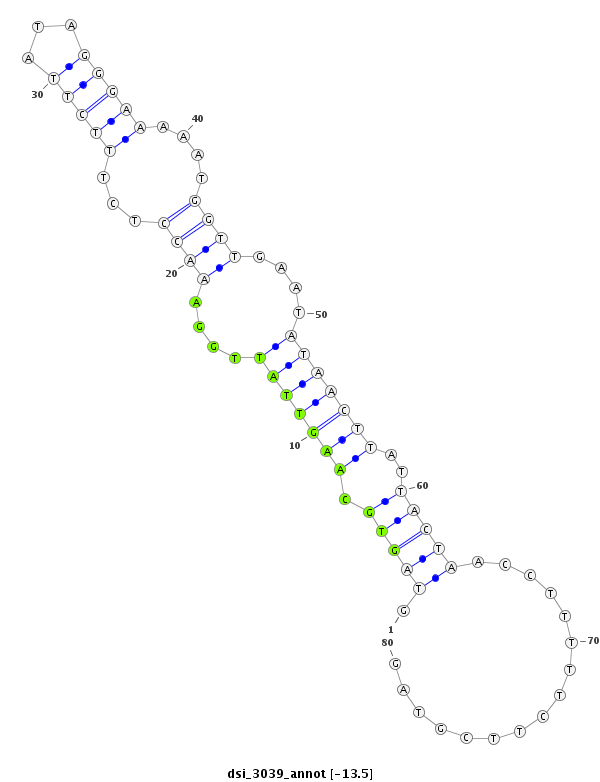
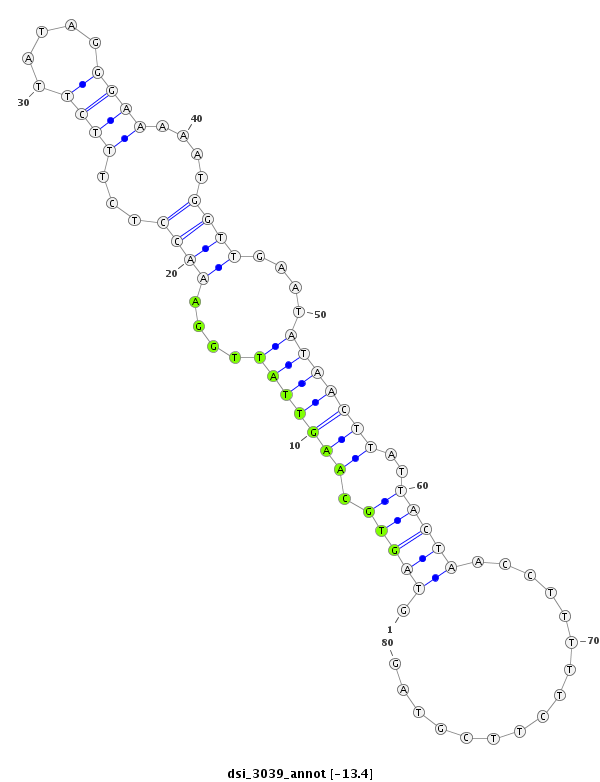
| -14.1 | -13.5 | -13.4 |
 |
 |
 |
five_prime_UTR [3r_5617202_5617350_-]; exon [3r_5617128_5617350_-]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------################################# CATTTTCATTCTAATTCTGGCGATTTTGTACTTTAATTTATTTTTTGCAGGTAGTGCAAGTTATTGGAAACCTCTTTCTTATAGGGAAAAATGGTTGAATATAACTTATTACTAACCTTTTTCTTCGTAGCATATCTGTAATTCTGGGCAGGCGTTGACCCCTGACATTTCTCAAAATGG **************************************************.(((((.(((((((....((((..((((((...))))))...))))....)))))))..)))))................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR553485 Chicharo_3 day-old ovaries |
SRR618934 dsim w501 ovaries |
SRR553486 Makindu_3 day-old ovaries |
SRR902009 testis |
GSM343915 embryo |
SRR553488 RT_0-2 hours eggs |
SRR553487 NRT_0-2 hours eggs |
M024 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................GTGCAAGTTATTGGA................................................................................................................ | 15 | 0 | 1 | 9.00 | 9 | 8 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................ACTAACCTGTTTGTTCGTAGCA................................................ | 22 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................AGTGCAAGTTATTGGATACA............................................................................................................ | 20 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................GTGCAAGTTATTGGGAACC............................................................................................................ | 19 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................TGCAAGTTATTGGAAC.............................................................................................................. | 16 | 1 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................TCTATCTTATAGGGAA............................................................................................ | 16 | 1 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................TTCTTAGAGGGACAAGTGG...................................................................................... | 19 | 3 | 10 | 0.40 | 4 | 0 | 0 | 0 | 0 | 0 | 2 | 2 | 0 |
| .......................................................GCAACTTATCGGAAACCCCT......................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................GTGCAAGTTATTGGTA............................................................................................................... | 16 | 1 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....TTCATTCGAATTCTCGCG.............................................................................................................................................................. | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................................................................TAGGCAAAAACGGTTGACTA............................................................................... | 20 | 3 | 9 | 0.22 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................AACTCTAGGCAGGCGTAGA...................... | 19 | 3 | 14 | 0.14 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............TTCTGGCGACTTTTTACT.................................................................................................................................................... | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................GGTGCAAGTTATTGG................................................................................................................. | 15 | 1 | 11 | 0.09 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................TTGTAAACCTCTTTC...................................................................................................... | 15 | 1 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........................................................................................TGGTTGAATATAACTGT........................................................................ | 17 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................TTTTTGAAGCTGGTGCAAGT....................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................TCAGGGCAGGCGGTGA...................... | 16 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
|
GTAAAAGTAAGATTAAGACCGCTAAAACATGAAATTAAATAAAAAACGTCCATCACGTTCAATAACCTTTGGAGAAAGAATATCCCTTTTTACCAACTTATATTGAATAATGATTGGAAAAAGAAGCATCGTATAGACATTAAGACCCGTCCGCAACTGGGGACTGTAAAGAGTTTTACC
**************************************************.(((((.(((((((....((((..((((((...))))))...))))....)))))))..)))))................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M024 male body |
GSM343915 embryo |
SRR553485 Chicharo_3 day-old ovaries |
|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................................CCCGTCCGCAATCGGGGA................. | 18 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 |
| .............................................................................................................................................................TGGGGACTATAAAGAGCTA.... | 19 | 3 | 13 | 0.08 | 1 | 0 | 1 | 0 |
| ............................................................................GGAATATACCTTTTTAC....................................................................................... | 17 | 2 | 20 | 0.05 | 1 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | 3r:5617318-5617497 - | dsi_3039 | CAT-------------------------TTTCATT------------CTAATTCTGGCGATTTTGTACTTTAATTTATTTTTTGCAGGTAGTGCAAG-----------TTATTGGAAACCTCTTTCT--TATAGGGAAAAATGGTTGAATATAACTTATTACT----AACC-----------------------------------------------------TTTTTCTTCGTAGCATATCTGTAATTCTGGGCAGGCGTTGACCC-----CTGACATTTCTC------AAAATGG |
| droSec2 | scaffold_25:110758-110935 - | AAA-------------------------TTTCTAT------------CTAATTCTGGCCTTTTTGTACTATAATTTATTTTTTGCAGGTAGTGCAAG-----------TTATTGGAAAGCTCTTTCT--TATAGGGAAAAATGATTGAATATAAATTATTACC----AACC-------------------------------------------------------TTTCTTTGTAGCATATTTGTAATTCTTGGCAGGCGTTGACAC-----CTGACAGTTCTC------AAAATGG | |
| dm3 | chr3R:15666439-15666599 + | T------------------------------CTAT------------CTAATTCTTTAGATTTTGTACTACCATAC-----CGGCAGGTAGTGAAAA-----------ATA-------CTTCTTTCT--AATGGGGAAAAATGTTCGAATAGAACTTATTACC----AACC-------------------------------------------------------TTTCTTTGTAGCAGATCTGTAATTCTGGGCAGGCGTTGACAG-----CTGACAGTTCTC------AAAATGG | |
| droEre2 | scaffold_4770:5996642-5996803 - | AGT-------------------------TTTCCAT------------GCAACACTGACGATTTGGTATTATCATTTATTTATGACC-GAAGTGGCGTTAATCCTGGGGTT-TTGAAAACCGCCTAGT--A-------AA------------------------AAAC----ATTTCAG-------------------------------------------------TTCTTGGCAGCAGATGTATAATTCTGGGCAGGCGTAGACAC-----TTGACAGTTCCC------AAAATGG | |
| droYak3 | 3R:4436066-4436278 + | TGT-------------------------G-TCTAT------------CCAA---TGGCGATTTTGTAATATCATTTATTTTCGCCT-GTAATGGCAATAATCTTAGTGTC-TTGGAAACCTCC--------TAGGGAAA------------------------AATC----ATTTCAGATTGAAATGGTTGAGAGAAACTCATTACCAGATGACCAACAAACATGTTTTTTCTGTAGCAAATGTATAATTCTAGGGCGGCGTTGACTCTTTTGTTGACAGTCCTC------AAAATGG | |
| droEug1 | scf7180000409804:1971560-1971780 + | AAT-------------------------TTTCTAT------------CCAATTCTGATGCTTCTATACTACCAAATCATTTTGCCA-ATGATGGCAATAGTCTCGGGGAT-ATGGAGAGCTCCTTAC-----AGCCAGA------------------------ATAG----AAATCAAGATGACATGGCCAATTTGAGCCCCCAACCAGTTGAGCCAAGAAAATGCCCTTGGTGTGGAACTTCTATAGTTCTTAGCAAGCCTTGACAC-----TTGACAGTTCTCAAAACAAAAATGG | |
| droBia1 | scf7180000302402:1439555-1439798 + | TGTGCCTTCTGTGCCTGTCACTGCCGATTTTCTAT------------CCCATCCTGGTGTTTTTCTACTACAATTTCTATCTACCC-GCAATGGCAATCGCATTTCAGCT-GTGGAGAACCGCTTAT--CATGGGAGTA------------------------ACAC----ATTGCAGGACAGAATAGCCGAAGAAAACTGAAAACCAGTTGACCAAAGAAAATGTCCCCTTTGTGGCAGATCTTTAACTCTGGGCAGGCGTTGACAC-----TTGACAGTTTTT-----AAAAATGG | |
| droTak1 | scf7180000415710:150707-150938 - | CCT-------------GTCACTGCCGATTTTCTAT------------CCAATCCTGGTGTTTTTCTATTATCATTTCTTCCTGCCC-GTCATGGCAATCGTATCGCAGTT-GTGTAGAACTCCTTAT--TATAGAGAGA------------------------ATAC----ATTTCAGGACGGAATAAGCGAAGAAAACTCAAAACCAGTTGACCAAAGGAATTGTCCCCTGTGTGGCAGATCTATAATTCTTGGCAGGCGTTGACAC-----TTGTCAGTTCTAA----AAAAATGG | |
| droEle1 | scf7180000491008:1243323-1243389 - | AAT-------------------------TTC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCCTTTATAGCAAAAAAATATTTCTGGGCAGGCTTTG-CAC-----TTGACAGTTAAA-----AAACATGG | |
| droRho1 | scf7180000776775:7721-7902 - | T---------------------------TTTCTAT------------CCAATTCTGGTAT-TTTCTACT-------------GCCT-GTTATAACAATTATTTCGCAGCT-CTGGATAAGTCCTTACTACCCAGGGAA-------------------------------------------------TTCGAAACCAATCTAAATCTAGTTAACCGAAAGAACTACCCTTTTTGTGGCAGATCTATAATTCTGGGCAGGCGTTT-CAC-----TTGACAGTTTTA------AACATGT | |
| droFic1 | scf7180000454126:689125-689301 + | CCT-------------------------AATACTA---T--------GAAACCATGACAATTATTTGCTTTCGTCTTTGATTTCCC-GTATT-ACAATCAGCTCAT-------------------------------------ATTAATTATGTATTATTATT----TAC-ATTTCCGGACGAGTTTGCTGTCAAAA------------------------------TTTTTAAAAAGTGATCTATATTTCCGGGTAGGCCTTGTCAA-----CTGACAGTGTTA------AACATGG | |
| droKik1 | scf7180000297926:155753-155848 - | GAC-------------------------ATTTTGTTCCTGAAACCATT--TCCCTGGC-------------------------------------------------------------------------------------------------------------ATTC-------------------------------------------------------TTAGCCTGTGTCAAATCTCTAGTCCTGAGTAGGCTTTGCCAC-----CTGACAGTTATG------GAAATGA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
Generated: 05/18/2015 at 05:38 PM