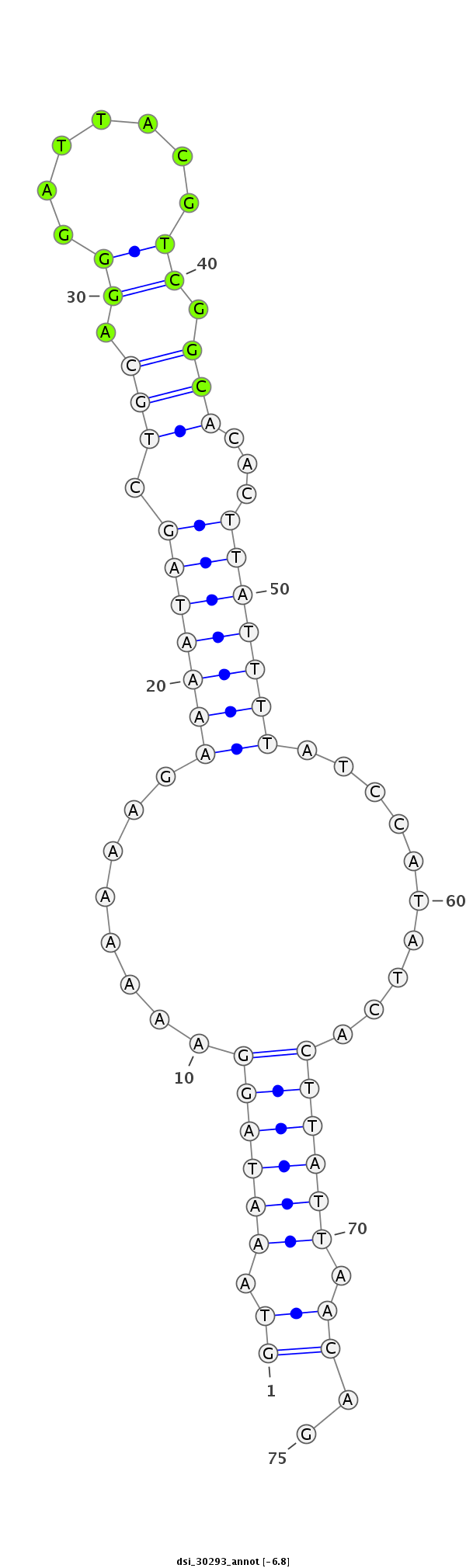
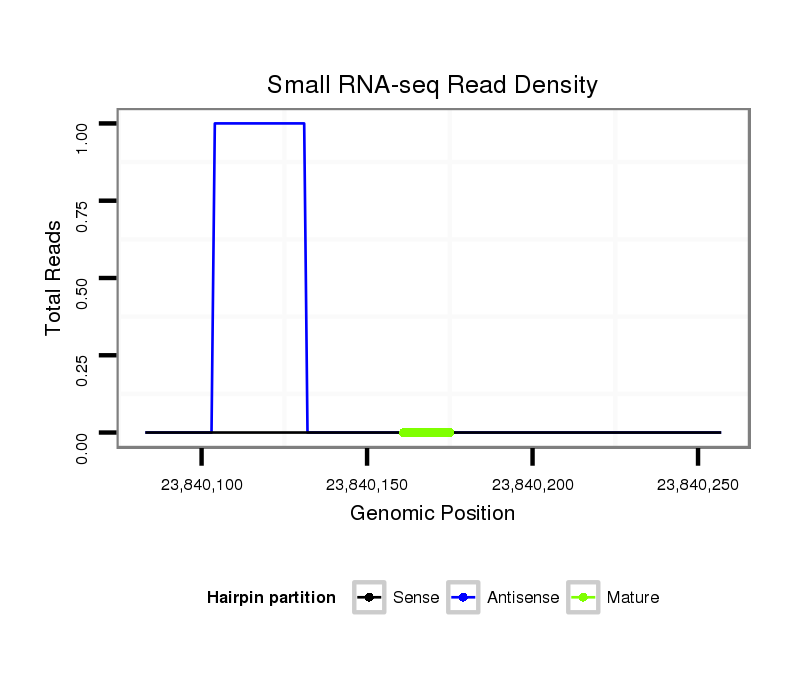

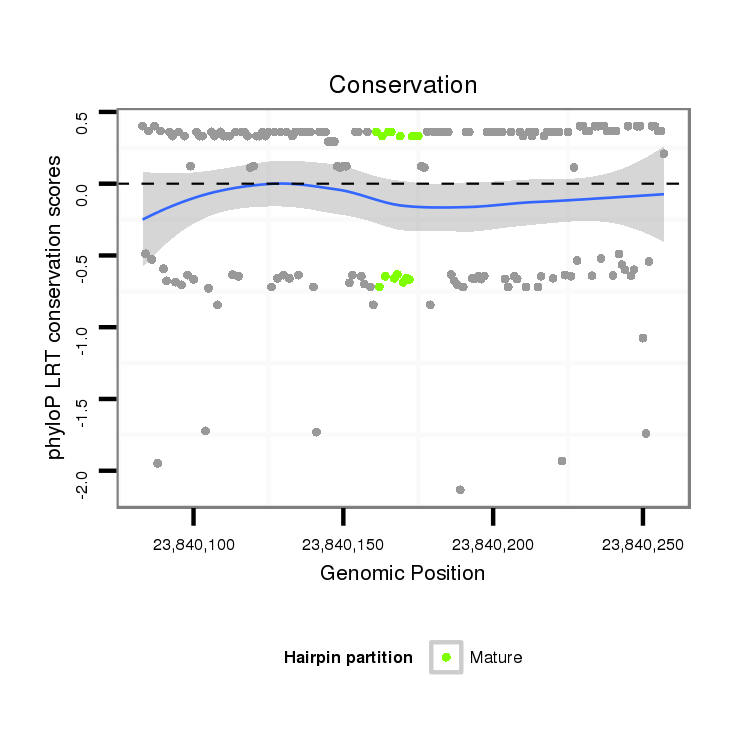
ID:dsi_30293 |
Coordinate:3r:23840133-23840207 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
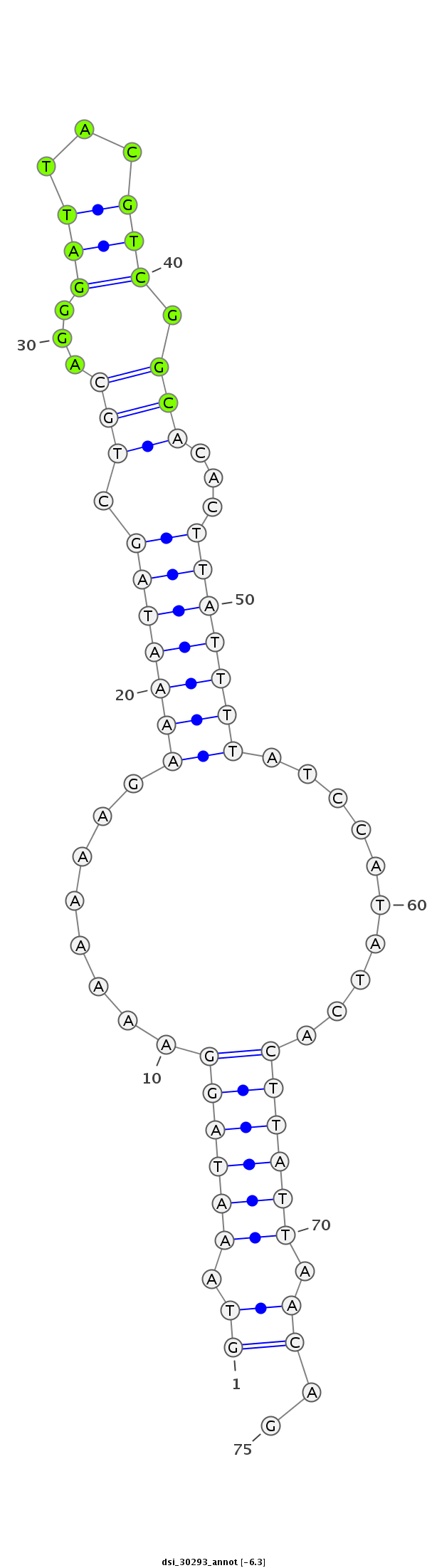
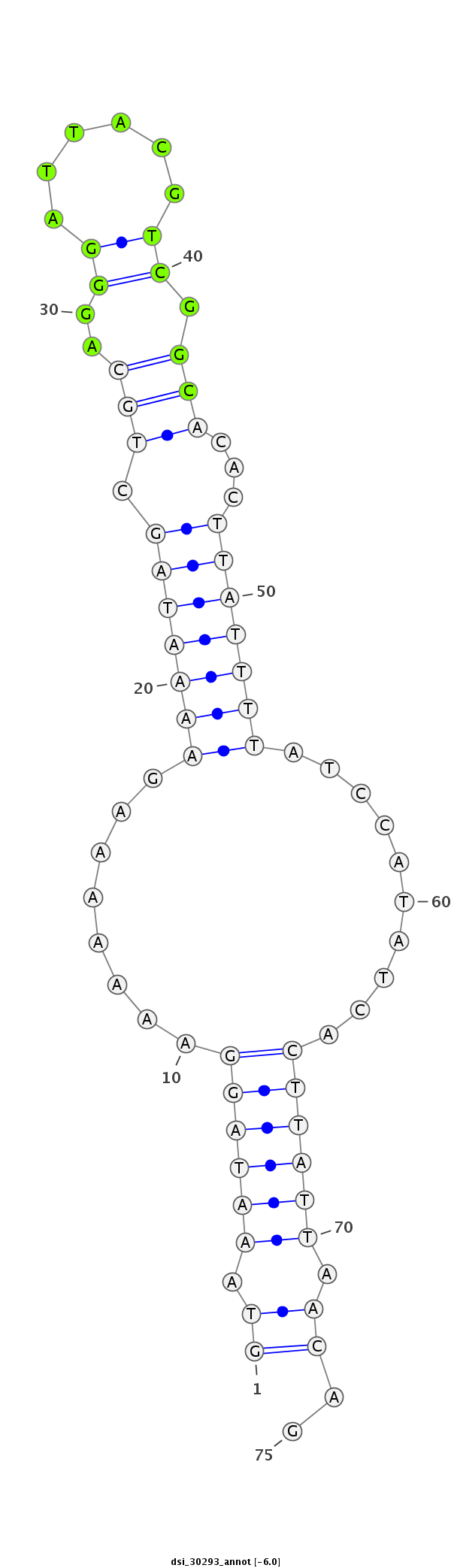
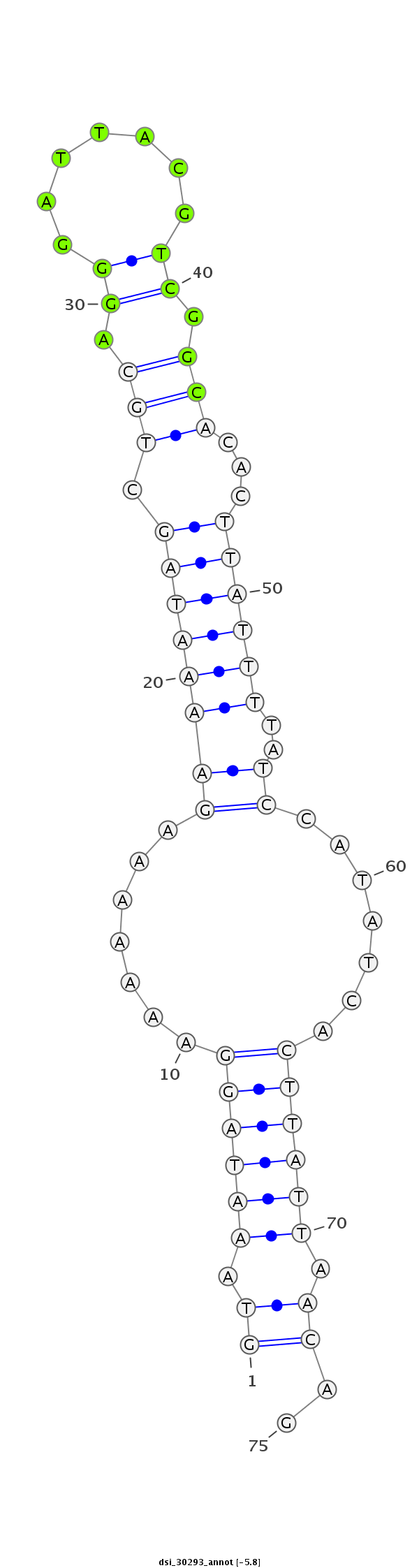
| -6.3 | -6.0 | -5.8 |
 |
 |
 |
intergenic
No Repeatable elements found
| mature | star |
|
ATCCAGGCATCATAGAAGTGCAGTCGAGCCTACATCCTCCAGTGATAAAAGTAAATAGGAAAAAAAGAAAATAGCTGCAGGGATTACGTCGGCACACTTATTTTATCCATATCACTTATTAACAGAAGGTCACGGTTTAACTACCATACGAAATTGGCGTGGAACTTGGTTAGCA
**************************************************((.((((((........(((((((.(((.((.......)).)))...)))))))..........)))))).))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR553488 RT_0-2 hours eggs |
SRR553486 Makindu_3 day-old ovaries |
SRR618934 dsim w501 ovaries |
SRR553485 Chicharo_3 day-old ovaries |
|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................TTAACAGGAGGTCTAGGTTTAA................................... | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ..............................................................................AGGGATTACGTCGGC.................................................................................. | 15 | 0 | 4 | 0.50 | 2 | 0 | 0 | 2 | 0 |
| .............................................TAAAAGTAACTGGGAAAAAAAAAAAA........................................................................................................ | 26 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 |
| ........................................................................................................................AACAGAAGGTCAGGG........................................ | 15 | 1 | 9 | 0.22 | 2 | 0 | 2 | 0 | 0 |
| ...................................................................AAAATAGCTGAAGGGA............................................................................................ | 16 | 1 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 |
| .....................................................TATAGGAAAAAAAGAAA......................................................................................................... | 17 | 1 | 9 | 0.11 | 1 | 1 | 0 | 0 | 0 |
| ..................................................................................ATTAAGTCGGCACAC.............................................................................. | 15 | 1 | 11 | 0.09 | 1 | 1 | 0 | 0 | 0 |
| ........................................AGTGATAAAAGAAAAT....................................................................................................................... | 16 | 1 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 |
|
TAGGTCCGTAGTATCTTCACGTCAGCTCGGATGTAGGAGGTCACTATTTTCATTTATCCTTTTTTTCTTTTATCGACGTCCCTAATGCAGCCGTGTGAATAAAATAGGTATAGTGAATAATTGTCTTCCAGTGCCAAATTGATGGTATGCTTTAACCGCACCTTGAACCAATCGT
**************************************************((.((((((........(((((((.(((.((.......)).)))...)))))))..........)))))).))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M024 male body |
M023 head |
SRR902009 testis |
GSM343915 embryo |
M053 female body |
SRR553487 NRT_0-2 hours eggs |
SRR553486 Makindu_3 day-old ovaries |
SRR618934 dsim w501 ovaries |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................CCTCAGCTTGGATGTTGGA......................................................................................................................................... | 19 | 3 | 20 | 11.20 | 224 | 192 | 31 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................CTCAGCTTGGATGTTGGA......................................................................................................................................... | 18 | 3 | 20 | 3.25 | 65 | 52 | 12 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................TCAGCTTGGATGTTGGAGG....................................................................................................................................... | 19 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................TCCTCAGCTCGGATGTTGGA......................................................................................................................................... | 20 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................CCTCAGCTTGGATGTTGGAG........................................................................................................................................ | 20 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....CCGCAGTATCTTCACGTCAGCTC................................................................................................................................................... | 23 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................TCAGCTCGGATGTAGGAAGA...................................................................................................................................... | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................TCAGCTCGGATGTAGGAGGTCACTATTT.............................................................................................................................. | 28 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................CGTCCCTAGTGCAGCCCTCT............................................................................... | 20 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................CCTCAGCTCGGATGTTGGAA........................................................................................................................................ | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................CGTCAGGTCGTATGTAGGGG........................................................................................................................................ | 20 | 3 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................AGAGCCAAAGTTATGGTATG.......................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................TAGGAGGTCTCTATT............................................................................................................................... | 15 | 1 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................CCTCAGCTAGGATGTTGGA......................................................................................................................................... | 19 | 3 | 9 | 0.11 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................TAGATGGTATGCCTTAA.................... | 17 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...................CCTCAGCTTGGATGTGGGA......................................................................................................................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................CCTCAGCTTGGATGTTGG.......................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | 3r:23840083-23840257 + | dsi_30293 | ATCCAGGCATCATAGAAGTGCAGTCGAGCCTACATCCTCCAGTGATAAAAGTAAATAGGAAAAAA------------------------------------------------------------------------------AGAAAATAGCT--------GCAGGGATTACGTCGGCACACTTATTTTATCCATATCACTTATTA-------------A-CAGAAGGTCACGGTTTAACTACCATACGAAATTGGCGTGGAACTTGGTTAGCA |
| droSec2 | scaffold_4:3343123-3343298 + | ATCCAGGCGTCATAGAAGTGCAGTCGAGCCTACATCCTCCAGTGATAAAAGTAAATAGGAAAAAA-----------------------------------------------------------------------------TAGAAAATAGGT--------GCAGGGATTACGTCGGCACACTTATTTTGTACATATCTCTTATTA-------------A-CAGAAGGTCACGGTTTAACTACCATACGAAATTGGCGTGGAACTTGCTTAGCA | |
| dm3 | chrUextra:97112-97149 - | AACTACGG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATACGGAATTGGAGCGGAGCTTACTTAGCA | |
| droEre2 | scaffold_4725:1655-1916 - | ATCCAAGCGTCTTCGG-GTGCTGTCCAGCCAATATC--CCAGTTATAGAAGTGAATATAAAAAAAGTTTCTATAAGTAGCCGGGGAGGCGAGAAGAGGAAATCACCAAGGTGGTAAAACCTAAGTCACCGCTCAGCCCTAGAT----GGTAAGTTTTTGGGATGATGAATTTCATCGGC--AGTTATTTAGGTAATATTTTTTATTAGGCTCGGCGATTGATAAAAAGTTCAAAGTTTAATCAT-TTACGGAATTGGAGTATAGATT--CTAGC- | |
| droYak3 | v2_chr2L_random_030:54805-55006 + | ATCCAGGCGTCATCGG-ATGCGTTCCAGCCAATATC--CCAGTTAAAGATGTGAATATTAAA---GTCTCTATATGTTG--------------------------------------------------------CCCTAGAT----AGTAAGTT-TTGGGATGATGAATATCGATGGC--AGTTATTTAGGTAATTATTTTTATTATACTCGGCGATTGGTAAAGAGTTCAAAGTTAAAGCAT-TTACGGAAATGGAGTGGAGCTT--CTAGC- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/19/2015 at 12:52 AM