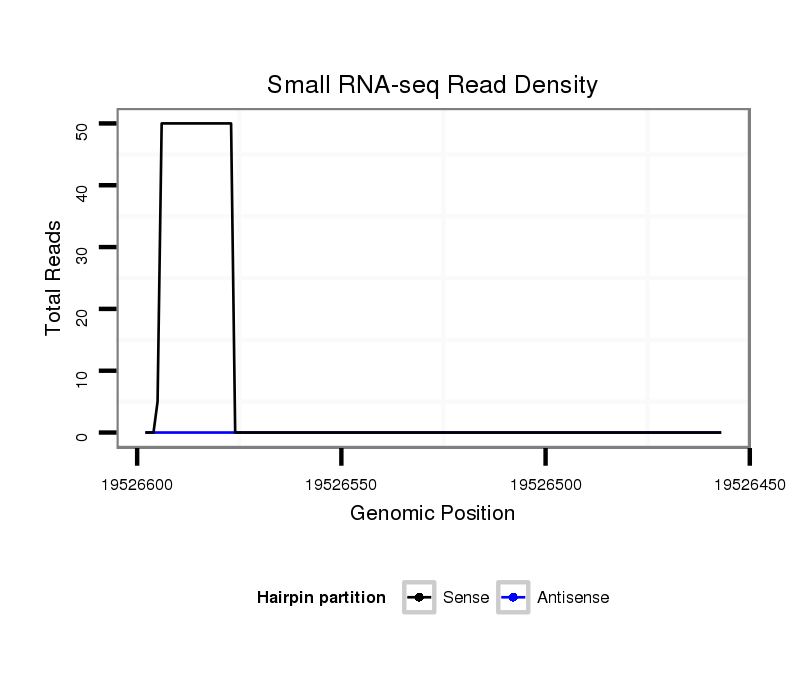
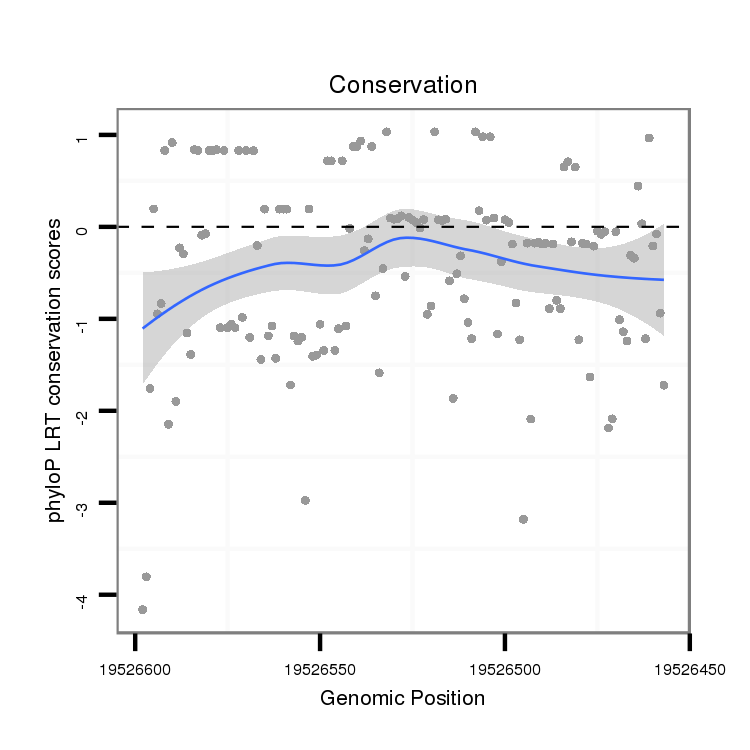
ID:dsi_225 |
Coordinate:x:19526507-19526548 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
intergenic
No Repeatable elements found
|
AAAGTTCCATCGTCTGTGGCATGATTCGGGCGACACGCCCGATCTCCCTTGCTTCTTGGAGGGCTGGCTCTTCCGGACCCTGACCTAGGCCGGCCATTTTCGGGGCTTCTTCTTCATGTTGCTTCTGATCGTCTGGGCTCAA
***********************************.((.(((.....((.((((.....((((((((.....)))).)))).....)))).))......)))..)).*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | O001 Testis |
SRR902009 testis |
O002 Head |
SRR618934 dsim w501 ovaries |
|---|---|---|---|---|---|---|---|---|---|
| ....TTCCATCGTCTGTGGCAT........................................................................................................................ | 18 | 0 | 3 | 45.00 | 135 | 132 | 0 | 3 | 0 |
| ...GTTCCATCGTCTGTGGCAT........................................................................................................................ | 19 | 0 | 3 | 5.00 | 15 | 15 | 0 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCGTAAT..................................................................................................................... | 22 | 3 | 9 | 1.56 | 14 | 0 | 14 | 0 | 0 |
| ....TTCCATCGTCTGTGGCGTTAT..................................................................................................................... | 21 | 2 | 9 | 0.67 | 6 | 0 | 6 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCGTGTT..................................................................................................................... | 22 | 3 | 11 | 0.45 | 5 | 0 | 5 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCGTGA...................................................................................................................... | 21 | 2 | 9 | 0.44 | 4 | 0 | 4 | 0 | 0 |
| ....TTCCATCGTCTGTGGCGTAATA.................................................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCAA........................................................................................................................ | 19 | 2 | 13 | 0.23 | 3 | 0 | 3 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCATTA...................................................................................................................... | 21 | 2 | 9 | 0.22 | 2 | 0 | 2 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCGTGTTT.................................................................................................................... | 23 | 3 | 10 | 0.20 | 2 | 0 | 2 | 0 | 0 |
| ....TTCCATCGTCTGTGGCGTAA...................................................................................................................... | 20 | 2 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 |
| .....TCCATCGTCTGTGGCGTTAT..................................................................................................................... | 20 | 2 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCGTAATT.................................................................................................................... | 23 | 3 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 |
| ...GTTCCATCGTCTGTGGCGTTA...................................................................................................................... | 21 | 2 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCA......................................................................................................................... | 18 | 1 | 10 | 0.10 | 1 | 0 | 1 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCAT........................................................................................................................ | 19 | 1 | 10 | 0.10 | 1 | 0 | 1 | 0 | 0 |
| ....TTCCATCGTCTGTGGCGTGT...................................................................................................................... | 20 | 2 | 10 | 0.10 | 1 | 0 | 1 | 0 | 0 |
| ...ATTCCATCGTCTGTGGCTTAA...................................................................................................................... | 21 | 3 | 11 | 0.09 | 1 | 0 | 1 | 0 | 0 |
| ....TTCCATCGTCTGTGGCGTAAA..................................................................................................................... | 21 | 3 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 |
| ...ATTCCATCGTCTGTGGTAT........................................................................................................................ | 19 | 2 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 |
| .....................................................TCTTTGAGGGCTGGC.......................................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| ......CCATCGTCTGTGGCGTATTT.................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 |
| .................................CGAGCCCGATCTCCCT............................................................................................. | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
|
TTTCAAGGTAGCAGACACCGTACTAAGCCCGCTGTGCGGGCTAGAGGGAACGAAGAACCTCCCGACCGAGAAGGCCTGGGACTGGATCCGGCCGGTAAAAGCCCCGAAGAAGAAGTACAACGAAGACTAGCAGACCCGAGTT
***********************************.((.(((.....((.((((.....((((((((.....)))).)))).....)))).))......)))..)).*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR618934 dsim w501 ovaries |
|---|---|---|---|---|---|---|
| .................................GTGCGGGCTAGAGGAA............................................................................................. | 16 | 1 | 3 | 0.33 | 1 | 1 |
| ...........................................................................CTGGGACTCGATCAGCCCG................................................ | 19 | 3 | 9 | 0.11 | 1 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | x:19526457-19526598 - | dsi_225 | AAAGTTCCATCG--------TCTGTGGCATGATTC---------GGGCGACACGCCCGATCTCCCTTGCTTCTTGGAGGGCTGGCTCTT--------------------------------------CCGGACCCTGACCTAGGCCGGCCATTTTCGGG-GCTTCTT------------CTTCATGTTGCTT-C-----TGATCGTCTGGGCTCA-A |
| droSec2 | scaffold_701:361-502 + | GCAGTTCCATCG--------TTTGTGGCATGATTC---------GGGCGCCACTCCCCACCGCACTTGCTCCTTGGAGGGCTGGCTCTT--------------------------------------CCGGACCCTGACCTAGGCCGGCCATTTTCGGC-GGTTCTT------------CTTCATGTTGCTT-C-----TGATCGTCTGTGCTCA-A | |
| dm3 | chrX:1722303-1722363 + | AAAGTTCAAGCA--------CCTGTGGCAGGCAGC---------TGCCGCCCTGCCCGCCACCCGCGGCGTCCT------------------------------------------------------------------------------------------------------------------------------------------TGA-G | |
| droYak3 | v2_chrUn_001:498571-498713 - | CGGGTG-GACCG--------------------------------------------------------------------CGGGTTAGGCCGTCGCCTACCCCTGCCTACCCCCTCGGCGGGCCCTTCGGGACTCTGACACAGAACGGCCAGCCTCGATTGGTGCTCCGTAGTGCGTGTATCCATGGTGGTTTCGAACTTTTTAT----GTCTCG-A | |
| droBia1 | scf7180000302292:301977-302026 - | TGAGTC--------------------------------------------------------------------------------------------------------------------------------------------GGCCATTTTTATT-GCATTCT------------CCTCATGTCACTT-C-----TCTTCC-------TTT-G | |
| droTak1 | scf7180000415363:171648-171672 - | GCTGCT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T-T-----CGCCCGCTCGGACTCA-G | |
| droRho1 | scf7180000770812:4136-4216 + | GGAGGT--------------------------------------------------------------------------GCGGCCCTT--------------------------------------CCGGAACCGGGTGCGCCGCAGCCAGTTCCTGG-ACTTCTC------------CACCAAGGTGGGC-C-----TCGTGGGCT----GCG-C | |
| droKik1 | scf7180000302468:359728-359767 - | TTC---------------------------------------------------------------------TTGGAGAGCTCGCTCTT--------------------------------------CCGCTGGCTAAACTGAGCCG---------------------------------------------------------------------- | |
| droPer2 | scaffold_9:120630-120683 + | CCT---------------------------------------------------------------CGCCTCAAGGACGGAAAGCTCTA--------------------------------------TCTGACCCTGAAGCAGGCCGGTCTTCTT-------------------------------------------------------------- | |
| droVir3 | scaffold_13049:7315885-7315897 - | CGCATT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCTCA-A | |
| droMoj3 | scaffold_3664:1423-1476 + | AAAGACCCATGGCCCTTGCCAATGAAGCATGAATCCGAGTAGTAGGGCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------ACACG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
Generated: 05/15/2015 at 03:22 PM