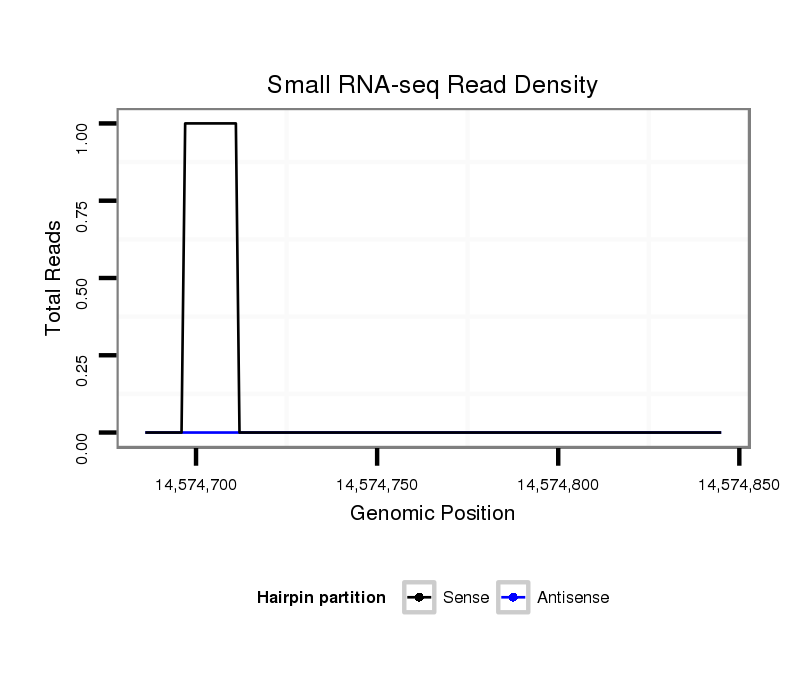
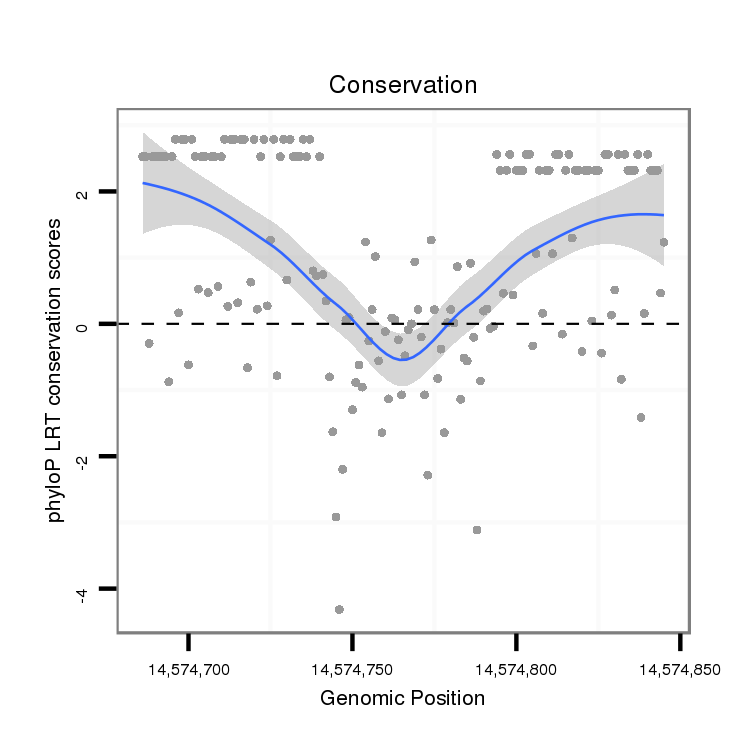
ID:dsi_21672 |
Coordinate:3l:14574736-14574795 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
intergenic
No Repeatable elements found
|
GCGCCGGCCGACAATTCCCCGCCGGACAAAAAGCTGCAACTCCAATGGGTGTAAGTATATTGAATTGGAATTCTGTTGCAACCAATCCTACAGAGATTCGACTGTTTTAGCCATGGCTATCGCGGCATTGATGCGCGGCGAAATCTCTGGGTTCTGCCCT
**************************************************..............(((((.((((((.............)))))).))))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR553488 RT_0-2 hours eggs |
SRR618934 dsim w501 ovaries |
SRR553486 Makindu_3 day-old ovaries |
GSM343915 embryo |
SRR553487 NRT_0-2 hours eggs |
|---|---|---|---|---|---|---|---|---|---|---|
| ...........CAATTCCCCGCCGGA...................................................................................................................................... | 15 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .................................................................TGGAATGCTGATGCAACC............................................................................. | 18 | 2 | 4 | 0.50 | 2 | 0 | 2 | 0 | 0 | 0 |
| .............................................TGGGTGTAGGAATATTGA................................................................................................. | 18 | 2 | 7 | 0.43 | 3 | 1 | 0 | 2 | 0 | 0 |
| ................................................................................................................ATGGCCATCGCGGCGTTGATC........................... | 21 | 3 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 |
| .............................................TGGGTGTAGGAATATTG.................................................................................................. | 17 | 2 | 13 | 0.23 | 3 | 3 | 0 | 0 | 0 | 0 |
| ...................................GCTGCTCCAATGGGTGTAAC......................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 |
| ...........CGATTCCCCGCCGGA...................................................................................................................................... | 15 | 1 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 |
| ..............................................GGGTGTAGGAATATTGA................................................................................................. | 17 | 2 | 15 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..................................................GTAAGTACATTGAGTGGGA........................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................AACTACAGAGATTCGA........................................................... | 16 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 |
|
CGCGGCCGGCTGTTAAGGGGCGGCCTGTTTTTCGACGTTGAGGTTACCCACATTCATATAACTTAACCTTAAGACAACGTTGGTTAGGATGTCTCTAAGCTGACAAAATCGGTACCGATAGCGCCGTAACTACGCGCCGCTTTAGAGACCCAAGACGGGA
**************************************************..............(((((.((((((.............)))))).))))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR553486 Makindu_3 day-old ovaries |
SRR553488 RT_0-2 hours eggs |
SRR618934 dsim w501 ovaries |
M023 head |
|---|---|---|---|---|---|---|---|---|---|
| .......................................................................AGACAACGTTCGTTA.......................................................................... | 15 | 1 | 8 | 0.75 | 6 | 6 | 0 | 0 | 0 |
| .......................................................................AGACAACGTTCGTTATCAT...................................................................... | 19 | 3 | 20 | 0.60 | 12 | 12 | 0 | 0 | 0 |
| ................................................................................TGGATAGGATGTGTCTAGGC............................................................ | 20 | 3 | 4 | 0.50 | 2 | 0 | 2 | 0 | 0 |
| .......................................................................AGACAACGTTCGTTATCATG..................................................................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 |
| .....................................................................................................................ATCGCGCCGTAACTA............................ | 15 | 1 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 |
| ...................................................................................................CTGACAAAATCGGAA.............................................. | 15 | 1 | 8 | 0.25 | 2 | 0 | 0 | 2 | 0 |
| .................................................................................GGATAGGATGTGTCTAGGC............................................................ | 19 | 3 | 14 | 0.14 | 2 | 0 | 2 | 0 | 0 |
| ......................................................................................GGATGTCTCTAAGCTTG......................................................... | 17 | 2 | 13 | 0.08 | 1 | 0 | 1 | 0 | 0 |
| .....................................................................TCAGACAACGTTCGTTA.......................................................................... | 17 | 2 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 |
| .............................................................................................................................GTAAGTACGCGCAGCATTA................ | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 1 |
| .....................................................................................GGGATGTCACTAAGCT........................................................... | 16 | 2 | 17 | 0.06 | 1 | 1 | 0 | 0 | 0 |
| ..............................ATCGACCTTGAGGTTACCA............................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | 3l:14574686-14574845 + | dsi_21672 | GCGCCGGCCGACAATTCCCCGCCGGACAAAAAGCTGCAACTCCAATGGGTGTAAGTATA--TTGA--A-------------T-------TG--------GAATTCTGTTGCAA---------CCAATCCTA-----CAGAGA----T------------TCGA-C-TGTTTTAGCCATGGCTATCGCGGCATTGATGCGCGGCGAAATCTCTGGGTTCTGCCCT |
| droSec2 | scaffold_0:7051123-7051283 + | GCGCCGGCCGACAATTCCCCGCCGGACAAAAAACTGCAGCTTCAATGGGTGTATGTATA--TCGA--A-------------T-------TC--------GACTTCTGTTTTAA---------CCAATCCTA-----CAGAGA----T------------TCGA-CTGTTCTTAGCCATGGCTATCGCGGCATTGATGCGCGGCGAAATCTCTGGGTTCTGCCCT | |
| dm3 | chr3L:14932847-14933006 + | GCGCCGGCCGACAATTCCCCGCCGGACAAAAAACTGCAACTCCAATGGGTGTTAGTATA--TTGA--A-------------T-------TC--------GACTTCCGTTTTAA---------CCAATCCTA-----CAGAGA----T------------TCGA-C-TGTTTTAGCCATGGCTATCGCGGCATTGATGCGCGGCGAAATCTCTGGGTTTTGCCCT | |
| droEre2 | scaffold_4784:14894043-14894201 + | GCGCCGGCGGACAATTCCCCGCCGGACAAAAAACTGCAGCTCCAATGGGTGTATGTATA--TGCA------------------------CC--------GACTTTCGTTTCA-----A---ACCATTCCTA-----CAGAGA----T------------TTGA-C-CGTTTCAGCCATGGCTACCGCGGCATTGACGCGCGCCGAAATCTCTGGGTTCTGCCCT | |
| droYak3 | 3L:15019414-15019575 + | GCGCCGGCGGACAACTCCCCGCCGGATAAAAAACTGCAGCTCCAATGGGTGTATGTATA--TCGA--A-------------CGA-----CC--------AACTTCCGATTCCG---------CCATTCCTA-----CAGCGA----T------------TTGA-A-TGTTTTAGCCATGGCTACCGCGGCATTGATGCGCGGCGAAATCTCTGGGTTCTGCCCT | |
| droEug1 | scf7180000409456:1191283-1191434 + | GCACCGGCCGACAATTCCCCGCCGGACAAAAAGCTGCAGCTACAATGGGTGTAAGTATA--GTAA--A-------------A-------TG--------AACCTAA----------AA---A---TCTTTA-----CTGAGA----T------------GTAA---TTTTCTAGCCATGGCTACCGTGGCATTGATGCACGGCGCAATCTCTGGGTCCTGCCCT | |
| droBia1 | scf7180000302428:4947548-4947707 - | GCGCCGGCGGACAATTCCCCGCCGGACAAAAAGCTGCAGCTCCAATGGGTGTAAGTATC--TGTATAT-------------ATGCTTTGAG----------------GATCT--GCAA---A---TCTCTA-----ATGAGA----T--------------GT-C-TTTTTTAGCCATGGCTACCGCGGCATCGATGCGCGGCGCAATCTCTGGGTCCTGCCCT | |
| droTak1 | scf7180000415149:82361-82517 - | GCGCCGGCGGACAACTCCCCGCCGGACAAAAAGCTTCAGCTCCAATGGGTGTAAGTATT--GTGA--A-------------A-------TAAATT-------TCAT-----A----AA---T---TCTTTG-----TTATGT----T------------GTGACCTTTTTTTAGCCACGGCTACCGCGGCATCGATGCGCGGCGCAATCTTTGGGTTTTGCCCT | |
| droEle1 | scf7180000491000:199047-199203 + | GCGCCGGCGGACAACTCCCCGCCGGACAAAAAGCTACAGCTCCAATGGGTGTAAGTTAA--AAAAAAA-------------TTATTGAA------------------AATA-----A----G---CCACTA-----CCAAAA----T------------GTAA-CACTTTCCAGCCATGGCTATCGCGGCATTGATGCGCGGCGAAACCTCTGGGTCCTGCCCT | |
| droRho1 | scf7180000778965:1969-2130 + | GCGCCGGCAGACAACTCCCCGCCGGACAAGAAACTGCAGCTCCAATGGGTGTAAGTACT--CAGA--A-------------T-------CA--------AACTTAAAATTCG-----ATATA---CCTATA-----CCAAGA----T------------GAAA-CTCCTTCTAGCCATGGCTACCGCGGCATTGATGCGCGGCGTAATCTCTGGGTCCTGCCCT | |
| droFic1 | scf7180000453807:389597-389758 + | GCGCCGGCGGACAACTCCCCGCCGGACAAAAAGCTGCAGCTCCAATGGGTGTAAGTATA--CATA--A-------------T-------TAAATC-------TTACATTTC--TTGA----A---TCAATATAAATT----------------------ATAACTGATTTTTAGCCATGGCTACCGCGGCATCGATGCGCGGCGCAATCTCTGGGTTCTGCCCT | |
| droKik1 | scf7180000302383:1090332-1090493 + | GCACCGGCGGACAATTCCCCGCCGGATAAAAAGCTGCAGCTACAATGGGTGTAAGTGGG--GGGT------------------CTTTAATATATC-------TT-----------AA----A---AGATTTTTTTAACTGGT----T---------A--TTG--ATGTCCTTAGCCATGGCTATCGCGGCATAGATGCGCGACGCAATCTGTGGGTGCTGCCCT | |
| droAna3 | scaffold_13337:4183775-4183934 - | GCCCCGGCGGACAACTCCCCGCCGGACAAAAAACTGCAGCTTCAATGGGTGTAAGTAGC--CAGG-AA-------------GTG-----------------CTTCAAATTCG-----A---A---TCCATA-----CTGAGA----TTA----------GAGA-CTTTTTTCAGCCATGGCTACCGCGGCATTGATGCCCGGCGTAATCTCTGGGTTCTGCCCT | |
| droBip1 | scf7180000395450:315777-315940 - | GCCCCGGCGGACAACTCCCCGCCGGACAAAAAACTGCAGCTTCAATGGGTGTAAGTAGC--ATGG-AA-------------GGG-----------------CTTCACATCCG-----A---A---TCTTTA-----CTAAGAATCCTTA----------AATA-CCTTTTCCAGCCATGGCTACCGCGGCATTGATGCCCGGCGTAATCTCTGGGTTCTGCCCT | |
| dp5 | XR_group6:810062-810222 - | GCGCCGGCTGACAACTCGCCGCCGGACAAAAAGTTGCAGCTCCAATGGGTGTAAGCTGG--TGCAGGA-------------GTGTTCACTGTACC------------------GCCA----G---TCACTGTATCAA----------------------GTAA-CTCTTTTCAGCCATGGCTACCGTGGCATCGATGCTCGCCGGAACCTTTGGGTCTTGCCGT | |
| droPer2 | scaffold_12:1489164-1489324 - | GCGCCGGCTGACAACTCGCCGCCGGACAAAAAGTTGCAGCTCCAATGGGTGTAAGCTGG--TGCAGGA-------------GTGTTCACTGTACC------------------TCCA----G---TCACTGTATCAA----------------------GTAA-CTACTTTCAGTCATGGCTACCGTGGCATCGATGCTCGCCGGAACCTGTGGGTCTTGCCGT | |
| droWil2 | scf2_1100000004949:574333-574391 - | GCACCGGCTGATAATTCACCGCCCGATAAAAAACTGCAATTGCAGTGGGTGTAAGTATA--------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droVir3 | scaffold_13049:17792441-17792614 - | GCGCCGGCAGAGAACTCGCCTCCAGACAAGAAATTACAGCTCCAGTGGGTGTAAGTAGT--TAATTAC--------CCACGA-----TGAA----------TCTCA--------ACGC---A---TGAATA-----CTGATT----C--CTCTTCCGGCCCGATCTCCATTTAGCCACGGCTATCGAGGCATCGATGCACGCCGCAACTTGTGGGTGCTGCCAA | |
| droMoj3 | scaffold_6680:22869978-22870157 - | GCACCGGCTGAAAACTCGCCCCCAGACAAAAAGCTACAGCTCCAGTGGGTGTGAGTATA--CATTTATGCCATAAGCCCCGT-----GCCA----------GCTCA--------GCAT---T---TGCTCA-----TTCATT----C--ACTCACTGTTTGG--CTTTCGATAGACACGGCTACAGAGGAATCGATGCGCGCCGCAATCTCTGGGTACTGCCAA | |
| droGri2 | scaffold_15110:18498981-18499146 - | GCGCCGGCAGAGAATTCGCCCCCAGACAAGAAACTACAGCTACAATGGGTGTAAGCACTAATTGA--A-------------TAA-CCA-CA----GAATTACTTTCATTTCAA---------ATATTATTA-----CGC--------------------TC---GTTATTTTAGCCATGGCTATCGAGGCATCGATGCACGCCGCAACTTGTGGGTGCTGCCAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/18/2015 at 08:42 AM