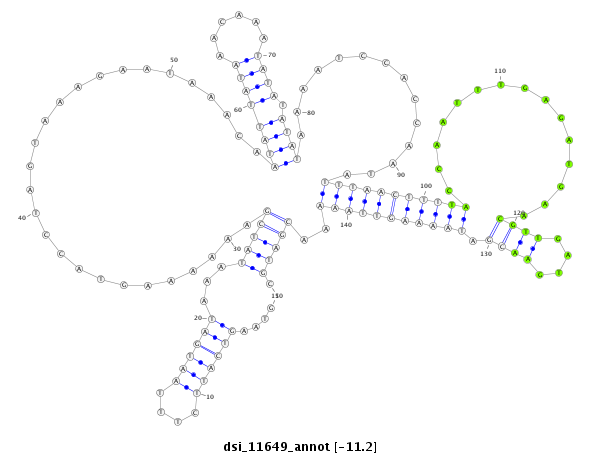
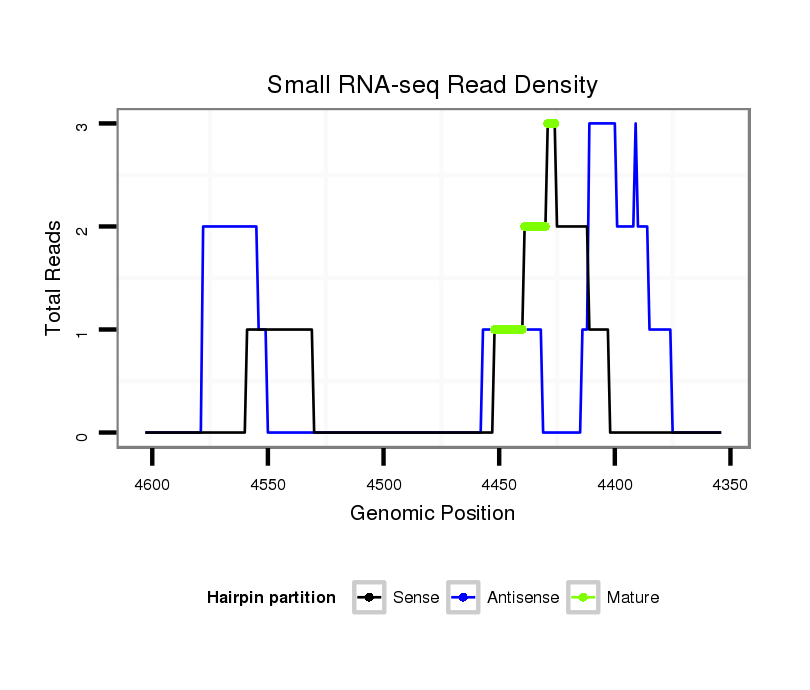
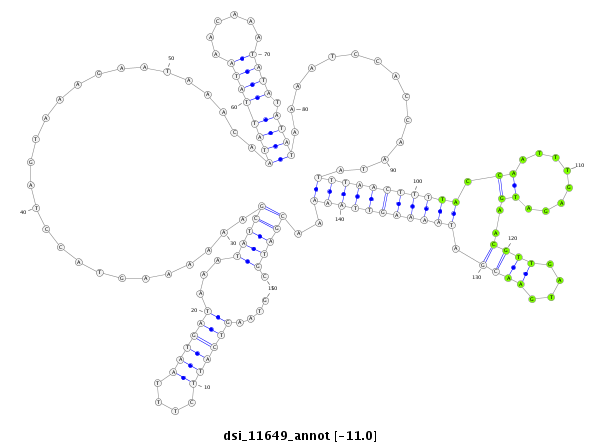
ID:dsi_11649 |
Coordinate:node_9712:4404-4553 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
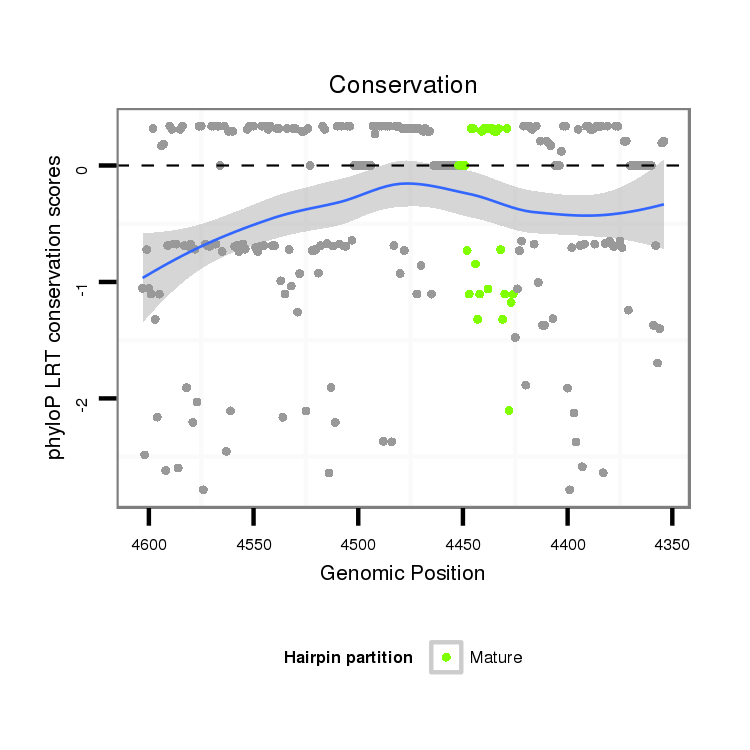
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -11.0 | -10.9 | -10.8 |
 |
 |
 |
intergenic
| Name | Class | Family | Strand |
| AT_rich | Low_complexity | Low_complexity | + |
|
TAACATGAAGTATACAAACAAACATTATTTAGCTTAACAACGACCTTCACGTAAGTCATTCTTTAATGATAAATATCGAAAAAAGTACCTAGTAAAGAATAAACAATATTATAAACAAATATATATATAAAATCCACCAATATTTAACTTTTACCAATTTGAGATGAACGTTGATGAACGATAAAAGTTAAAAACGATGCATTCTGGTGTTAAACGAAAACCTTTCTTAGTTTGAATTTGAATTAAGTGA
**************************************************....((((((....))))))...(((((...........................((((((((......)))).))))..............(((((((((((...............((((....)))).)))))))))))..))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR553487 NRT_0-2 hours eggs |
M053 female body |
SRR553488 RT_0-2 hours eggs |
M023 head |
M024 male body |
|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................................................................TGAACGATAAAAGTTAAAAACGATGCA................................................. | 27 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ............................................CTTCACGTAAGTCATTCTTTAATGATAAA................................................................................................................................................................................. | 29 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................TACCAATTTGAGATGAACGTTGATGAA........................................................................ | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................................................TGAACGTTGATGAACGATAAAAGTTAAA.......................................................... | 28 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................................................................................AATTTTTACCATGTTGAGATG.................................................................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................................................................................................................TAGAGAAGAACGTTGGTGA......................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 |
|
ATTGTACTTCATATGTTTGTTTGTAATAAATCGAATTGTTGCTGGAAGTGCATTCAGTAAGAAATTACTATTTATAGCTTTTTTCATGGATCATTTCTTATTTGTTATAATATTTGTTTATATATATATTTTAGGTGGTTATAAATTGAAAATGGTTAAACTCTACTTGCAACTACTTGCTATTTTCAATTTTTGCTACGTAAGACCACAATTTGCTTTTGGAAAGAATCAAACTTAAACTTAATTCACT
**************************************************....((((((....))))))...(((((...........................((((((((......)))).))))..............(((((((((((...............((((....)))).)))))))))))..))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR618934 dsim w501 ovaries |
SRR553486 Makindu_3 day-old ovaries |
SRR553487 NRT_0-2 hours eggs |
SRR553488 RT_0-2 hours eggs |
|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................................TTGCTTTTGGAAAGAA...................... | 16 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ..................................................................................................................................................TGAAAATGGTTAAACTCTACTTGCAA.............................................................................. | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ................................................................................................................................................................................................TTGCTACGTAAGACCACAATT..................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| ................................................................................................................................................................................................TTGCTACGTAAGACCACAATTTGCTT................................ | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| .........................ATAAATCGAATTGTTGCTGGAAGT......................................................................................................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| .........................ATAAATCGAATTGTTGCTGGAAGTGCAT..................................................................................................................................................................................................... | 28 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 |
| .............................................................................................................................................................................................TTTTTGCTACGTAAG.............................................. | 15 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .....................................................TCAGTACGTAATTACTAATTAT............................................................................................................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 |
| ...............................................................................................................................................................................................................ACAATTTGCTTTAGGTAAG........................ | 19 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 |
| .....................................................................................................................................................................................................................TGCTGTTGGAAAGAA...................... | 15 | 1 | 10 | 0.10 | 1 | 0 | 0 | 1 | 0 |
| ...............................................................................................................................................................................................TTTGCCACGAAAGACCA.......................................... | 17 | 2 | 15 | 0.07 | 1 | 1 | 0 | 0 | 0 |
| ........................AATAAGTCGAATTGT................................................................................................................................................................................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| ..........AGATGTTTGTTTGTA................................................................................................................................................................................................................................. | 15 | 1 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | node_9712:4354-4603 - | dsi_11649 | TAACATG-AAGT-ATACAAACA--AACATTATTTAGCTTAACAACGACCTTCAC-GTAA-GTCATTC-------TTT------------------AATGATAAATATCGAAAAAAGTACCTAGTAAAGAATAAACAATATTATAAACA--------------AATATATATATA--AAAT--------CCACCAATATTTAACTTTTACCAA-TTTGAGATGAACGTTGATGAACGATAAAAGTTAAAAACGATGCATTCTGGTGTTAAACGAAAACCTTTCTTAGTTTGAATTTGAATTAAGTGA |
| droSec2 | scaffold_71:3658-3824 + | TT-----------CCACTTCCACAGATTCTTTTATATTTTA-AAT---TCTTTGAGTAATTTTACTC-------TCCAGAATCCATAATCTA----------------------AGCACCCAACATAACATA---------A-AAAAACTGTTGTTTTACGAAAGGTA---------------------------------------------------------------------TAGAAGATCATTAC-----ATTTATAGGCATAACGTAAATCTTATTTATTTC-----------TGCAGA | |
| dm3 | chr3LHet:1703739-1703984 - | ACATTTTTCTGTGTCACTTTCA--GTTTCCATTCAATTTTA-AACGTCTCTTTCAGTAATTTTACTC-------TCCAGAATC-------TAAGTAACTTTATATCCCGTA-AATGCACCATACAAAACATA---------A-AAAAACTGTAATTTTACGAAATGTACATATACGAATTCATACTGACCACCT----------------AT-TTTTTGATAAACGTTTTTACTGAATAGAAGATAATTACGT----TTTCTATGCGTAACGTAAAACTTATTTATTTC-----------TGCAGA | |
| droEre2 | scaffold_4929:26525901-26526139 + | ACGTTTTTTT---CCACTTTCATGGCTTATTTTAAATTTTA-CAGGCCTCGTTCAGTAATTGTACTCGTTAACTTCCCGCATC-------CACGGAATATTGAATACCGCA-TTACCACTACAAAAAACATG---------ATAAACACAGTTGTTTTACGAAAGGTACAAATACGAATTAATACTGAACACCT----------------TTATTGTTGATAAACGTCTTTTCTGATAAAAAGATA-----------TTCATTGGCATAACGTAAAATTAATTTTT---------------G---- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 04:05 PM