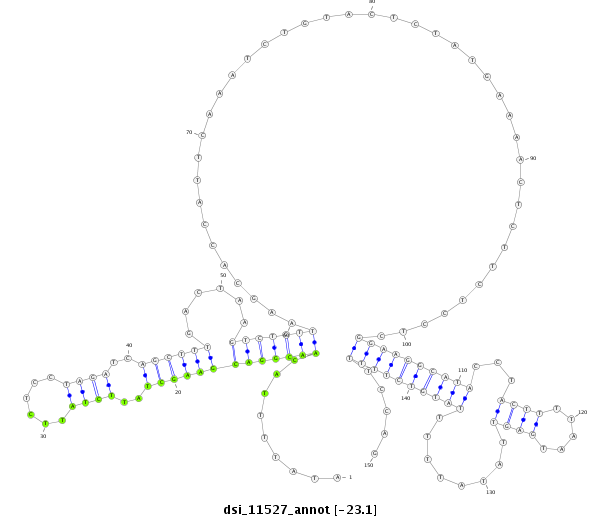
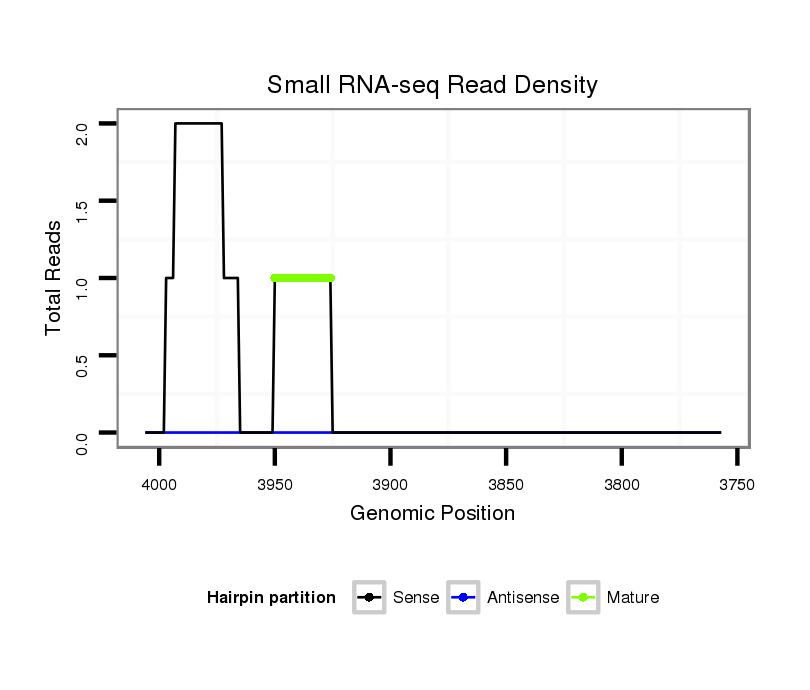
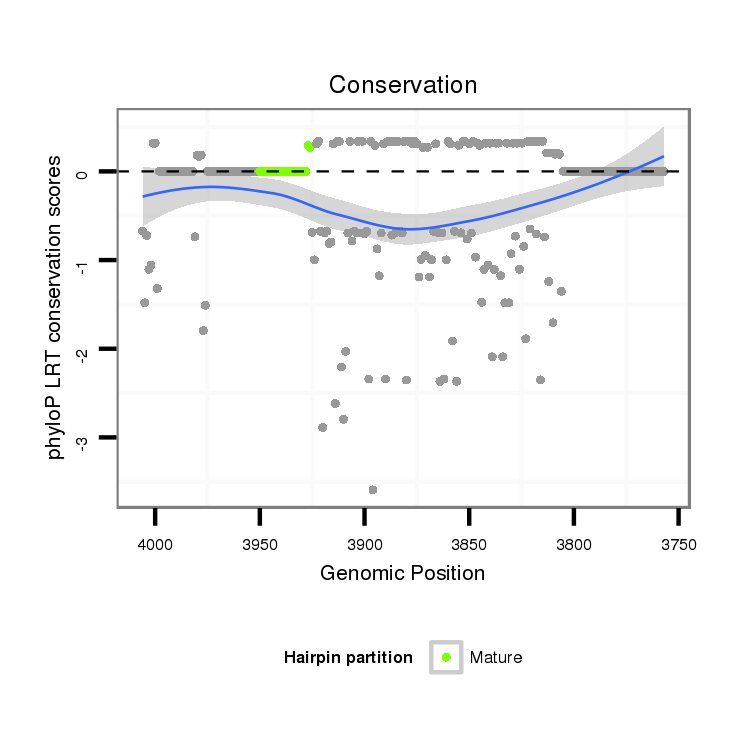
ID:dsi_11527 |
Coordinate:node_9712:3807-3956 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -22.9 | -22.8 | -22.6 |
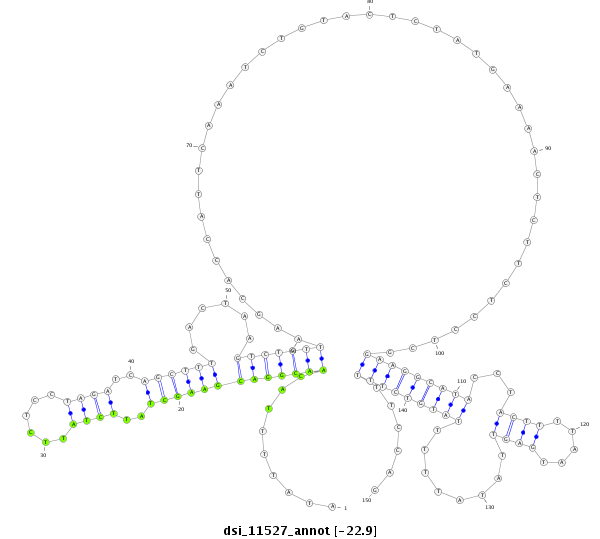 |
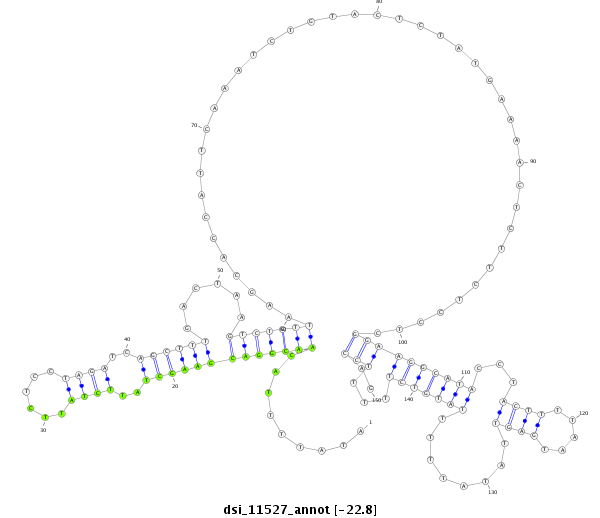 |
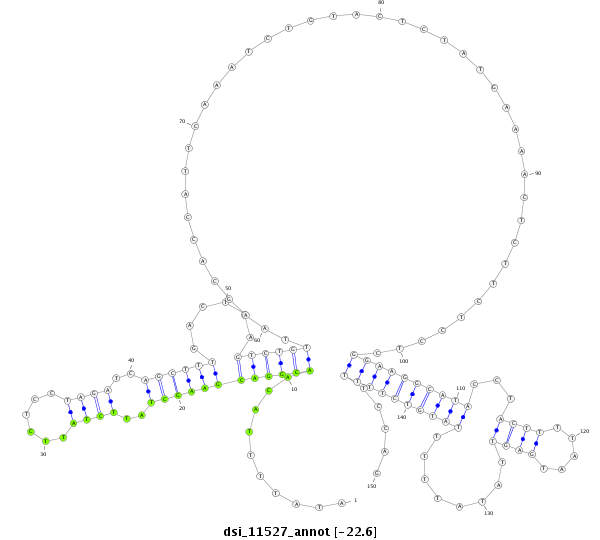 |
intergenic
No Repeatable elements found
|
AGAACTTGTTAAATAAAATTGGCAACTGTCGGGCTTCGTGATTAAACTAAATATTTTACAACGGACGAAGCTATTCTATTCTCCTAGATCAGCTTTGACTAAGTCTGTTAAGCACCATTCAAATCTGTACTCTATGAAAACTCTTCTCCTCGGAAGGCATACCTACTTTTAATGAGTTATATTTTTATGTCTTTTTCCAGGAAGTTGGAAACGGCTTGGGTTACATATCCATAAGTCTACATAAATAGCT
**************************************************.........(((((((((((((..((((......))))..))))))......)))))))..........................................((((((((((...((((.....))))........)))))))))).....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR902008 ovaries |
SRR553488 RT_0-2 hours eggs |
SRR902009 testis |
SRR553485 Chicharo_3 day-old ovaries |
SRR553487 NRT_0-2 hours eggs |
SRR553486 Makindu_3 day-old ovaries |
SRR618934 dsim w501 ovaries |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..AACTTGTTAAATGAAATTGGCACA................................................................................................................................................................................................................................ | 24 | 3 | 3 | 2.00 | 6 | 6 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............TAAAATTGGCAACTGTCGGGCTTCGTGA................................................................................................................................................................................................................. | 28 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................TACAACGGACGAAGCTATTCTATTC......................................................................................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..AACTTGTTAAATGAAATTGGCAC................................................................................................................................................................................................................................. | 23 | 2 | 2 | 1.00 | 2 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........TAAATAAAATTGGCAACTGTCGGGC........................................................................................................................................................................................................................ | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................GTCGGGCTTAGTGAT................................................................................................................................................................................................................ | 15 | 1 | 5 | 0.40 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 |
| ..........AAATAAAATTTACAACCGTCG........................................................................................................................................................................................................................... | 21 | 3 | 11 | 0.27 | 3 | 0 | 0 | 3 | 0 | 0 | 0 | 0 |
| .................ATTGGCAACTGGCGGGTTT...................................................................................................................................................................................................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................TCTGGCTTCGTGATT............................................................................................................................................................................................................... | 15 | 1 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................TCGGGCTTAGTGATT............................................................................................................................................................................................................... | 15 | 1 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .............................................................................................................................................................................................TCTTTGTCCAGGAAGTAGAA......................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........AATAAAATTTACAACCGTCG........................................................................................................................................................................................................................... | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................GTTGGAACGGGCTTGG............................... | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TCTTGAACAATTTATTTTAACCGTTGACAGCCCGAAGCACTAATTTGATTTATAAAATGTTGCCTGCTTCGATAAGATAAGAGGATCTAGTCGAAACTGATTCAGACAATTCGTGGTAAGTTTAGACATGAGATACTTTTGAGAAGAGGAGCCTTCCGTATGGATGAAAATTACTCAATATAAAAATACAGAAAAAGGTCCTTCAACCTTTGCCGAACCCAATGTATAGGTATTCAGATGTATTTATCGA
**************************************************.........(((((((((((((..((((......))))..))))))......)))))))..........................................((((((((((...((((.....))))........)))))))))).....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR553487 NRT_0-2 hours eggs |
SRR902009 testis |
SRR553486 Makindu_3 day-old ovaries |
M025 embryo |
SRR618934 dsim w501 ovaries |
|---|---|---|---|---|---|---|---|---|---|---|
| ............................................................................TTAAGAGGATCTAGTCGAAA.......................................................................................................................................................... | 20 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................................................TAGGCATGGGATACTTTTGA............................................................................................................ | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .....................................................AAAAGGTTGCCTGCATCG................................................................................................................................................................................... | 18 | 2 | 8 | 0.25 | 2 | 0 | 0 | 0 | 1 | 1 |
| .........................................................................................................................................................................................................................CCCACTGTATAGGTATTC............... | 18 | 1 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 |
| ........................................................................................................GAAAATTGGTGGTAAGTCTA.............................................................................................................................. | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................................................................................................................................ACAGAAAAAGGTCCC................................................ | 15 | 1 | 16 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droSim2 | node_9712:3757-4006 - | dsi_11527 | AGAACTTGTTAAATAAAATTGGCAACTGTCGGGCTTCGTGATTAAACTAAATATTTTACAACGGACGAAGCTATTCTATTCTCCTAGATCAGCTTTGACTAAGTCTGTTAAGCACCATTCAA-ATCTGTACTCTATGAAAACTCTTCTC-------CTCG---------------------------------------------------------GAAGGCAT----ACCTACTTTTAA---TGAGTTAT-ATT---------------------------------------------------------TTTATGTCTTTTTCCAGGAAGTTGGAAACGGCTTGGGTTACATATCCATAAGTCTACATAAATAGCT |
| droSec2 | scaffold_71:3658-3824 + | T------------------------------------------------------------------------------TCCACTTCCACAGATTCTTTTATATTTTAAAT---TCTTTGAGTAATTTTACTC-------TCCAGAATCCATAATCTAA---------------------GCACCCAACATAACATAA-AAAAACTGTTGTTTTACGAAAGGTAT-----------------------------------------------------AGAAGATCATTACAT-TTATAGGCATAACGTAAATCTTATTTATTTCTGCAGA------------------------------------------------- | |
| dm3 | chr3LHet:1703739-1703984 - | ACATTTTT-----------------CTGTGT--------------------------------------------------CACTTTCA--GTTTCCATTCAATTTTAAACGTCTCTTTCAGTAATTTTACTC-------TCCAGAATC-------TAAGTAACTTTATATCCCGTAAATGCACCATACAAAACATAA-AAAAACTGTAATTTTACGAAATGTACATATACGAATTCATACTGACCACCTAT-TTTTTGATAAACGTTTTTACTGAATAGAAGATAATTACGTTTTCTATGCGTAACGTAAAACTTATTTATTTCTGCAGA------------------------------------------------- | |
| droEre2 | scaffold_4929:26525901-26526139 + | ACGTTTTT-----------------T-----------------------------------------------------TCCACTTTCATGGCTTATTTTAAATTTTACAGGCCTCGTTCAGTAATTGTACTCGTTAACTTCCCGCATC-------CACGGAATATTGAATACCGCATTACCACTACAAAAAACATGATAAACACAGTTGTTTTACGAAAGGTACAAATACGAATTAATACTGAACACCTTTATTGTTGATAAACGTCTTTTCTGATAAAAAGATA------T-TCATTGGCATAACGTAAAATTAATTTTTG--------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 04:05 PM