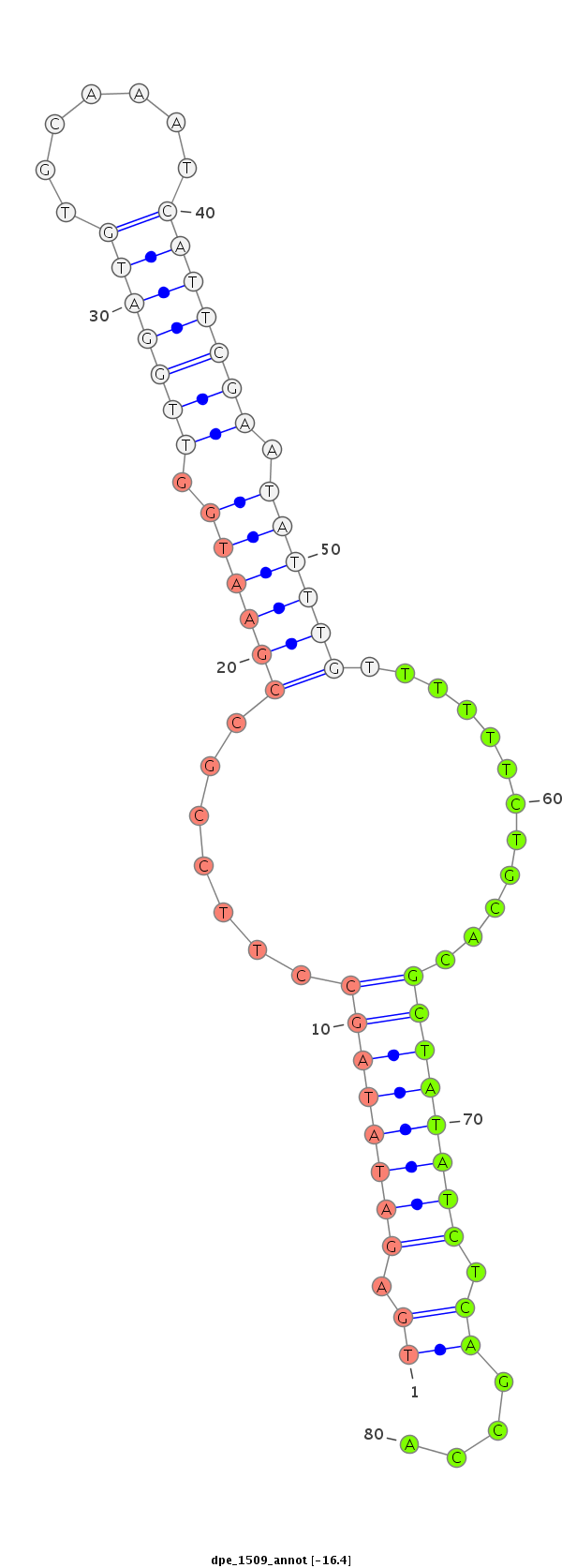
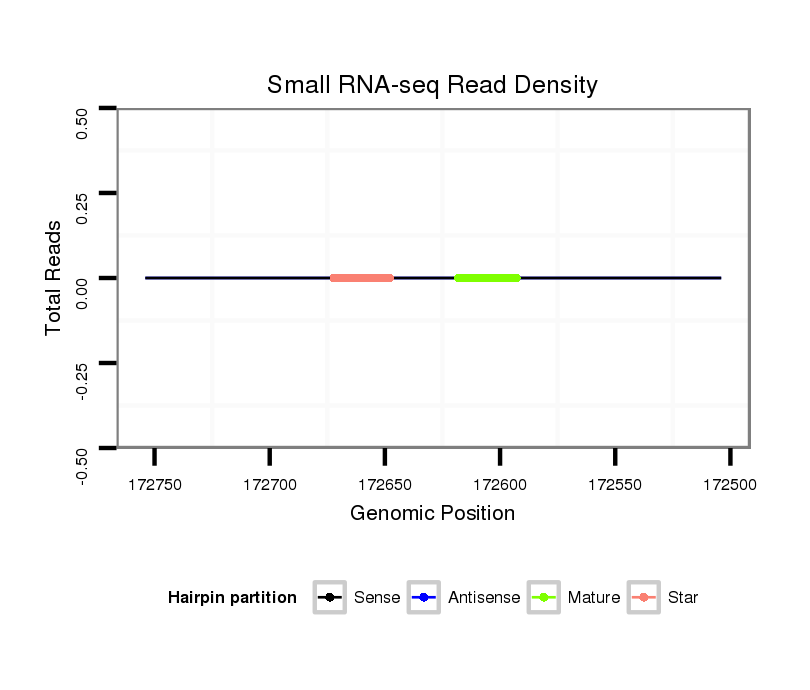
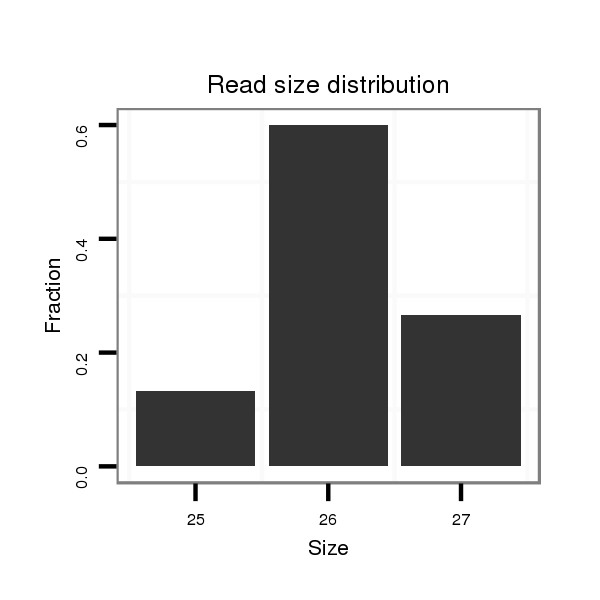
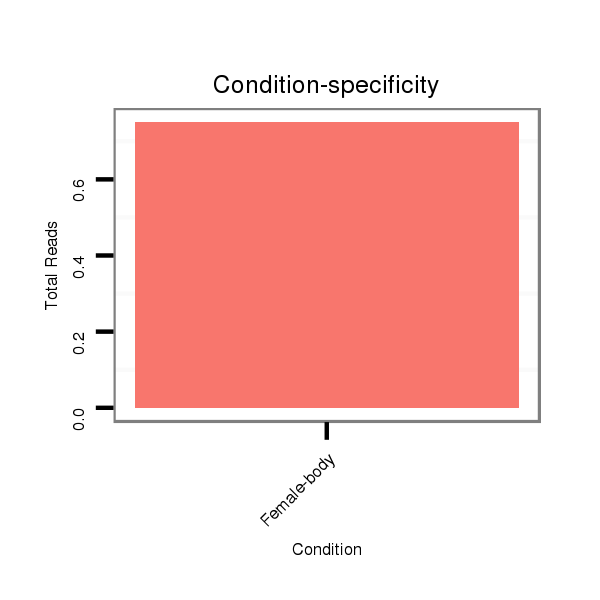

ID:dpe_1509 |
Coordinate:scaffold_35:172554-172704 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -24.1 | -24.1 |
 |
 |
exon [dper_GLEANR_16743:5]; CDS [Dper\GL15522-cds]; intron [Dper\GL15522-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TCGAGATTTTGGTGCCGAACGACAGTCTGGATCCCCACGATCCTGGGAAAGTGGGTCCTGCTCCGATCTGGCGCCATATATCTGAGATATAGCCTTCCGCCGAATGGTTGGATGTGCAAATCATTCGAATATTTGTTTTTTCTGCACGCTATATCTCAGCCATACGGCAGCCGATCGGAGCGTGGCATGGCTCTGCGTGATCGGGAGAGTCCTGCCGACCGATTGGCACCAGAAAAAAGGACATCCTTTTC **********************************************************************************((.((((((((.......((((((.(((((((.......))))))).))))))............)))))))).))....***************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M042 female body |
M021 embryo |
V042 embryo |
|---|---|---|---|---|---|---|---|---|
| ........................................................................................................................................TTTTTCTGCACGCTATATCTCAGCCA......................................................................................... | 26 | 0 | 20 | 0.35 | 7 | 7 | 0 | 0 |
| .......................................................................................................................................TTTTTTCTGCACGCTATATCTCAGCCA......................................................................................... | 27 | 0 | 20 | 0.20 | 4 | 4 | 0 | 0 |
| ................................................................................................................................................................................................CTGCGTGATCGGGAGAGTC........................................ | 19 | 0 | 20 | 0.10 | 2 | 0 | 2 | 0 |
| ................................................................................................................................................................................................CTGCGTGATCGGGAGAGTCCTGCC................................... | 24 | 0 | 20 | 0.10 | 2 | 2 | 0 | 0 |
| ..................................................................................TGAGATATAGCCTTCCGCCGAATGG................................................................................................................................................ | 25 | 0 | 20 | 0.10 | 2 | 2 | 0 | 0 |
| ............................................................................................................................................TCTGCACGCTATATCTCAGCCATACG..................................................................................... | 26 | 0 | 20 | 0.05 | 1 | 1 | 0 | 0 |
| ..................................................................................TGAGATATAGCCTTCCGCCGAATGGT............................................................................................................................................... | 26 | 0 | 20 | 0.05 | 1 | 1 | 0 | 0 |
| ......TTTTGGTGCCGAACGACAGTCTGG............................................................................................................................................................................................................................. | 24 | 0 | 20 | 0.05 | 1 | 1 | 0 | 0 |
| ....................................................................................................CGTACGGTTGGATGTGCGA.................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 |
| ......................CAGTCTGGTTCCCCACGATCCTGG............................................................................................................................................................................................................. | 24 | 1 | 20 | 0.05 | 1 | 1 | 0 | 0 |
| ................................................................................................................................................................................................CTGCGTGATCGGGAGAGTCCTG..................................... | 22 | 0 | 20 | 0.05 | 1 | 1 | 0 | 0 |
|
AGCTCTAAAACCACGGCTTGCTGTCAGACCTAGGGGTGCTAGGACCCTTTCACCCAGGACGAGGCTAGACCGCGGTATATAGACTCTATATCGGAAGGCGGCTTACCAACCTACACGTTTAGTAAGCTTATAAACAAAAAAGACGTGCGATATAGAGTCGGTATGCCGTCGGCTAGCCTCGCACCGTACCGAGACGCACTAGCCCTCTCAGGACGGCTGGCTAACCGTGGTCTTTTTTCCTGTAGGAAAAG
*****************************************************************************************((.((((((((.......((((((.(((((((.......))))))).))))))............)))))))).))....********************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M042 female body |
M021 embryo |
|---|---|---|---|---|---|---|---|
| ........AACCACGGCTTGCTGTCAGACCTA........................................................................................................................................................................................................................... | 24 | 0 | 20 | 0.15 | 3 | 3 | 0 |
| ..........CCACGGCTTGCTGTCAGACC............................................................................................................................................................................................................................. | 20 | 0 | 20 | 0.10 | 2 | 0 | 2 |
| .........ACCACGGCTTGCTGTCAGACCTA........................................................................................................................................................................................................................... | 23 | 0 | 20 | 0.10 | 2 | 2 | 0 |
| .......AAACCACGGCTTGCTGTCAGACCTA........................................................................................................................................................................................................................... | 25 | 0 | 20 | 0.10 | 2 | 0 | 2 |
| .....................................................................................................................................................................................CACCGTACCGAGACGCACTAGCCCTCT........................................... | 27 | 0 | 17 | 0.06 | 1 | 1 | 0 |
| ..........................................................................................................................................................................................................................GGCTAACCGTGGTCTTTTTTCCTGT........ | 25 | 0 | 20 | 0.05 | 1 | 1 | 0 |
| ...TCTAAAACCACGGCTTGCTGTCAGA............................................................................................................................................................................................................................... | 25 | 0 | 20 | 0.05 | 1 | 1 | 0 |
| ....................................................................................................................................................................................................................ACGGCTGGCTAACCGTGGTCTTT................ | 23 | 0 | 20 | 0.05 | 1 | 1 | 0 |
| ..................................................................................................................................................................................................................GGACGGCTGGCTAACCGTGGTCTT................. | 24 | 0 | 20 | 0.05 | 1 | 1 | 0 |
| ......AAAACCACGGCTTGCTGTCAGACCTA........................................................................................................................................................................................................................... | 26 | 0 | 20 | 0.05 | 1 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droPer2 | scaffold_35:172504-172754 - | dpe_1509 | TCGAGATTTTGGTGCCGAACGACAGTCTGGATCCCCACGATCCTGGGAAAGTGGGTCCTGCTCCGATCTGGCGCCATATATCTGAGATATAGCCTTCCGCCGAATGGTTGGATGTGCAAATCATTCGAATATTTGTTTTTTCTGCACGCTATATCTCAGCCATACGGCAGCCGATCGGAGCGTGGCATGGCTCTGCGTGATCGGGAGAGTCCTGCCGACCGATTGGCACC-----------AGAAAAAAGGACATCCTTTTC |
| dp5 | XR_group6:9727489-9727739 + | TCGAGATTTTGGTGCCGAACGACAGTCTGGATCCCCACGATCCTGGGAAAGTGGGTCCTGCTCCGATCTGGCGCCATATATCTGAGATATAGCCTTCCGCCGAATGGTTGGATGTGCAAATCATTCGAATATTTGTTTGTTCTGCACGCTATATCTCAGCTATACGGCAGCCGATCGGAGCGTGGCATGGCTCTGCGTGATCGGGAGAGTCCTGCCGACCGATTGGCATC-----------AGAAAAAAGGACATCCTTTTC | |
| droAna3 | scaffold_13047:463699-463933 + | ACGATCTTTGGATGCATTTCGACTGATTTTGGGGCGCTGATTCCGAAAATGTAGGGCATTGCCAGATCGGATGAGAAATGGCTGAGATATGGGCATTTGAAAAATGT---------------------------GTACTTTCTTTAGATCATATCTCGGCCATTTCTCAACCGATCTGGCAACGCTGTGCATTTTTGGAATCAGGACTCGGATGTCCTTCGAACTGCATCTTTACTTTTTCGAAATCGAACAACTCATTTTC |
| Species | Read alignment | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droPer2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
Generated: 06/08/2015 at 04:20 PM