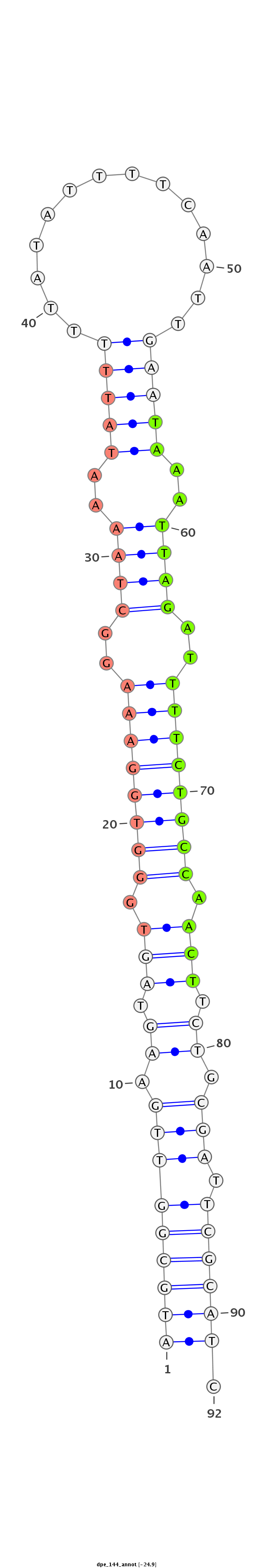
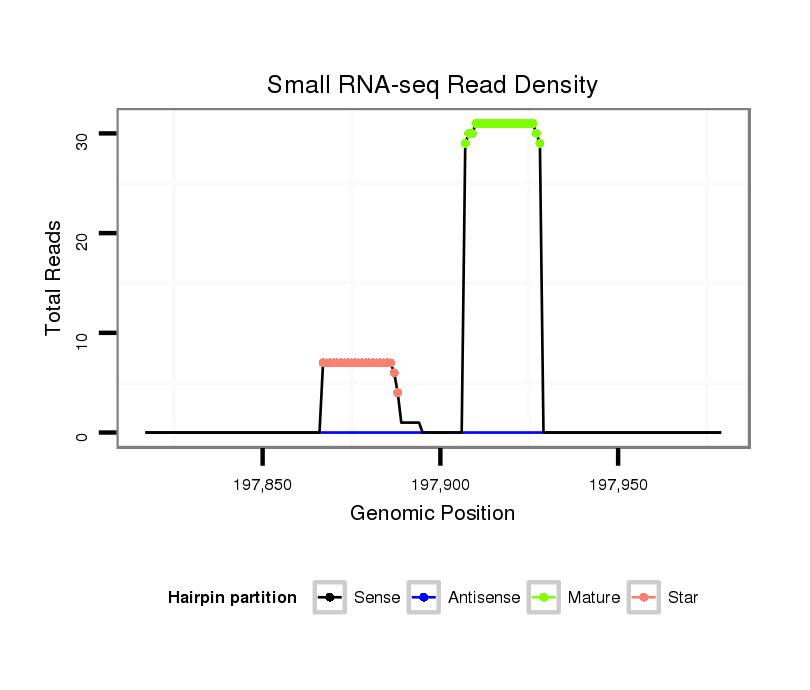
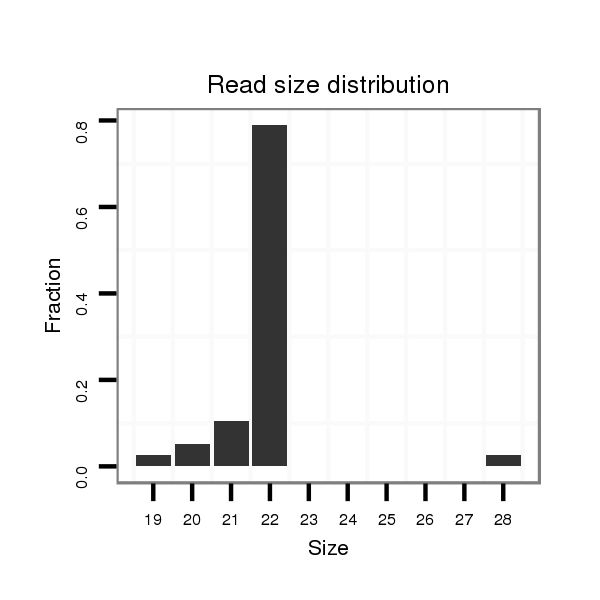
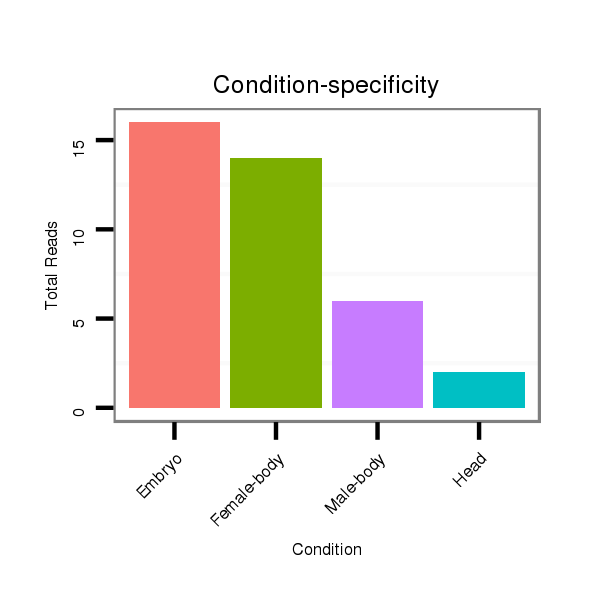
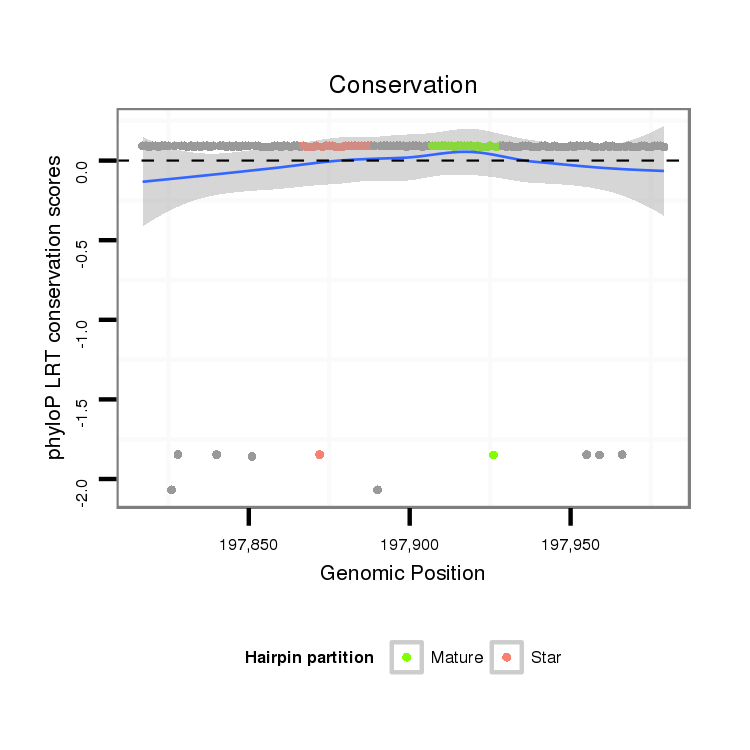
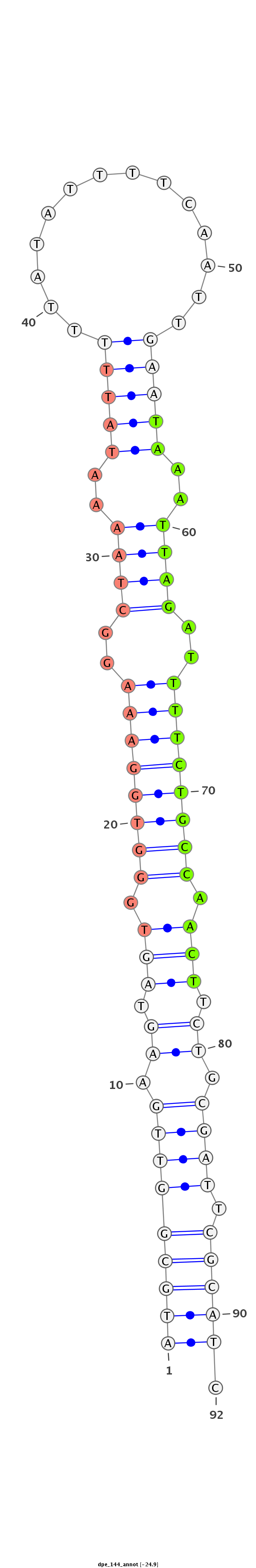
ID:dpe_144 |
Coordinate:scaffold_13:197867-197929 + |
Confidence:confident |
Class:Testes-restricted |
Genomic Locale:intergenic |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -24.9 | -24.6 | -24.6 |
 |
 |
 |
intergenic
No Repeatable elements found
|
ATCATCATATTGTACTGTGAAGAGAACACTGTTTTATGCGGTTGAAGTAGTGGGTGGAAAGGCTAAAATATTTTTATATTTTCAATTGAATAAATTAGATTTTCTGCCAACTTCTGCGATTCGCATCGACCCCTGTTTGTGCACTCTCGGGAGCGATCGTTTC
***********************************(((((((((.((.(((.((((((((..((((..(((((..............)))))..))))..)))))))).))).)).))).)))))).************************************ |
Read size | # Mismatch | Hit Count | Total Norm | Total | M021 embryo |
M042 female body |
V111 male body |
V057 head |
V050 head |
|---|---|---|---|---|---|---|---|---|---|---|
| ..........................................................................................TAAATTAGATTTTCTGCCAACT................................................... | 22 | 0 | 1 | 27.00 | 27 | 14 | 12 | 1 | 0 | 0 |
| ..................................................TGGGTGGAAAGGCTAAAATATT........................................................................................... | 22 | 0 | 1 | 3.00 | 3 | 0 | 1 | 1 | 0 | 1 |
| ..........................................................................................TAAATTAGATTTTCTGCCAACC................................................... | 22 | 1 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 |
| ..................................................TGGGTGGAAAGGCTAAAATAT............................................................................................ | 21 | 0 | 1 | 2.00 | 2 | 0 | 0 | 2 | 0 | 0 |
| ..........................................................................................TAAATTAGATTTTCTGCCAACG................................................... | 22 | 1 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 |
| ..................................................TGGGTGGAAAGGCTAAAATATTTCC........................................................................................ | 25 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..................................................TGGGTGGAAAGGCTAAAATA............................................................................................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| .............................................................................................ATTAGATTTTCTGCCAACT................................................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................TAAATTAGATTTTCTGCCAA..................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................................AAATTAGATTTTCTGCCAACT................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................TAAATTAGATTTTCTGCCAAC.................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..................................................TGGGTGGAAAGGCTAAAATATTTTTATA..................................................................................... | 28 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..............................GTTTTATGCGGTTGAAGTAG................................................................................................................. | 20 | 0 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 |
| ....TGATATTGTAAGGTGAAGA............................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
|
TAGTAGTATAACATGACACTTCTCTTGTGACAAAATACGCCAACTTCATCACCCACCTTTCCGATTTTATAAAAATATAAAAGTTAACTTATTTAATCTAAAAGACGGTTGAAGACGCTAAGCGTAGCTGGGGACAAACACGTGAGAGCCCTCGCTAGCAAAG
************************************(((((((((.((.(((.((((((((..((((..(((((..............)))))..))))..)))))))).))).)).))).)))))).*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V057 head |
|---|---|---|---|---|---|---|
| ..........................................................................................................................................CACCTGAGAACCCCCGCTAG..... | 20 | 3 | 6 | 0.17 | 1 | 1 |
| ...........................................................................................................................................ACCTGAGAGCCCCCTCTAG..... | 19 | 3 | 15 | 0.07 | 1 | 1 |
| Species | Coordinate | ID | Type | Alignment |
|---|---|---|---|---|
| droPer2 | scaffold_13:197817-197979 + | dpe_144 | confident | ATCATCATATTGTACTGTGAAGAGAACACTGTTTTATGCGGTTGAAGTAGTGGGTGGAAAGGCTAAAATATTTTTATATTTTCAATTGAATAAATTAGATTTTCTGCCAACTTCTGCGATTCGCATCGACCCCTGTTTGTGCACTCTCGGGAGCGATCGTTTC |
| dp5 | XL_group1e:8366435-8366597 - | dps_73 | confident | ATCATCATAGTATACTGTGAAGAAAACACTGTTTCATGCGGTTGAAGTAGTGGGTAGAAAGGCTAAAATATTTGTATATTTTCAATTGAATAAATTAGATTTTCTGCCAGCTTCTGCGATTCGCATCGACCCCTGTTTATGCGCTCTCGAGAGCGATCGTTTC |
| Species | Read alignment | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droPer2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
Generated: 10/20/2015 at 06:48 PM