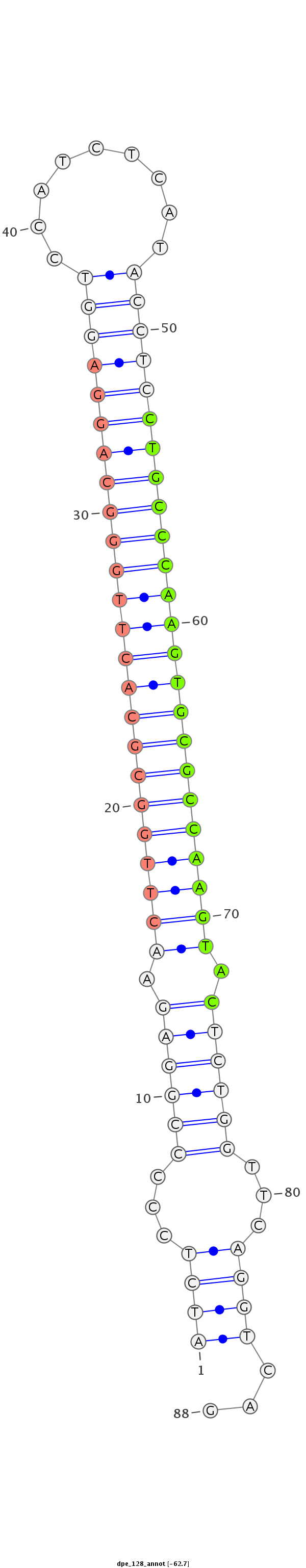
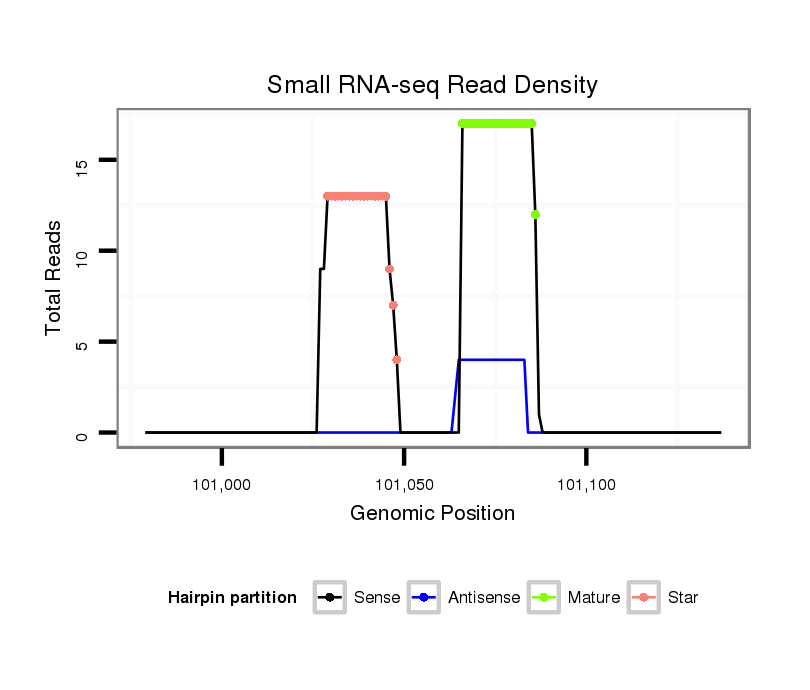
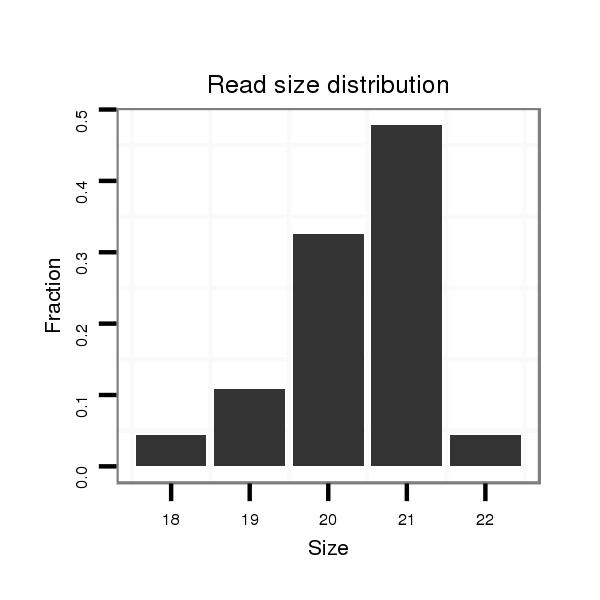
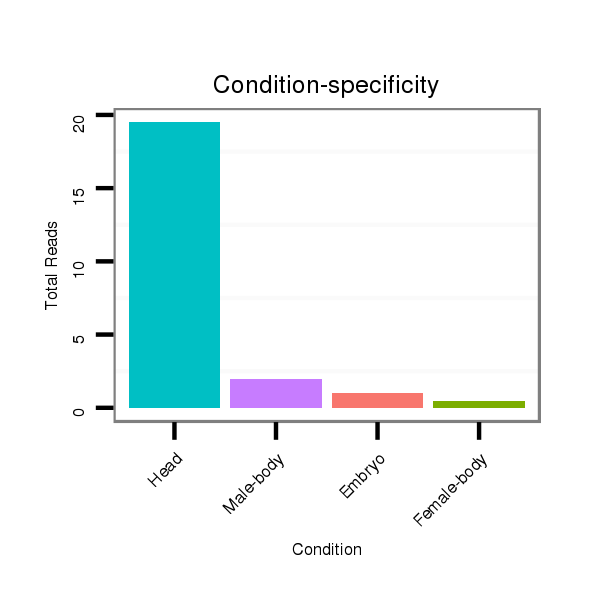
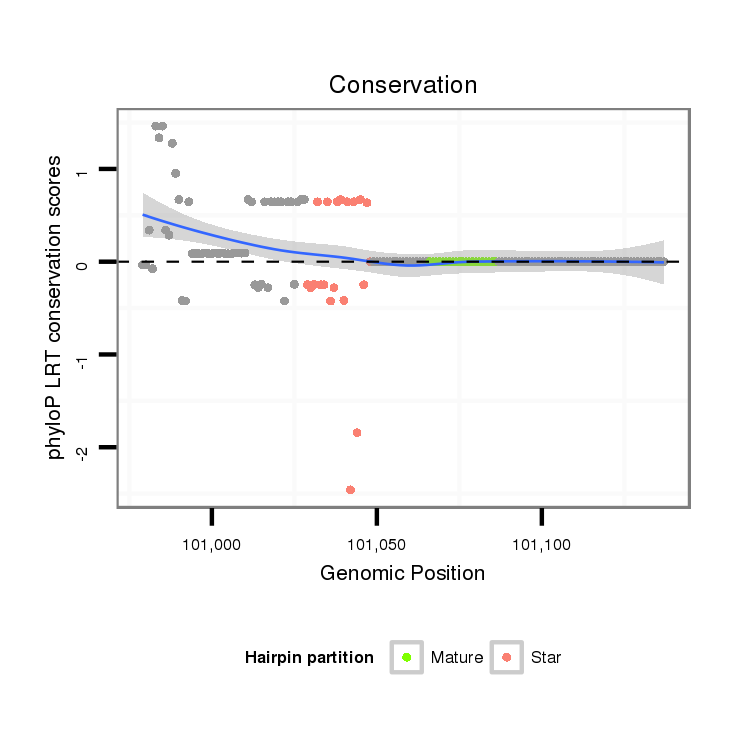
ID:dpe_128 |
Coordinate:scaffold_26:101029-101087 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
intergenic
No Repeatable elements found
| mature | star |
|
GAAAACATCGAATCGCTCCTGCACACCCATTAACGATCTCCCCCGGAGAACTTGGCGCACTTGGGCAGGAGGTCCATCTCATACCTCCTGCCCAAGTGCGCCAAGTACTCTGGTTCAGGTCAGGCCAGAATTCTCTTTACACTGAGGGATTTACACAAT
***********************************((((...((((((.((((((((((((((((((((((((.........)))))))))))))))))))))))).))))))...))))...************************************ |
Read size | # Mismatch | Hit Count | Total Norm | Total | V050 head |
V057 head |
V042 embryo |
V111 male body |
M042 female body |
|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................CTGCCCAAGTGCGCCAAGTAC................................................... | 21 | 0 | 1 | 11.00 | 11 | 5 | 5 | 1 | 0 | 0 |
| .......................................................................................CTGCCCAAGTGCGCCAAGTA.................................................... | 20 | 0 | 1 | 5.00 | 5 | 3 | 0 | 0 | 2 | 0 |
| ................................................AACTTGGCGCACTTGGGCA............................................................................................ | 19 | 0 | 1 | 4.00 | 4 | 2 | 2 | 0 | 0 | 0 |
| ..................................................CTTGGCGCACTTGGGCAGGA......................................................................................... | 20 | 0 | 2 | 2.50 | 5 | 4 | 0 | 0 | 0 | 1 |
| ................................................AACTTGGCGCACTTGGGCAG........................................................................................... | 20 | 0 | 1 | 2.00 | 2 | 0 | 2 | 0 | 0 | 0 |
| ................................................AACTTGGCGCACTTGGGCAGGA......................................................................................... | 22 | 0 | 1 | 2.00 | 2 | 0 | 0 | 2 | 0 | 0 |
| ..................................................CTTGGCGCACTTGGGCAGG.......................................................................................... | 19 | 0 | 2 | 2.00 | 4 | 2 | 2 | 0 | 0 | 0 |
| ................................................AACTTGGCGCACTTGGGCAGG.......................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................CTGCCCAAGTGCGCCAAGTACT.................................................. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................................AACGTGGCGCACTTGGGCAGT.......................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................CTGCACAAGTGGGCCAAGTACT.................................................. | 22 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................CTGCCCAAGTGCGCCAAGTAT................................................... | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................................AACTTGGCGCACATGGGCA............................................................................................ | 19 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................CTGCCCAAGTGCGCCACGTA.................................................... | 20 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................CTGCCCAAGTGCGCCAAGT..................................................... | 19 | 0 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| .................................................ACTTGGCGTACTTGGGCAGGAG........................................................................................ | 22 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..................................................CTTGGCGCGCTTGGGCAGGA......................................................................................... | 20 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| ....................................................TGGCGCACTTGGGCAGGA......................................................................................... | 18 | 0 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...................................................TTGGCGCACTTGGGCAGG.......................................................................................... | 18 | 0 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..................................................CTTGGCGCACTTGAGCAGGA......................................................................................... | 20 | 1 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...................................................TTGGCTCACTTGGGCAGGA......................................................................................... | 19 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 |
| .................................................ACTTGGCGCACTTGGGCAGG.......................................................................................... | 20 | 0 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 |
| ..................................................CGGGGCGCACTTGGGCAGGT......................................................................................... | 20 | 3 | 14 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..............................................AGAGCTTGACGCACTTGG............................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| ........................................CCCCGGCTAACTTCGCGC..................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 |
|
CTTTTGTAGCTTAGCGAGGACGTGTGGGTAATTGCTAGAGGGGGCCTCTTGAACCGCGTGAACCCGTCCTCCAGGTAGAGTATGGAGGACGGGTTCACGCGGTTCATGAGACCAAGTCCAGTCCGGTCTTAAGAGAAATGTGACTCCCTAAATGTGTTA
************************************((((...((((((.((((((((((((((((((((((((.........)))))))))))))))))))))))).))))))...))))...*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V050 head |
V057 head |
M042 female body |
|---|---|---|---|---|---|---|---|---|
| .....................................................................................AGGACGGGTTCACGCGGTTC...................................................... | 20 | 0 | 2 | 2.50 | 5 | 4 | 0 | 1 |
| ......................................................................................GGACGGGTTCACGCGGTTC...................................................... | 19 | 0 | 2 | 2.00 | 4 | 2 | 2 | 0 |
| .................................................TGAACCGCGTGAACCCGTC........................................................................................... | 19 | 0 | 2 | 0.50 | 1 | 0 | 1 | 0 |
| .....................................................................................AGGACGGGTTCACTCGGTT....................................................... | 19 | 1 | 2 | 0.50 | 1 | 0 | 0 | 1 |
| .....................................................................................AGGACGGGTTCGCGCGGTTC...................................................... | 20 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 |
| ......................................................................................GGACGGGTTCACGCGGTT....................................................... | 18 | 0 | 2 | 0.50 | 1 | 0 | 1 | 0 |
| ......................................................................................GGACGGGTTCACGCGGTTCA..................................................... | 20 | 0 | 2 | 0.50 | 1 | 0 | 0 | 1 |
| ....................................................................................GAGGACGGGTTCATGCGGTTCA..................................................... | 22 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 |
| .....................................................................................AGGACGGGTTCACGCGGT........................................................ | 18 | 0 | 2 | 0.50 | 1 | 1 | 0 | 0 |
| .....................................................................................AGGACGAGTTCACGCGGTTC...................................................... | 20 | 1 | 2 | 0.50 | 1 | 1 | 0 | 0 |
| .....................................................................................TGGACGGGTTCACGCGGGGC...................................................... | 20 | 3 | 14 | 0.07 | 1 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droPer2 | scaffold_26:100979-101137 + | dpe_128 | GA-AAACATCGAATCGCTCCTGCACACCCATTAACGATCTCCCCCGGAGAACTTGGCGCACTTGGGCAGGAGGTCCATCTCATACCTCCTGCCCAAGTGCGCCAAGTACTCTGGTTCAGGTCAGGCCAGAATTCTCTTTACACTGAGGGATTTACACAAT |
| dp5 | XL_group1a:494885-494952 + | GA-AAACATCGAATCGCTCCTGCACACCCATTAACGATCTCCCCCGGAGAACTTGGCGCACTTGCGAAG------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_12929:1713906-1713914 - | AA-AGACATC------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBip1 | scf7180000396543:179203-179254 - | AA-GAACACAGAAGAG-----------------ACATCCACCCCAGGGGAATACGATGATCTGGAGCAAG------------------------------------------------------------------------------------------ | |
| droEle1 | scf7180000491011:1091596-1091605 + | AG-AGACATCG----------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000779506:879254-879263 + | AA-AGACATCG----------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000301760:1057028-1057037 + | AA-AGACATCG----------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415179:44316-44327 + | GAAAGACATCGA---------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409277:421554-421563 + | AG-AGACATCG----------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droPer2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/15/2015 at 02:32 PM