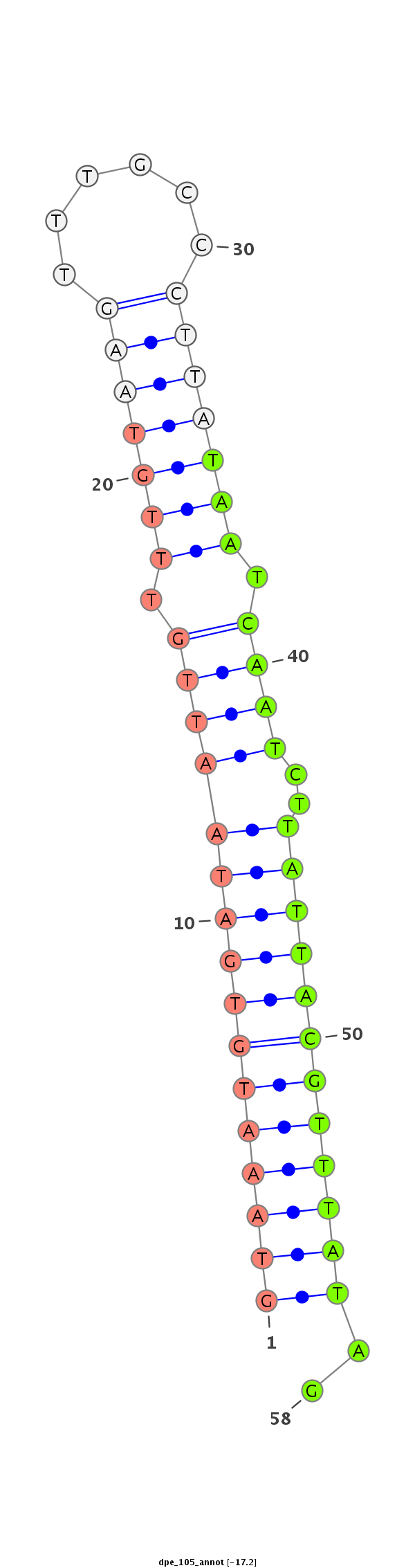
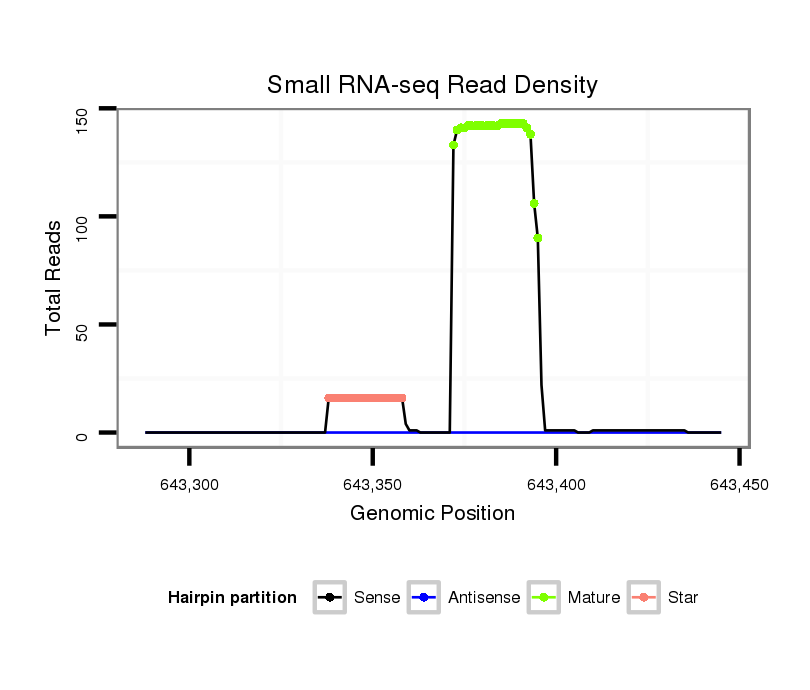
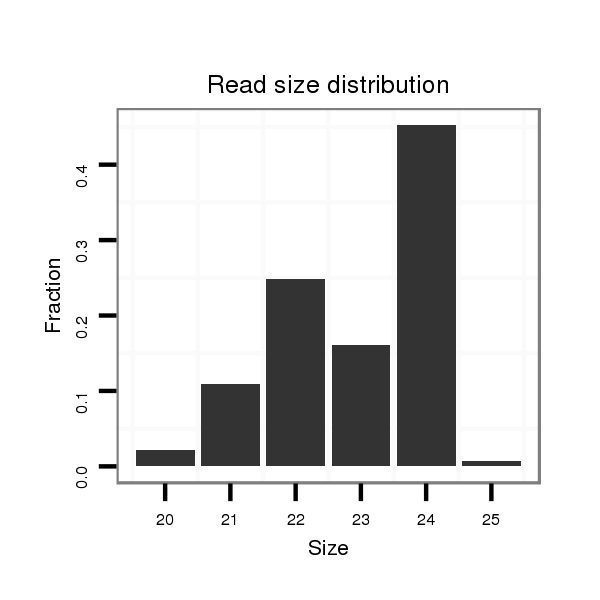
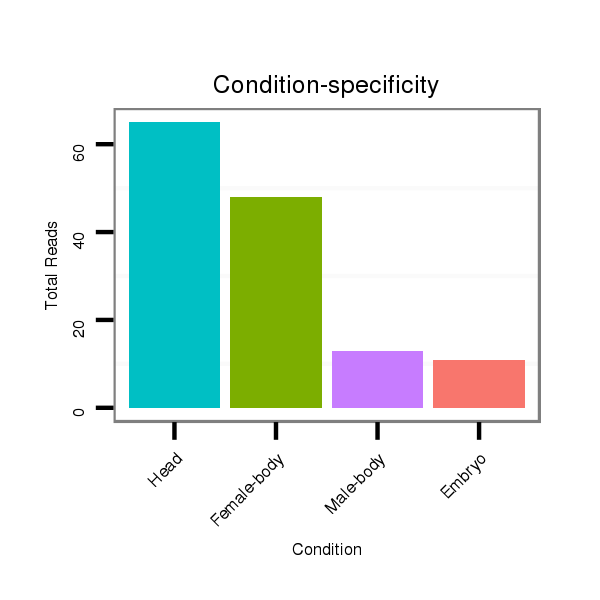
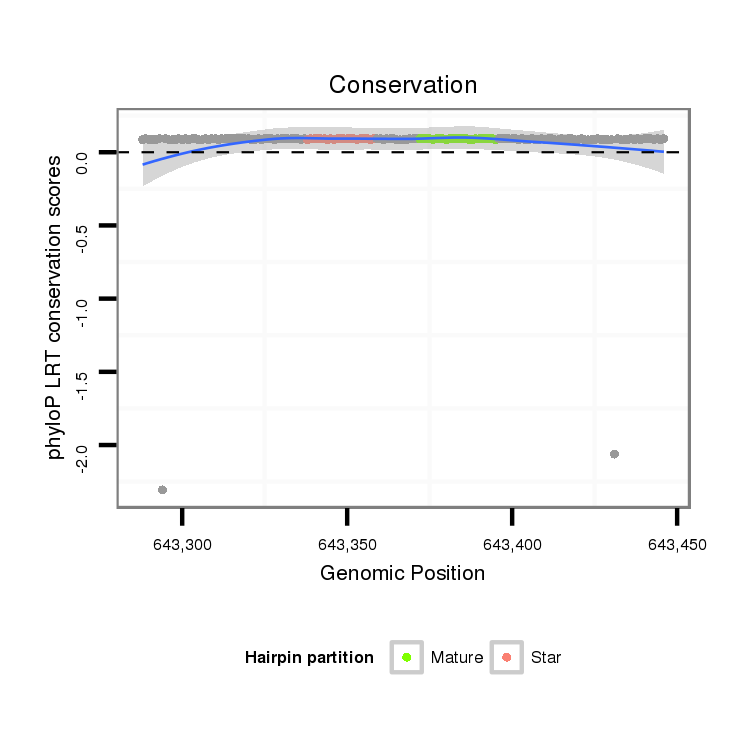
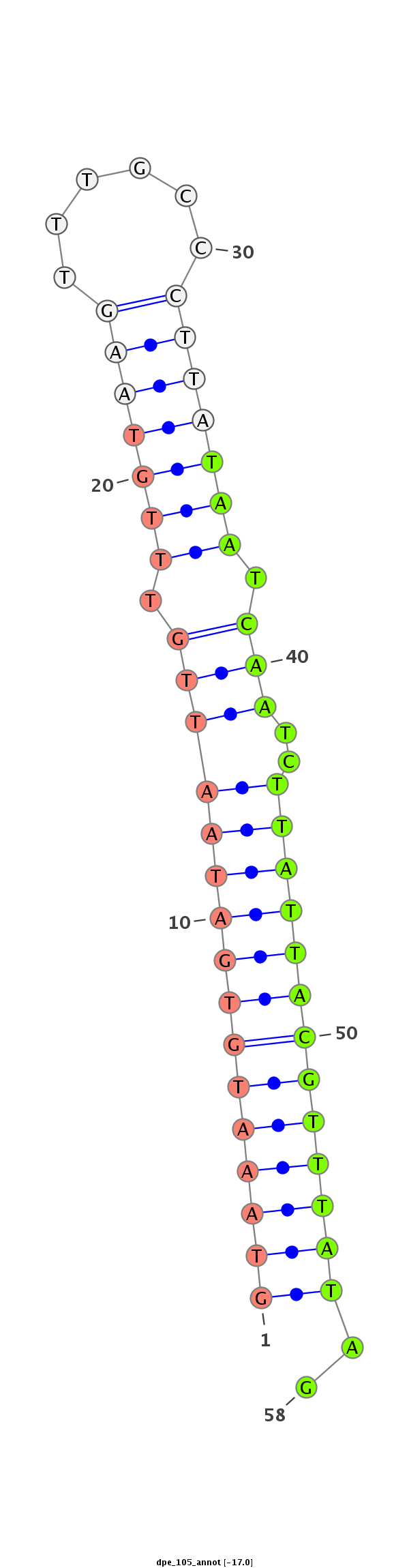
ID:dpe_105 |
Coordinate:scaffold_10:643338-643395 + |
Confidence:Known Ortholog |
Class:Mirtron |
Genomic Locale:intergenic |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -17.0 | -16.2 | -16.2 |
 |
 |
 |
intergenic
No Repeatable elements found
|
CTGCCACAGCGACAGAAGCAGAAGTTTATAAAACCAGAAACAAATTTTGTGTAAATGTGATAATTGTTTGTAAGTTTGCCCTTATAATCAATCTTATTACGTTTATAGTATAATGAGCTGGTCTAGCTCATCGTCGCCTGCCGGTGCCTCTGATTCTC
**************************************************((((((((((((((((.(((((((......))))))).))))..))))))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M042 female body |
V050 head |
V057 head |
V111 male body |
V042 embryo |
M021 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................TAATCAATCTTATTACGTTTATAG.................................................. | 24 | 0 | 1 | 62.00 | 62 | 23 | 17 | 8 | 6 | 8 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTTAT.................................................... | 22 | 0 | 1 | 31.00 | 31 | 9 | 10 | 10 | 1 | 1 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTTATAGT................................................. | 25 | 0 | 1 | 19.00 | 19 | 5 | 5 | 6 | 0 | 0 | 3 |
| ....................................................................................TAATCAATCTTATTACGTTTATA................................................... | 23 | 0 | 1 | 16.00 | 16 | 7 | 4 | 4 | 0 | 1 | 0 |
| ..................................................GTAAATGTGATAATTGTTTGT....................................................................................... | 21 | 0 | 1 | 12.00 | 12 | 0 | 5 | 1 | 6 | 0 | 0 |
| .....................................................................................AATCAATCTTATTACGTTTATAG.................................................. | 23 | 0 | 1 | 6.00 | 6 | 5 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTTATAT.................................................. | 24 | 1 | 1 | 4.00 | 4 | 1 | 0 | 3 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTTA..................................................... | 21 | 0 | 1 | 3.00 | 3 | 1 | 0 | 2 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTTATAGA................................................. | 25 | 1 | 1 | 3.00 | 3 | 2 | 1 | 0 | 0 | 0 | 0 |
| ..................................................GTAAATGTGATAATTGTTTGTA...................................................................................... | 22 | 0 | 1 | 3.00 | 3 | 1 | 1 | 1 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTT...................................................... | 20 | 0 | 1 | 2.00 | 2 | 0 | 1 | 0 | 0 | 1 | 0 |
| ....................................................................................TAATCAATGTTATTACGTTTAT.................................................... | 22 | 1 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTTGTAG.................................................. | 24 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................................TAATACATCTTATTACGTTTAT.................................................... | 22 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................TACTCGATTTTATTACGTTTATAGT................................................. | 25 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTCCGTTTAT.................................................... | 22 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..................................................GTAAATGTCATAATTGTTTGTAAGTT.................................................................................. | 26 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................AATCCATCTTATTACGTTTATAG.................................................. | 23 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................TAATCAATGTTATTACGTTTATAA.................................................. | 24 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACGATTAT.................................................... | 22 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................CTAGCTCATCGTCGCCTGCCGGTGCC.......... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................................TAATCAGTCTTATTACGTTTATA................................................... | 23 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACTTTT...................................................... | 20 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................TAATAAATCTTATTACGTTT...................................................... | 20 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..................................................GTAAATGTGATAATTGTTTGTAAGT................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTTATCGT................................................. | 25 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................................TAATCGATCTTATTCCGTTTAT.................................................... | 22 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTCATAGT................................................. | 25 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................................TAATCAATCTTATTACGTTTATAA.................................................. | 24 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................AATCAATCTTATTACGTTTATAGT................................................. | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................TACGTTTATAGTATAATGAGC........................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................TAATCAATATTATTACGTTTATAG.................................................. | 24 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................TAATCAATATTATTACGTCTAT.................................................... | 22 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................ATCAATCTTATTACGTTTAT.................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................GTAAATGTGATAATTGTGTGT....................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................CAATCTTATTACGTTTATAGT................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................TAATCAATCTTATTACGTTTATAGTAA............................................... | 27 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................TCATCCATCTTATTACGTTTATAG.................................................. | 24 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................TAATCAAGCTTACTACGTTTATAC.................................................. | 24 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................GGTAATTTTACCCTTATAATC..................................................................... | 21 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
|
GACGGTGTCGCTGTCTTCGTCTTCAAATATTTTGGTCTTTGTTTAAAACACATTTACACTATTAACAAACATTCAAACGGGAATATTAGTTAGAATAATGCAAATATCATATTACTCGACCAGATCGAGTAGCAGCGGACGGCCACGGAGACTAAGAG
**************************************************((((((((((((((((.(((((((......))))))).))))..))))))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total |
|---|
| Species | Coordinate | ID | Type | Alignment |
|---|---|---|---|---|
| droPer2 | scaffold_10:643288-643446 + | dpe_105 | confident | CTGCCACAGCGACAGAAGCAGAAGTTTATAAAACCAGAAACAAATTTTGTGTAAATGTGATAATTGTTTGTAAGTTTGCCCTTATAATCAATCTTATTACGTTTATAGTATAATGAGCTGGTCTAGCTCATCGTCGCCTGCCGGTGCCTCTGATTCTCA |
| dp5 | 4_group4:1639545-1639703 + | dps-mir-2564 | Known_mirbase | CTGCCAGAGCGACAGAAGCAGAAGTTTATAAAACCAGAAACAAATTTTGTGTAAATGTGATAATTGTTTGTAAGTTTGCCCTTATAATCAATCTTATTACGTTTATAGTATAATGAGCTGGTCTAGCTCATCGTCGCCTGCCGTTGCCTCTGATTCTCA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
Generated: 10/20/2015 at 07:04 PM