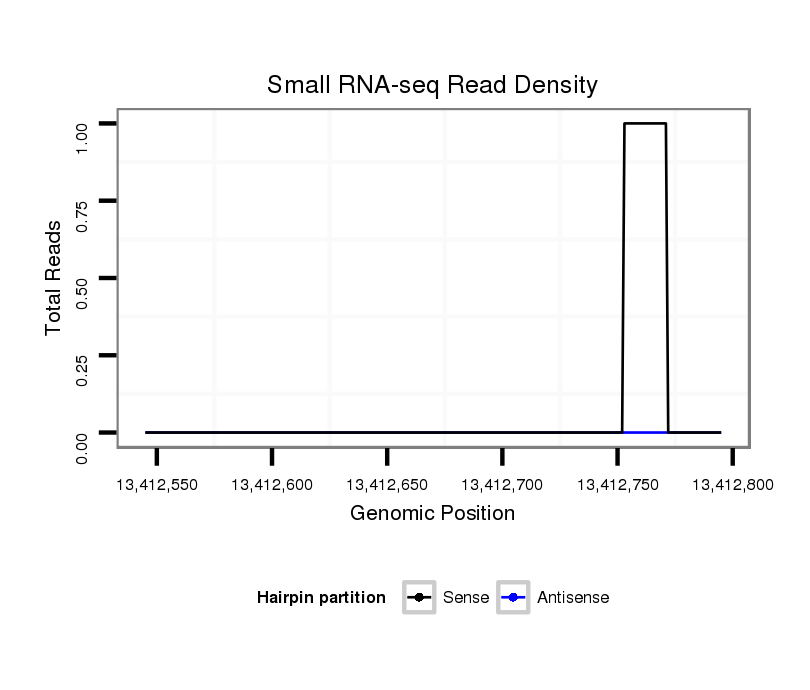
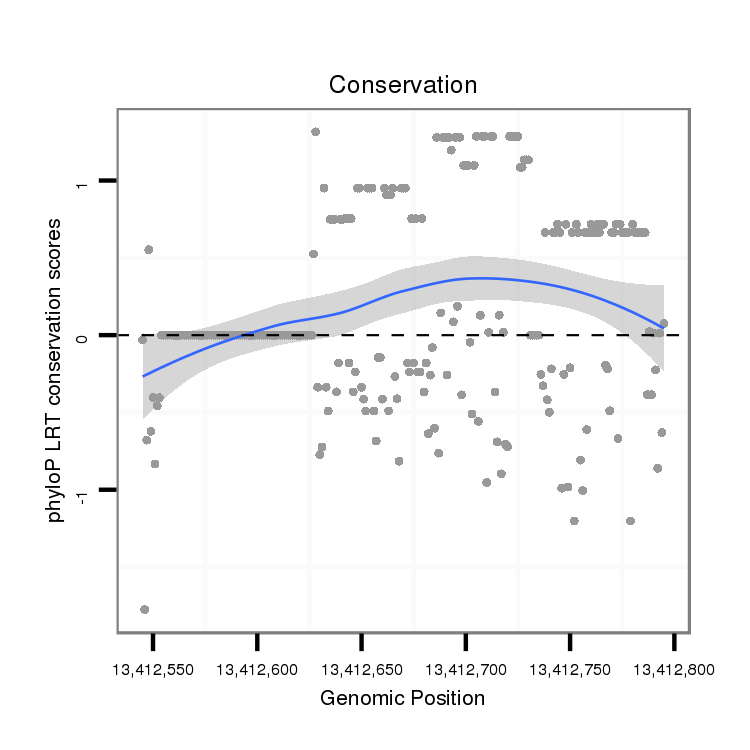
ID:dmo_772 |
Coordinate:scaffold_6473:13412595-13412745 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dmoj_GLEANR_16592:2]; CDS [Dmoj\GI15889-cds]; intron [Dmoj\GI15889-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CGATAGCTCTGAGCTATGCCGAATAACGGTAGTAGTCGATAATCTAGTGCTTACAGCTTAACCACCATTTCCGAAATGTATTCTCCTTCTGCCTGCCAATATATTCATTAAACATTTCACACTGTTAGTTGATAACTGTACATCTTCTGATTAATTTGATTTCAAATTTCCATTCGAATTTGCATTCGAATCACACTGCAGTCTAACATCCGCCGAGCTGTTGTCCGTTTCGCCAACGCCGCTCACCTCGG **************************************************...(((................(((((((................................))))))).....(((((..(((........)))..))))).((((((....))))))...((((((((....))))))))....)))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M046 female body |
V110 male body |
M060 embryo |
|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................ATCTTCTGATTAATTTTCTTAC........................................................................................ | 22 | 3 | 4 | 1.50 | 6 | 6 | 0 | 0 |
| .............................................................................................................................................ATCTTCTGATTAATTTTCTT.......................................................................................... | 20 | 2 | 8 | 1.38 | 11 | 10 | 1 | 0 |
| ................................................................................................................................................................................................................TCCGCCGAGCTGTTGTCCG........................ | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 |
| .................................................CTTACAGGTTAACCAGCGTTT..................................................................................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 |
| ................................................................................................................................................................................AATTTTCATTCGAATCACAC....................................................... | 20 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 |
| .........................................................................................................................................GAATATCTTCTGATTAATTT.............................................................................................. | 20 | 2 | 6 | 0.17 | 1 | 1 | 0 | 0 |
| ....................................CGACGATCTATTGCTTACA.................................................................................................................................................................................................... | 19 | 3 | 13 | 0.08 | 1 | 1 | 0 | 0 |
| .............................................................................................................................................ATCTTCTGATTAATTTTCT........................................................................................... | 19 | 2 | 14 | 0.07 | 1 | 1 | 0 | 0 |
| ...................................................TACAGGTTAACCAGCGTTT..................................................................................................................................................................................... | 19 | 3 | 14 | 0.07 | 1 | 0 | 1 | 0 |
|
GCTATCGAGACTCGATACGGCTTATTGCCATCATCAGCTATTAGATCACGAATGTCGAATTGGTGGTAAAGGCTTTACATAAGAGGAAGACGGACGGTTATATAAGTAATTTGTAAAGTGTGACAATCAACTATTGACATGTAGAAGACTAATTAAACTAAAGTTTAAAGGTAAGCTTAAACGTAAGCTTAGTGTGACGTCAGATTGTAGGCGGCTCGACAACAGGCAAAGCGGTTGCGGCGAGTGGAGCC
**************************************************...(((................(((((((................................))))))).....(((((..(((........)))..))))).((((((....))))))...((((((((....))))))))....)))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V110 male body |
M046 female body |
V056 head |
V049 head |
|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................TAGAAGACTAATTAAAAGAA.......................................................................................... | 20 | 2 | 8 | 11.50 | 92 | 40 | 51 | 0 | 1 |
| .............................................................................................................................................TAGAAGACTAATTAAAAGAATG........................................................................................ | 22 | 3 | 4 | 3.00 | 12 | 9 | 3 | 0 | 0 |
| .............................................................................................................................................TAGAAGACTAATTAAAAGA........................................................................................... | 19 | 2 | 14 | 1.50 | 21 | 9 | 12 | 0 | 0 |
| ..........................................................................................................................................TTATAGAAGACTAATTAAA.............................................................................................. | 19 | 2 | 20 | 1.10 | 22 | 16 | 5 | 1 | 0 |
| ......................................................................................................................................TGACTGGTAGAAGGCTAATTA................................................................................................ | 21 | 3 | 2 | 1.00 | 2 | 2 | 0 | 0 | 0 |
| .........................................................................................................................................CTTATAGAAGACTAATTAAA.............................................................................................. | 20 | 2 | 6 | 0.50 | 3 | 1 | 2 | 0 | 0 |
| .......................................................................................................................................................................GAGGTATGCTTAAACGTAAGGT.............................................................. | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 |
| .............................................................................................................................................TAGAAGACTAATTAAAAG............................................................................................ | 18 | 2 | 20 | 0.20 | 4 | 0 | 4 | 0 | 0 |
| ........................................................................................................................................................................AGGTAAGCTGAAATGTAAGGT.............................................................. | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 |
| .......................................................................................................................................................................TAGATAAGATTAAACGTAAGCT.............................................................. | 22 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 |
| ................ACCTCTTATTCCCATCATC........................................................................................................................................................................................................................ | 19 | 3 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 |
| .....................................................................................................................................................................................GGTTAGCTTGGTGTGACGTC.................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 |
| .............................................................................................................................................TGGAAGACTAATTAAAAGAA.......................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droMoj3 | scaffold_6473:13412545-13412795 + | dmo_772 | CGATAGCTCTGAGCTATGCCGAATAACGGTAGTAGTCGATAATCTAGTGCTTACAGCTTAACCACCATTTCCGAAATGTATTCTCCTTCTGCCTGCCAATATATTCATTAAACATTTCACACTGTTAGTTGATAACT----GTACAT-CTTCTGATTAATTTGATTTCAAATTTCCATTCGAATTTGCATTCGAATCACACTGCAGTCTAACATCCGCCGAGCTGTTGTCCGTTTCGCCAACGCCGCTCACCTCGG |
| droVir3 | scaffold_12928:1611045-1611138 - | CTT--------------------------------------------------------------------------------TTGATTA-------------TTTGATAATTGCCTCTCATATTTA---------------------------------------------------------------------TCCTACGCAGATTGACGTCGTCGGAGCTGTTGTCCGTGTCGCCGACGCCGCCAGCCACGG | |
| droGri2 | scaffold_15203:8168122-8168210 + | CATTCAGAT-------------------------------------------------------------------------CT---------------------------------------------------------TCAAAGTTTTTTGATTAT-------------------------------------TCCAACGCAGCTTTGCCTCCACGGAGCTGTTAAGCGTTTCGCCCACGCCGCCAGCTGCAG | |
| droWil2 | scf2_1100000004768:2568699-2568760 + | ACT------------------------------------------------------------------------------------------------------------------------------------------ATCCATATTTTTGCTTATTTTATTTACAAATTTTTAGTTTAATTTGCATT--------------------------------------------------------TAAATAACA | |
| droRho1 | scf7180000776895:24481-24586 + | TTC--------------------------------------------------------------------------------TTGCTTTGCCGACCAACAGTTTTATTAAAAGCTTCACACAATTAAATAAATAACACTCACCTAAATTTCTGATAAATTTAGTTATAACATTTCACAAAAATTT---------------------------------------------------------------------- | |
| droYak3 | 2L:21239248-21239277 + | TGA--------------------------------------------------------------------------------T---------------------------------------------------------------------------------TTTAAGTTTCAGTTTGAATTTGCATT----------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/17/2015 at 11:00 PM