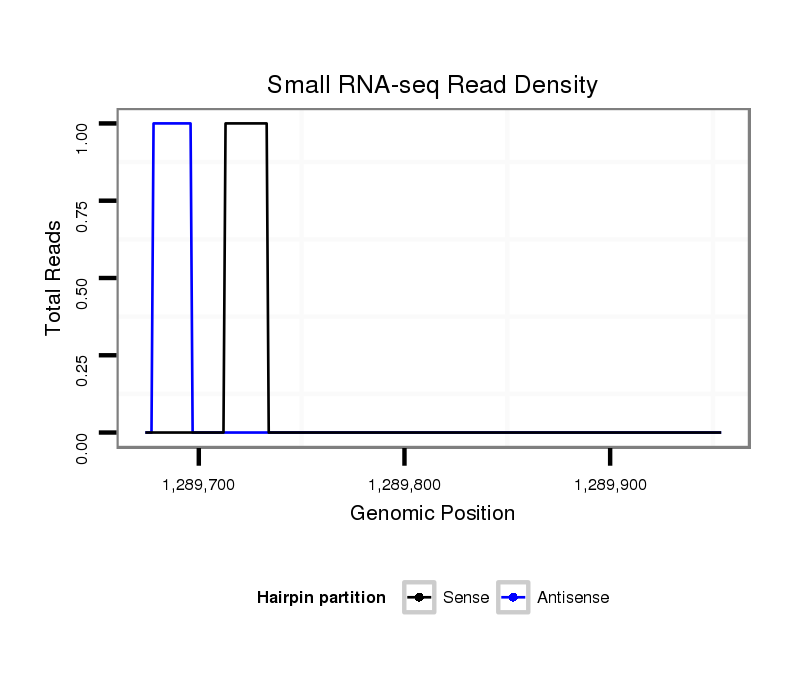
ID:dmo_488 |
Coordinate:scaffold_6328:1289724-1289904 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dmoj\GI16343-cds]; exon [dmoj_GLEANR_17065:1]; exon [dmoj_GLEANR_17065:2]; CDS [Dmoj\GI16343-cds]; intron [Dmoj\GI16343-in]
No Repeatable elements found
| ##################################################-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CCGGAGAAGTACCACAAGCTCAACAAGCTCATAGAGCTGCTGGAGAGCAAGTAAGCCTAGACCACATAGCTTAAAAACTTGTTGAGAATTTTAATATACTTTACACTATTATATCTTAATGTAAATATTTATGGGCTAAATGGGATAAATTATAATCTTCACATATTAGAAATTAGAAACTGATTCGCATAAATTGTATAATTGGCTAATGAAAACGCTTTGTGCTTTTAGTATACCACCTGAGGACTTCTATCGGCGTTATCCGCTGGCCAAAATTGAGG **************************************************((((...((..(((..(((((((.(((..((((((((....(((((..........)))))..))))))))......))).)))))))..)))..))..))))......((((........(((((...((((((.(((.....)))..)))))))))))..........)))).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M046 female body |
V110 male body |
|---|---|---|---|---|---|---|---|
| ...............................................................................................................................................................................GAAACTGATTCGCCTAAAC....................................................................................... | 19 | 2 | 2 | 7.00 | 14 | 14 | 0 |
| ............................................................................................................................................................................TCTGAAACTGATTCGCCTAAA........................................................................................ | 21 | 3 | 4 | 1.25 | 5 | 5 | 0 |
| .............................................................................................................................................................................CTGAAACTGATTCGCCTAAA........................................................................................ | 20 | 3 | 13 | 1.08 | 14 | 13 | 1 |
| ...........................................................................................................................................................................ATCTGAAACTGATTCGCCTAAA........................................................................................ | 22 | 3 | 3 | 1.00 | 3 | 3 | 0 |
| .......................................CTGGAGAGCAAGTAAGCCTAG............................................................................................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 |
| ..........................................................................................................................................................................AATCTGAAACTGATTCGCCTAAA........................................................................................ | 23 | 3 | 2 | 0.50 | 1 | 1 | 0 |
| ..............................................................................................................................................................................TGAAACTGATTCGCCTAAA........................................................................................ | 19 | 2 | 2 | 0.50 | 1 | 1 | 0 |
| ............................................................................................................................................................................................CTAAATTGTATAATTGGG........................................................................... | 18 | 2 | 9 | 0.44 | 4 | 4 | 0 |
| ................................................................................................................................................................................................ATTGTATAATTGGATATTGA..................................................................... | 20 | 2 | 3 | 0.33 | 1 | 0 | 1 |
| ................................................................................................................................................................................AAACTGATTCGCCTAAAC....................................................................................... | 18 | 2 | 7 | 0.29 | 2 | 2 | 0 |
|
GGCCTCTTCATGGTGTTCGAGTTGTTCGAGTATCTCGACGACCTCTCGTTCATTCGGATCTGGTGTATCGAATTTTTGAACAACTCTTAAAATTATATGAAATGTGATAATATAGAATTACATTTATAAATACCCGATTTACCCTATTTAATATTAGAAGTGTATAATCTTTAATCTTTGACTAAGCGTATTTAACATATTAACCGATTACTTTTGCGAAACACGAAAATCATATGGTGGACTCCTGAAGATAGCCGCAATAGGCGACCGGTTTTAACTCC
**************************************************((((...((..(((..(((((((.(((..((((((((....(((((..........)))))..))))))))......))).)))))))..)))..))..))))......((((........(((((...((((((.(((.....)))..)))))))))))..........)))).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V110 male body |
M046 female body |
V056 head |
|---|---|---|---|---|---|---|---|---|
| ............................................................................................................................................................................AGACTTTGACTAAGCGGATTT........................................................................................ | 21 | 3 | 4 | 6.75 | 27 | 17 | 9 | 1 |
| ....TCTTCATGGTGTTCGAGTT.................................................................................................................................................................................................................................................................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 |
| ..........................................................................................................................................................................TTAGACTTTGACTAAGCGGATTT........................................................................................ | 23 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 |
| ...........................................................................................................................................................................TAGACTTTGACTAAGCGGATTT........................................................................................ | 22 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 |
| ............................................................................................................................................................................AGGCTTTGACTAAGCGGATT......................................................................................... | 20 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 |
| .............................................................................................................................................................................GACTTTGACTAAGCGGATTT........................................................................................ | 20 | 3 | 13 | 0.08 | 1 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droMoj3 | scaffold_6328:1289674-1289954 + | dmo_488 | CCGGAGAAGTACCACAAGCTCAACAAGCTCATAGAGCTGCTGGAGAGCAAGTAAGCCTA-GACCACATAG-----------------------------------CTTAAAAACTTGTTGAGAATTTTAATATACTTTACACTATTATATCTTAATGTAAATATTTATGGGCTAAATGGGATAAATTATAATCTTCACATATTAGAAATTAGAAACTGATTCGCATAAATTGTATAATTGGCTAATGAAAACGCTTTGTGCTTTTAGTATACCACCTGAGGACTTCTATCGGCGTTATCCGCTGGCC---------AAAATTGAGG |
| droVir3 | scaffold_13042:2271984-2272042 - | CAGGAGCAATATCGCAAGCTCAACATGCTCGTCGAGCTGCTGGAAAGCGAGTAAGTCCA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15203:4871759-4871937 + | TCAGTGCAATATCGCCATCTGGAAATTTTCTTGCGGCTGCTGGAGAGCGAGTAAGTTATCGATAATATATAATCGATAACTGATGTAATAATTCCATTAATCAAATTTTGAAA---------------------------------------------------------------------------------------------------------------------------------------------------TTTGTAGTATGGCTCCATCGGAATTCTTTCGCCGATTGCCCGGGGATAAGATTGATAAAATTGAGG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/17/2015 at 10:25 PM