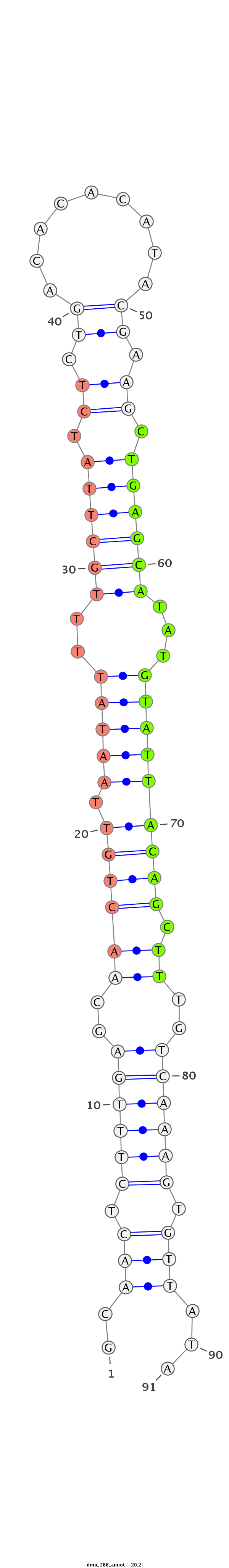
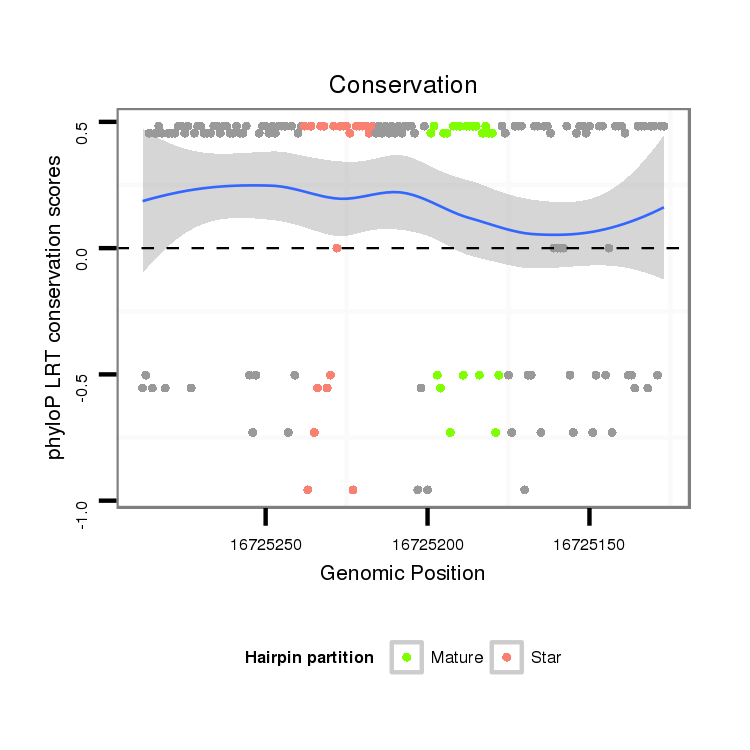
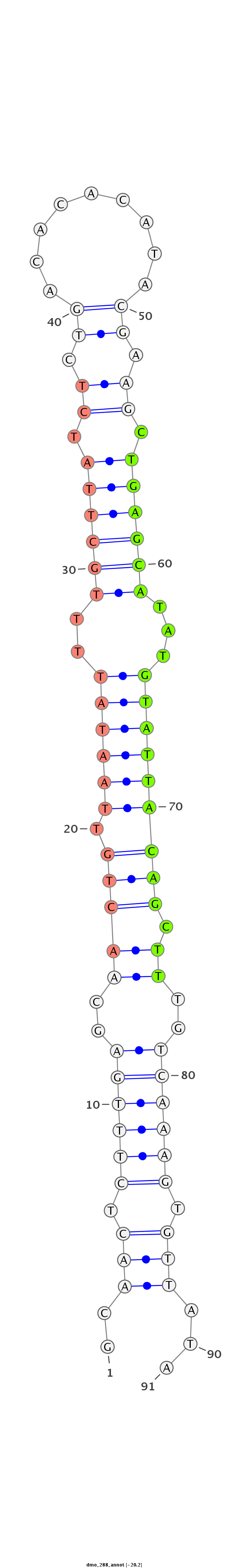
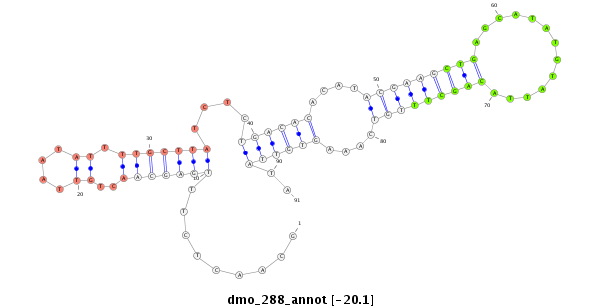
ID:dmo_288 |
Coordinate:scaffold_6680:16725177-16725238 - |
Confidence:candidate |
Class:Canonical miRNA |
Genomic Locale:intron |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.2 | -20.1 | -20.0 |
 |
 |
 |
intron [Dmoj\GI11698-in]
No Repeatable elements found
|
AGCACTCAGCGAACTTGTTCGCATGAACCAGCTACGCAACTCTTTGAGCAACTGTTAATATTTTGCTTATCTCTGACACACATACGAAGCTGAGCATATGTATTACAGCTTTGTCAAAGTGTTATACACACAGAGTAGAGGTTGGGTTAGTGTTATAATAAA
***********************************..(((.((((((..((((((.(((((..((((((.((.((.........)).)).))))))...))))))))).))..)))))).)))...************************************ |
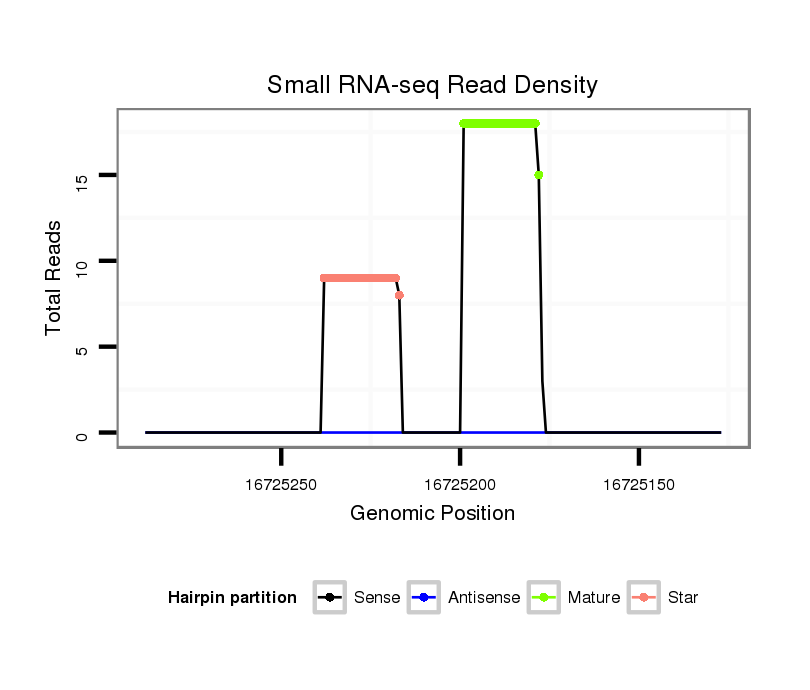
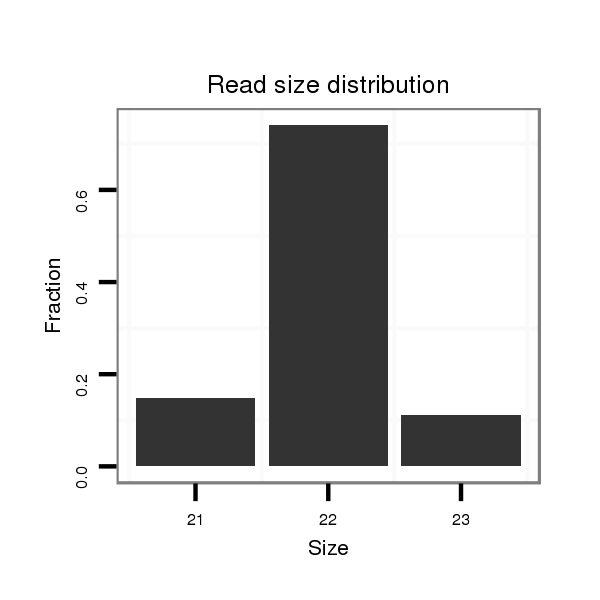
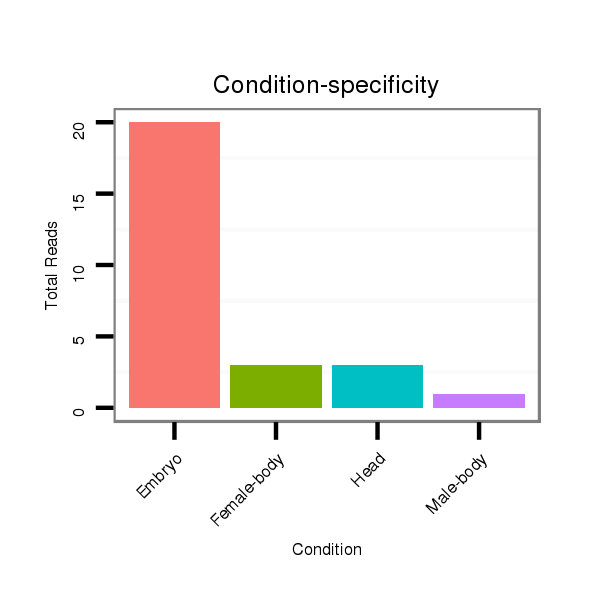
Read size | # Mismatch | Hit Count | Total Norm | Total | M060 embryo |
M046 female body |
V049 head |
V041 embryo |
V110 male body |
V056 head |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................CTGAGCATATGTATTACAGCTT................................................... | 22 | 0 | 1 | 12.00 | 12 | 7 | 1 | 3 | 0 | 1 | 0 |
| ..................................................ACTGTTAATATTTTGCTTATCT.......................................................................................... | 22 | 0 | 1 | 8.00 | 8 | 8 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................CTGAGCATATGTATTACAGCTTT.................................................. | 23 | 0 | 1 | 3.00 | 3 | 2 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................CTGAGCATATGTATTACAGCT.................................................... | 21 | 0 | 1 | 3.00 | 3 | 2 | 0 | 0 | 1 | 0 | 0 |
| ..................................................ACTGTTAATATTTTGCTTATCC.......................................................................................... | 22 | 1 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..................................................ACTGTTAATATTTTGCTTATC........................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................CGGAGCATATGTATTACAGCTT................................................... | 22 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................CTGAGCATATGTCTTACAGCTT................................................... | 22 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................CGGAGCATATGTATTACAGCTTT.................................................. | 23 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................CGGTGCATATGTATTACAGCTT................................................... | 22 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............CTTGTGCGCGTGAACCAGCTT................................................................................................................................ | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| .............CTTGTCCCCGTGAACCAGCT................................................................................................................................. | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| .............CTGGTCCGCTTGAACCAGCT................................................................................................................................. | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TCGTGAGTCGCTTGAACAAGCGTACTTGGTCGATGCGTTGAGAAACTCGTTGACAATTATAAAACGAATAGAGACTGTGTGTATGCTTCGACTCGTATACATAATGTCGAAACAGTTTCACAATATGTGTGTCTCATCTCCAACCCAATCACAATATTATTT
************************************..(((.((((((..((((((.(((((..((((((.((.((.........)).)).))))))...))))))))).))..)))))).)))...*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total |
|---|
| Species | Coordinate | ID | Type | Alignment |
|---|---|---|---|---|
| droMoj3 | scaffold_6680:16725127-16725288 - | dmo_288 | candidate | AGCACTC--AGCGAACTTGTTCGCAT---------------------GAACCAGCTACGCAACTCTTTGAGCAACTGTTAATATTTTGCTTATCT-----------------CTGACACACATACG----AAGCTGAGCATAT----GTATTACAGCTTTGTCAAAGTGTTATACACACAGAGTAGAGGTTGGGTTAGTGTTATAATAAA |
| droVir3 | scaffold_13049:22129255-22129458 - | TACTCTCTATGCGAACTAGTTCGCATACACTTATCTGTAAATACGAAGAACCAGCTGAACAACTCTTTTAACAAGTTATATCA-TTTGGTTATCTGCATATTTATGCACACACTGACACACATACCCATATACCTATGCCTATATCTATATTGCAGCGCTGCAAAACCATTCTAC----AACGTAGATATTA-TTTAGCAATATTATGAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
Generated: 10/20/2015 at 07:02 PM