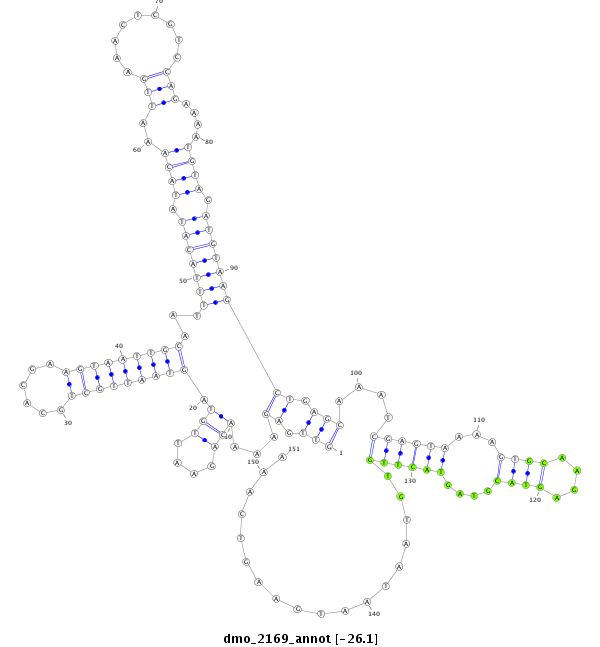



ID:dmo_2169 |
Coordinate:scaffold_6540:33763339-33763489 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
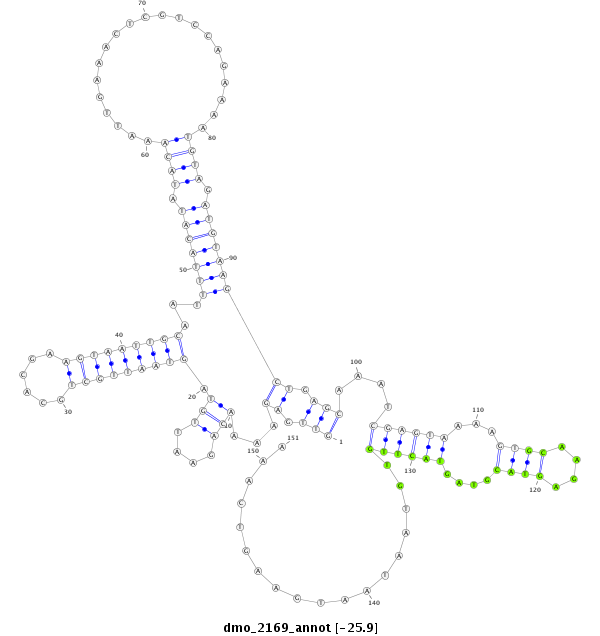
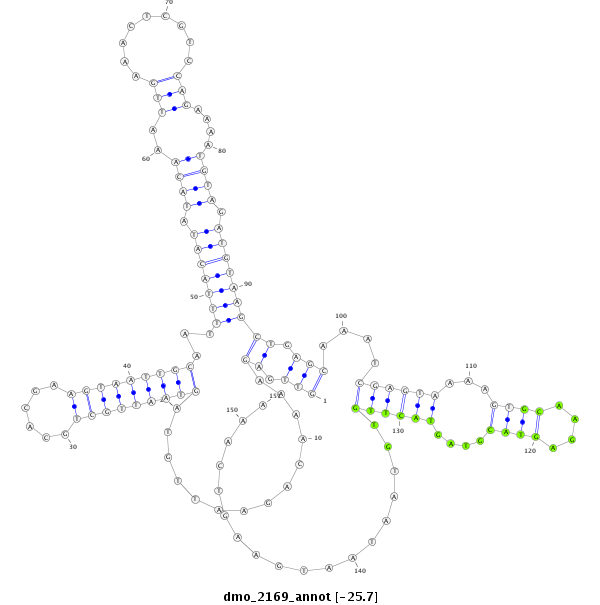
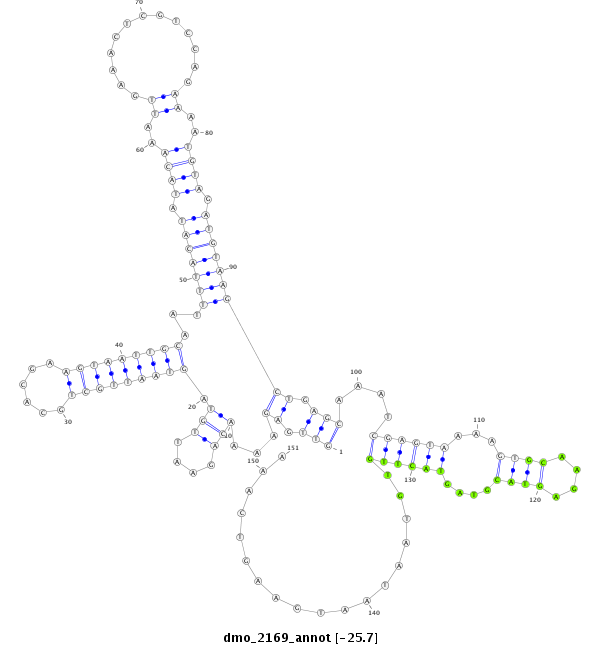
| -25.9 | -25.7 | -25.7 |
 |
 |
 |
CDS [Dmoj\GI21947-cds]; exon [dmoj_GLEANR_7205:1]; intron [Dmoj\GI21947-in]
No Repeatable elements found
| -#################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AATGGGTGGCAGCGCTGCCAAACTATGTGGCAACGCCAAGATTCGCCGCAGTTGAGAAAACAGAATTGTAGTAATTGCTGCACGAAGTAATTGCAATTTTACATATACAAATTGAAACTCGTCCAGAAAATGTAGATGTAAGCTGAGCAAATCGAGTAAAAGTGCAAGAGTACGTAGTACTTGTGTAATAATGAAGTCAAACGAAATATATGTATGTGTGGTATCTCTGTGTTTGTGTTGTTTGTAGAATA **************************************************(((.((...(((....))).(((((((((......)))))))))...(((((((.((((..(((.........)))....)))).))))))))).)))....((((((...((((....))))....))))))..................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V110 male body |
M060 embryo |
V041 embryo |
M046 female body |
V056 head |
V049 head |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................................................................................................................................................GAGGTATCTCTGTGTACGT............... | 19 | 3 | 20 | 1.25 | 25 | 21 | 0 | 0 | 3 | 0 | 1 |
| .........................................................................................................................................................................................................................GAGGTATCTCTGTGTACGTGT............. | 21 | 3 | 17 | 1.06 | 18 | 9 | 0 | 0 | 9 | 0 | 0 |
| ...................................................................................................................................................................GCAAGAGTACGTAGTACTTGTG.................................................................. | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................CTCGTCCAGAAAATGTAGATGTAAGCCG.......................................................................................................... | 28 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................GCAAATCGCGTAAAAGGGCA..................................................................................... | 20 | 2 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................GGTGTAATTGTGAAGTCAA................................................... | 19 | 3 | 20 | 0.10 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| .................................................AACTGAGAAAACAGAATTG....................................................................................................................................................................................... | 19 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................ATGGAGCAACGCCAAGAGTC............................................................................................................................................................................................................... | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................................................................................................................................................................GTTTGTGGTATCGCTGTGTT.................. | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
|
TTACCCACCGTCGCGACGGTTTGATACACCGTTGCGGTTCTAAGCGGCGTCAACTCTTTTGTCTTAACATCATTAACGACGTGCTTCATTAACGTTAAAATGTATATGTTTAACTTTGAGCAGGTCTTTTACATCTACATTCGACTCGTTTAGCTCATTTTCACGTTCTCATGCATCATGAACACATTATTACTTCAGTTTGCTTTATATACATACACACCATAGAGACACAAACACAACAAACATCTTAT
**************************************************(((.((...(((....))).(((((((((......)))))))))...(((((((.((((..(((.........)))....)))).))))))))).)))....((((((...((((....))))....))))))..................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M046 female body |
V056 head |
|---|---|---|---|---|---|---|---|
| .......................................CTAATCGGCGTCAACCATTTTG.............................................................................................................................................................................................. | 22 | 3 | 3 | 0.67 | 2 | 2 | 0 |
| .....................TGATACACCGATGCGATCCT.................................................................................................................................................................................................................. | 20 | 3 | 7 | 0.14 | 1 | 0 | 1 |
| ..........................................ATCGGCGTCAACCATTTTG.............................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droMoj3 | scaffold_6540:33763289-33763539 - | dmo_2169 | AATGGGTGGCAGCGCTGCCAAACTATGTGGCAACGCCAAGATTCGCCGCAGTTGAGAAAA-CAGAATTGTAGTAATTGCTGCACGAAGTAATTGCAATTTTA-----------CATATACAA--ATTGAAACTCGTCCAGA--AAAT--GTAGATGTAAGCTGAG-----------------------------------------CAAATCGAGTAAAAGTGCAAGA---GTACGT----AGTA----CTTGTGTAATAATGAAGTCAAACGAAATATATGTATGTGT---GGTATCT---CTGTGTTTGTGTTGTTTGTAGAATA--------- |
| droVir3 | scaffold_12822:1122346-1122609 - | AGCGTGTGGCA---ACGCCAAAC-ATCTGGCAACGCCAGAAGTCGCCGGAGTTGAGAAAA-CAAAATTTCAGCAAATGCTGTAAGAAATTACTGAATAATTGGTTAA----AACAAATAAAA--TGTGAATAGCGTACAGT--AAATACCTTGCTGTGGCCTCTA-----------------------------------------TAATGCAGGCGAGAGTGTAATT---GTGGGAAAGAAGCTGTTGC-----------TGATGT-GAGCATAATTGATGTATGTACTAATGTACATATATTGTGTTTGT---GTTTGTAAAAGAGAATAAAAA | |
| droGri2 | scaffold_15074:2704717-2704989 + | ATCGTCTGCTGGTAATCGCAGTC-ATCTGGCAACGCCAAAAGTCGCCGGAGTTGAAAAAAAAAGAATTTGTGCCATTTGTGTAAGAATTTATTATTAATTTGATTAACTAAAGCTATT-GAATTTGTTAAACGAGTACGAATAAAGT-------------GCCTGTGAAACTTGTTAAAGTCCAGCAGAGCATTTGTGGCTCGCTTTTAAGCCGGCGTTA-----ATTGTTGTGCG--ACAAGTTG---------TAATATTTATGTTGAG--------------------ACATATGTAAATTTTATTTGT---GTTTGTAAAATA--------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/18/2015 at 02:02 AM