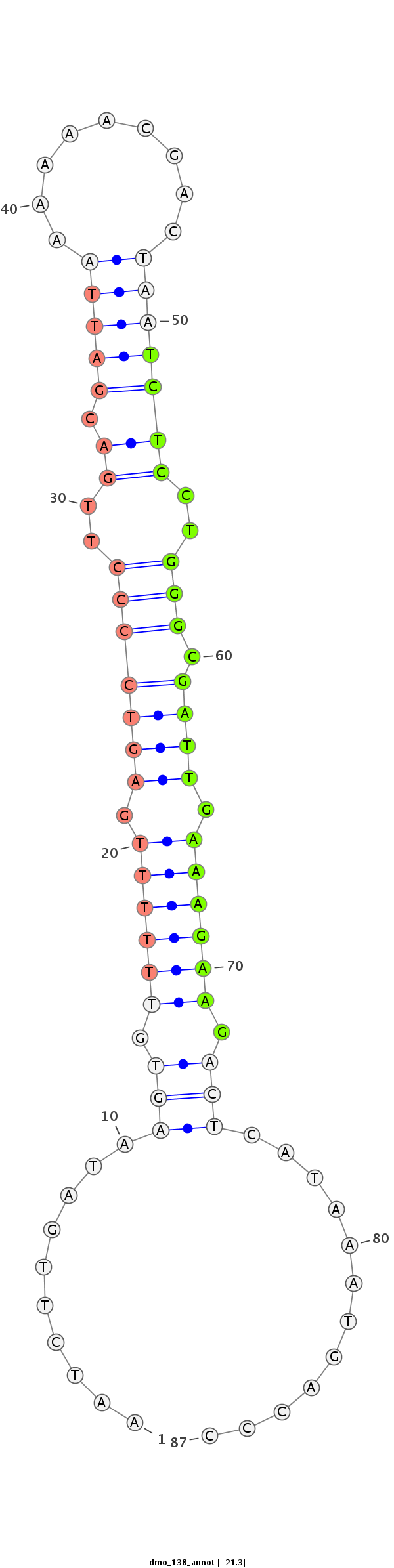
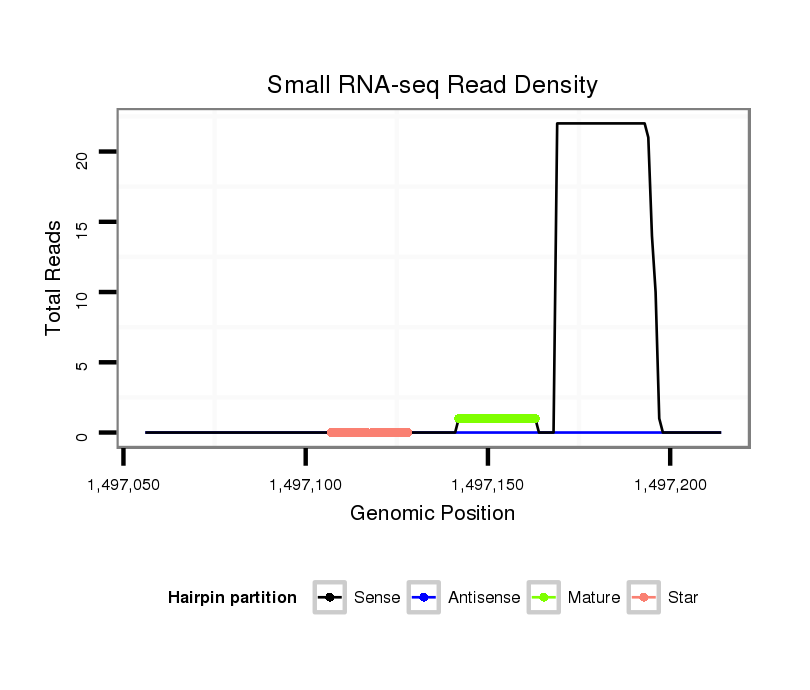
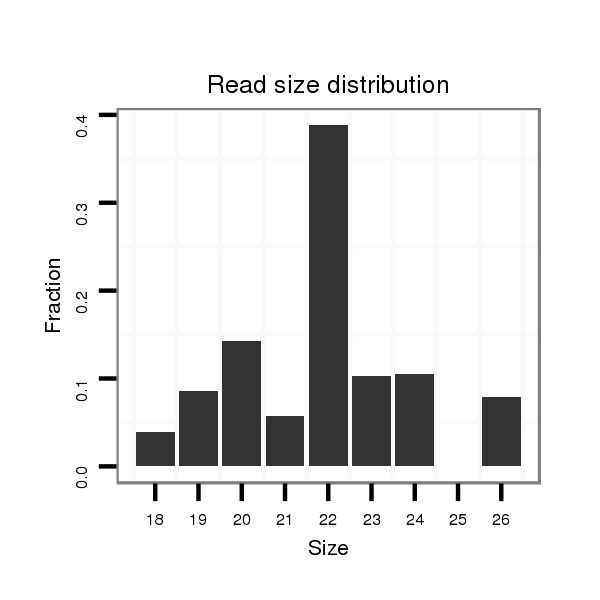
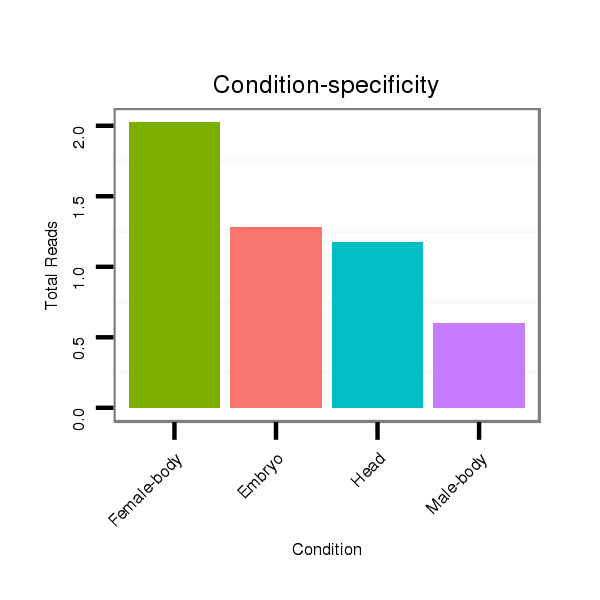
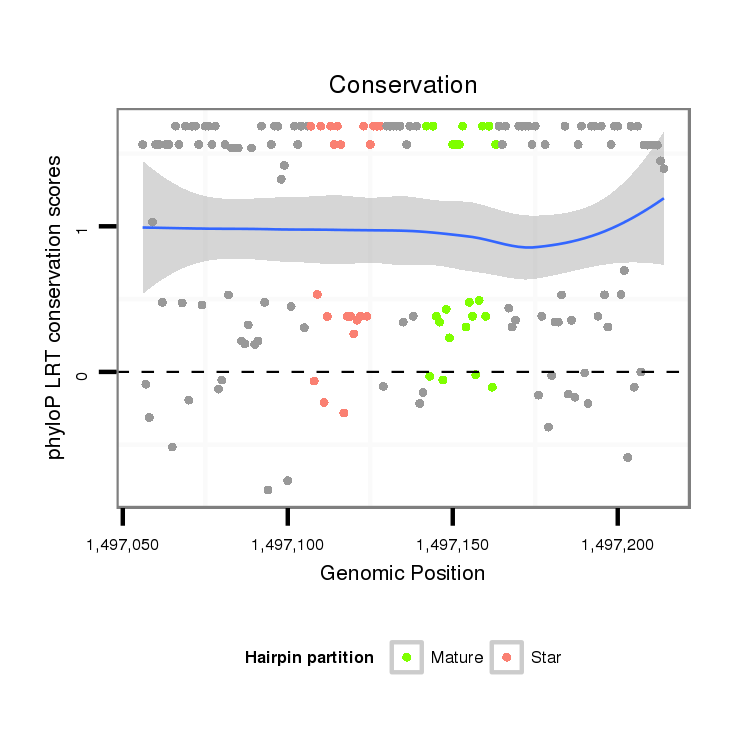
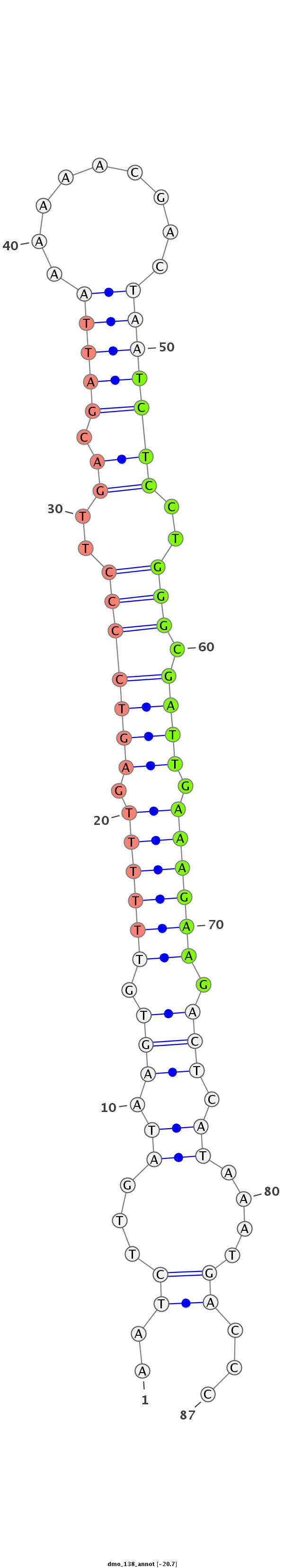
ID:dmo_138 |
Coordinate:scaffold_6540:1497106-1497164 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.7 | -20.3 |
 |
 |
intergenic
No Repeatable elements found
| mature | star |
|
CTCCCCAGCAAGCATTTGGAAGACTGTTTTAACAGAAATCTTGATAAGTGTTTTTTGAGTCCCCTTGACGATTAAAAAACGACTAATCTCCTGGGCGATTGAAAGAAGACTCATAAATGACCCTTCCATATGCTCAAAGTATGATTGCTTTCAAATACA
************************************..........(((.((((((.(((((((..((.(((((.........)))))))..))).)))).)))))).)))............************************************ |
Read size | # Mismatch | Hit Count | Total Norm | Total | M046 female body |
M060 embryo |
V110 male body |
V056 head |
V041 embryo |
V049 head |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................TAAATGACCCTTCCATATGCTCAAAGTA.................. | 28 | 0 | 8 | 9.63 | 77 | 31 | 46 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATGACCCTTCCATATGCTCAAAG.................... | 26 | 0 | 8 | 7.88 | 63 | 54 | 6 | 0 | 0 | 3 | 0 |
| .................................................................................................................TAAATGACCCTTCCATATGCTCAAAGT................... | 27 | 0 | 8 | 4.63 | 37 | 27 | 6 | 3 | 0 | 1 | 0 |
| ......................................................................................TCTCCTGGGCGATTGAAAGAAG................................................... | 22 | 0 | 5 | 1.60 | 8 | 4 | 1 | 2 | 1 | 0 | 0 |
| .................................................................................................................TAAATGACCCTTCCATATGCTCAAA..................... | 25 | 0 | 8 | 1.50 | 12 | 7 | 5 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATGACCCTTCCATATGCTCAAAGTAT................. | 29 | 0 | 8 | 1.13 | 9 | 7 | 2 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AAATGACCCTTCCATATGCTCAAAGTA.................. | 27 | 0 | 8 | 0.75 | 6 | 5 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AAATGACCCTTCCATATGCTCAAAGT................... | 26 | 0 | 8 | 0.63 | 5 | 5 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................CTAAATGACCCTTCCATATGCTCAAAGTA.................. | 29 | 1 | 8 | 0.50 | 4 | 2 | 2 | 0 | 0 | 0 | 0 |
| ........................................................................................TCCTGGGCGATTGAAAGAAG................................................... | 20 | 0 | 5 | 0.40 | 2 | 0 | 0 | 1 | 0 | 0 | 1 |
| ...................................................TTTTTGAGTCCCCTTGACGATT...................................................................................... | 22 | 0 | 8 | 0.38 | 3 | 1 | 0 | 0 | 1 | 0 | 1 |
| .........................................................................AAAAAACGACTAATCTCCTGGGCG.............................................................. | 24 | 0 | 6 | 0.33 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................TTGAAAGAAGACTCATAAATGACCCT................................... | 26 | 0 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AAATGACCCTTCCATATGCTCAAAG.................... | 25 | 0 | 8 | 0.25 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..................................................TTTTTTGAGTCCCCTTGACGATT...................................................................................... | 23 | 0 | 8 | 0.25 | 2 | 0 | 0 | 0 | 1 | 1 | 0 |
| .................................................................................................................TAAATGACCCTTCCATATGCTCAA...................... | 24 | 0 | 8 | 0.25 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AAATGACCCTTCCATATGCTCAAA..................... | 24 | 0 | 8 | 0.25 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATGACCCTTCCATATGCTCA....................... | 23 | 0 | 8 | 0.25 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................ATAAATGACCCTTCCATATGCTCAAAG.................... | 27 | 0 | 8 | 0.25 | 2 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................CGATTGAAAGAAGACTCATA............................................ | 20 | 0 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................................TCTCCTGGGCGATTGAAAGA..................................................... | 20 | 0 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................TCTCCGGGGCGATTGAAAGAAG................................................... | 22 | 1 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................TTAAAAAACGACTAATCTCCTGGGCG.............................................................. | 26 | 0 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................TCCTGGGCGATTGAAAGAAGAC................................................. | 22 | 0 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................AAAACGACTAATCTCCTGGGCGATTG.......................................................... | 26 | 0 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................ATCTCCTGGGCGATTGAAAGAAGA.................................................. | 24 | 0 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................TCTCCTGGGCGATTGAAA....................................................... | 18 | 0 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| ......................................................................................TCTCCTGGGCGATTGAAAGAAGA.................................................. | 23 | 0 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................CTCCTGGGCGATTGAAAGA..................................................... | 19 | 0 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................ACTCATAAATGACCCTTCCATATG........................... | 24 | 0 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................AAAACGACTAATCTCCTGGGC............................................................... | 21 | 0 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................TGCTCAAAGTATGCGTGCCTT........ | 21 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................GCTCAAAGTATGATTGCTTT........ | 20 | 0 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................AAAACGACTAATCTCCTGG................................................................. | 19 | 0 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATGACCTTTCCATATGCTCAAAGTA.................. | 28 | 1 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................TTTTGAGTCCCCTTGACGATT...................................................................................... | 21 | 0 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................TTTTTTTAGTCCCCTTGACGATT...................................................................................... | 23 | 1 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................................................................................................TAAATGACCCTTCCATATGCTC........................ | 22 | 0 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................................AATGACCCTTCCATATGCTCAAAGTA.................. | 26 | 0 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................CTGACCCTTCCATATGCTCAAAGTA.................. | 25 | 1 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATGACCCTTCCATATGC.......................... | 20 | 0 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AGATGGCCCTTCCATATGCTC........................ | 21 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...............................................................................................................CATAAATGACCCTTCCATATGCTCAA...................... | 26 | 0 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................ATGACCCTTCCATATGCTCAA...................... | 21 | 0 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATAACCCTTCCATATGCTCAAAGTA.................. | 28 | 1 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................TTTTTTGAGTCCCCTTGACG......................................................................................... | 20 | 0 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................................CATAAATGACCCTTCCATATGCTCAAAG.................... | 28 | 0 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATGACCCTGCCATATGCTCA....................... | 23 | 1 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATTACCCTTCCATATGCTCAAAGTA.................. | 28 | 1 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAATTGACCCTTCCATATGCTCAAAGTA.................. | 28 | 1 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................TGAATGACCCTTCCATATGCTCAAAGTAT................. | 29 | 1 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATGACCCTTCCATATGCTAAAA..................... | 25 | 1 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................TAAATGACCATTCCATATGCTCAAAGTA.................. | 28 | 1 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................GAAGACTCATAAATGACCCTT.................................. | 21 | 0 | 9 | 0.11 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................AAAGAAGACTCATAAATGACCT.................................... | 22 | 1 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................TTGAGTCCCCTTGACGATT...................................................................................... | 19 | 0 | 14 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................TTTGAGTCCCCTTGACGATTAAA................................................................................... | 23 | 0 | 14 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
|
GAGGGGTCGTTCGTAAACCTTCTGACAAAATTGTCTTTAGAACTATTCACAAAAAACTCAGGGGAACTGCTAATTTTTTGCTGATTAGAGGACCCGCTAACTTTCTTCTGAGTATTTACTGGGAAGGTATACGAGTTTCATACTAACGAAAGTTTATGT
************************************..........(((.((((((.(((((((..((.(((((.........)))))))..))).)))).)))))).)))............************************************ |
Read size | # Mismatch | Hit Count | Total Norm | Total | M046 female body |
V110 male body |
V056 head |
M060 embryo |
V041 embryo |
|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................TACCGGGAAGGTATGCCA......................... | 18 | 3 | 20 | 3.35 | 67 | 52 | 14 | 1 | 0 | 0 |
| ..........................................................CAGGGGAACTGCTAATTTTTTGCTGAT.......................................................................... | 27 | 0 | 11 | 0.27 | 3 | 3 | 0 | 0 | 0 | 0 |
| ................................................................................................CTAACTTTCTTCTGAGTATTTACTGGG.................................... | 27 | 0 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 |
| .....................................................................CTATTTTTTTGCTGATTAGAGGACCCG............................................................... | 27 | 1 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................CGAGTTTCATACTAACGAA......... | 19 | 0 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................AAGGTATACGAGTTTCATACTAACGA.......... | 26 | 0 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................TGAGTATTTACTGGGAAGGTATACGAG........................ | 27 | 0 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 |
| .........................................................................TTTTTTGCTGATTAGAGGACCCGCT............................................................. | 25 | 0 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 |
| ....................................................AAAACTCAGGGGAACTGCTAATTT................................................................................... | 24 | 0 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................ATTTACTGGGAAGGTATACGAGTTT..................... | 25 | 0 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................GGGAAGGTATACGAGTTTCATACTAA............. | 26 | 0 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TTACTGGGAAGGTATACGAGTTTCATAC................ | 28 | 0 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................................................TACTGGGAAGGTGTACTAGTGT..................... | 22 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................................................................ACTGGGAAGGTATACGAGTTT..................... | 21 | 0 | 8 | 0.13 | 1 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................CTTCTGAGTATTTACTGGGAAGGTAT............................. | 26 | 0 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 |
| ...........................................................................................................................AAGGTATACGAGTTTCATACTA.............. | 22 | 0 | 9 | 0.11 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................CAGGGGAACTGCTAATTTTTTGCTGATT......................................................................... | 28 | 0 | 10 | 0.10 | 1 | 1 | 0 | 0 | 0 | 0 |
| .........................................................TCAGGGGAACTGCTAATTTTTTGCTGAT.......................................................................... | 28 | 0 | 11 | 0.09 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................................................CTACTGGGAAGGTATGCCA......................... | 19 | 3 | 13 | 0.08 | 1 | 0 | 0 | 1 | 0 | 0 |
| ......TCGTTCGTAAACCTTCTGACAAAATT............................................................................................................................... | 26 | 0 | 13 | 0.08 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................GGAGGTATACGAGTTTCATA................. | 20 | 1 | 13 | 0.08 | 1 | 1 | 0 | 0 | 0 | 0 |
| ........GTTCGTAAACCTTCTGACAAAATTGT............................................................................................................................. | 26 | 0 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................TTTCTTCTGAGTATTTACTGGG.................................... | 22 | 0 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...........CGTAAACCTTCTGACAAAATTGTCTTT......................................................................................................................... | 27 | 0 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................AGGGGAACTGCTAATTTTTTGC.............................................................................. | 22 | 0 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...........CGTAAACCTTCTGACAAAATTGTCTT.......................................................................................................................... | 26 | 0 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................TCTTCTGAGTATTTACTGGG.................................... | 20 | 0 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................CAGGGGAACTGCTAATTTTTTGCT............................................................................. | 24 | 0 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................CAGGGGAACTGCTAATTT................................................................................... | 18 | 0 | 15 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...GGGTCGTTCGTAAACCTTC......................................................................................................................................... | 19 | 0 | 16 | 0.06 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................CAGGGGAACGGCTGATTG................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................................................CTACTGGGAAGGTATGCC.......................... | 18 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droMoj3 | scaffold_6540:1497056-1497214 + | dmo_138 | CTCCCCAGCAAGCATTTGGAAGACTGTTTTAACAGAA---ATCTTGATAAGTGTTTTTTGAGTCCCCTTGACGATTAAAAAACGACTAATCTCCTGGGCGATTGAAAGAAGACTCATAAATGACCCT-----------TCCATATGCTCAAAGTATGATTGCTTTCAAATA-CA |
| droWil2 | scf2_1100000004936:1001923-1002081 - | CCACCCTGCAAGCATTTGGAAGACTGTTTTAACAGAAATCGTCTT---GAGTCTTTTTTGAGTCCCCTTGACGATTAAAAAACGACTAATCTCCTGGGCGATCGAAAGAAGACTTATAAATGACCCT-----------TCCATATGCTCAAAGTATGATTGGTTTGAAATA-CA | |
| droBip1 | scf7180000395829:1972-2129 - | C---CCAGCCAGCAATTGGAAGATTGTTTTTGCATTAAGCACCTTGATGAGTCTTCTTCGAGTCGTTGAAATGATTGAAAAAAGATTCGTTTTATTCGCGACTAGAAAATGACTCGAAAATGAGTCG-----------CAAATGATCTTCAAATACGATT--TATAAAATAGCA | |
| droKik1 | scf7180000301533:13118-13279 + | CTACCCAGCAAGCACTTGGAAGATCGC---------AATCATCTTGAGGAGTCTTTATGGAGTCATTGAAATGATTAAAAAAAGATTTGTCTTACGCGCGACTAAAAAATGACTCGAAAATGACTCCAAAAAGAGTCGTAAGTTACCTCTAAATACGATT--TATAAAATAGC- | |
| droFic1 | scf7180000452721:923-1072 - | C---CCAGCCAGCAATTGAAAGACTGTTTTTGCATTAAGCACCTTGATGAGTCTTCTTCGAGTCGTTGAAATGATTGAAAAAAGATTCGTTTTATGCGCGACTAACAAATGACTCGAAAATGACTCG-----------AAAATGATCTTCAAATACGATT--TATA-------- | |
| droEug1 | scf7180000407349:2194-2351 - | C---CCAGCAAGTAATTGGAAGATCGTTTTTGAATTAATTAGCTTGACGAGTCTTCTTCCAGTCGTTGAAATGATTAAAAAAAGATTCTTTTTACGCGCGACTAGAAAAAGACTCGAAAATGAGTCG-----------TAAATGATCTCCAAATGCGATA--TTTAAAATAGCA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/15/2015 at 02:37 PM