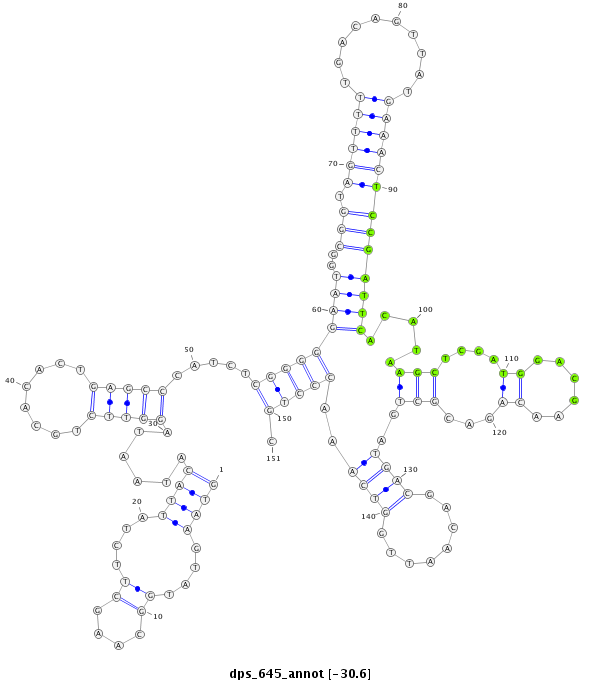
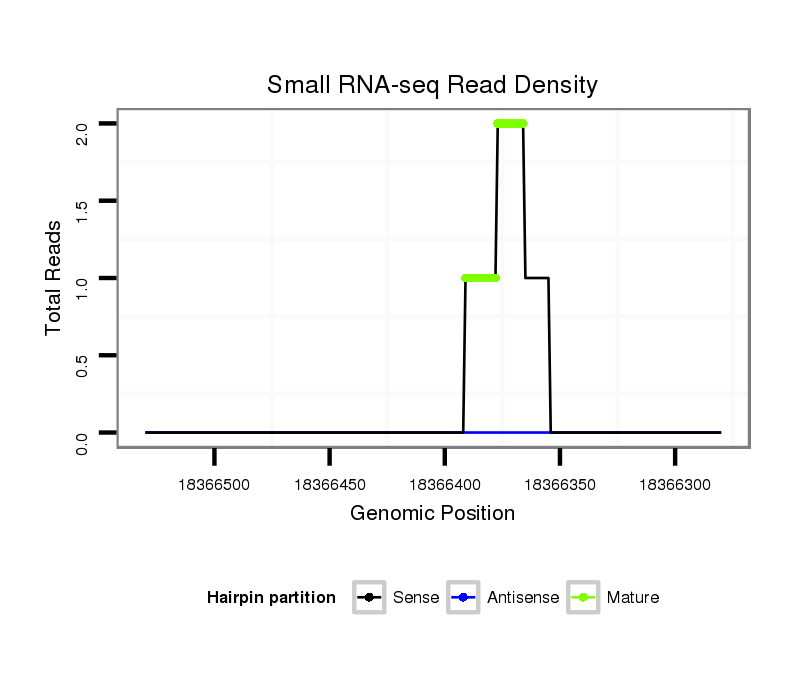
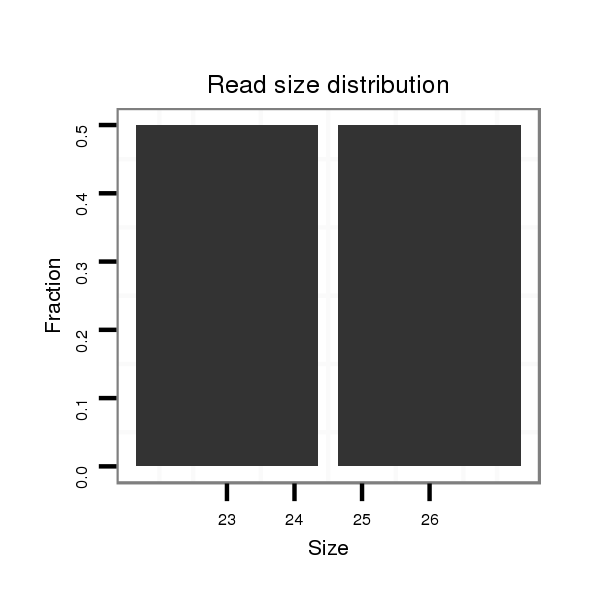
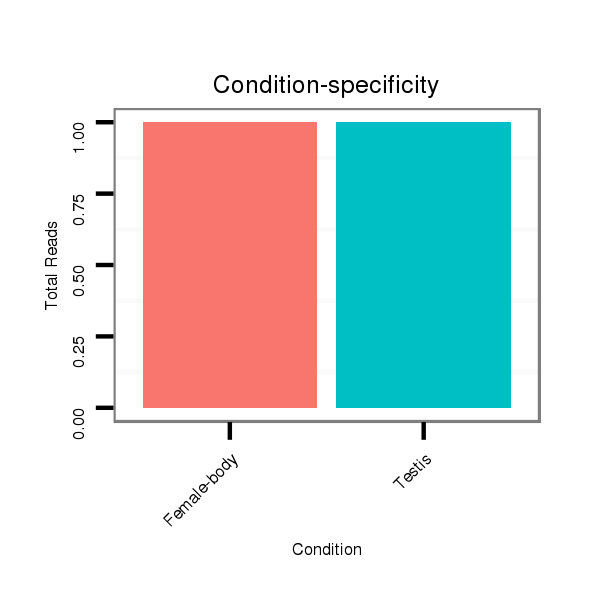
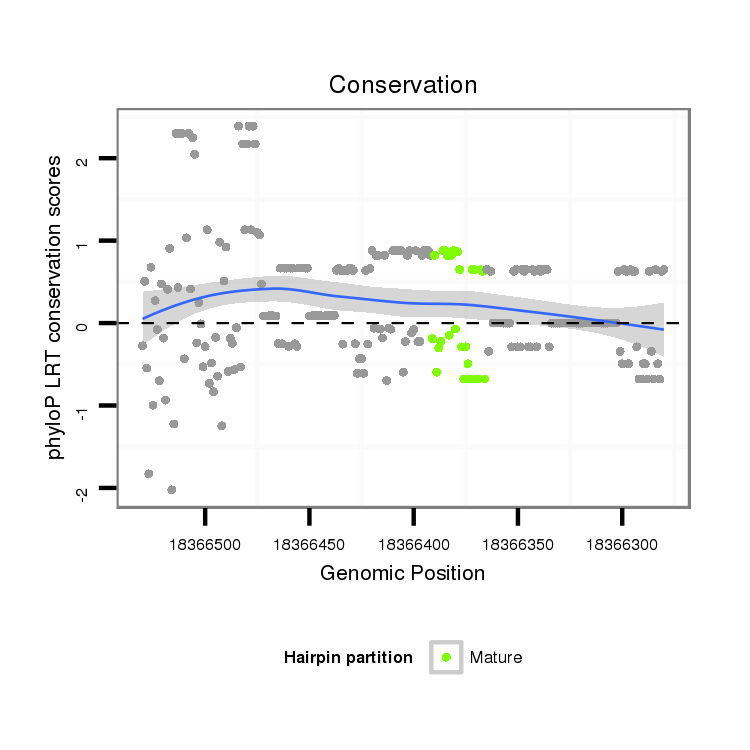
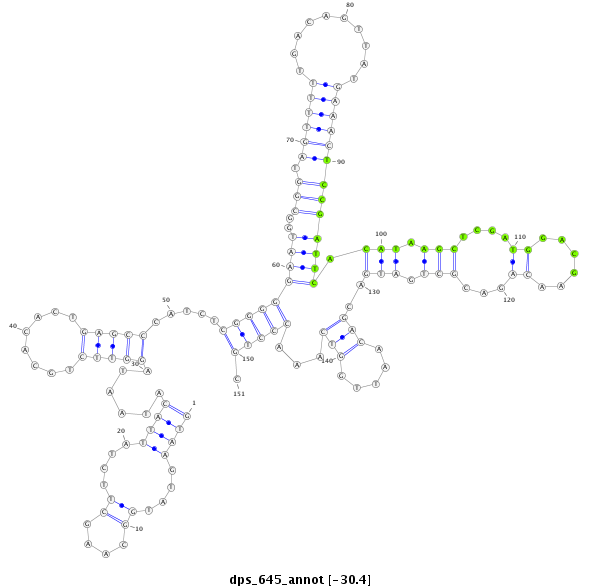
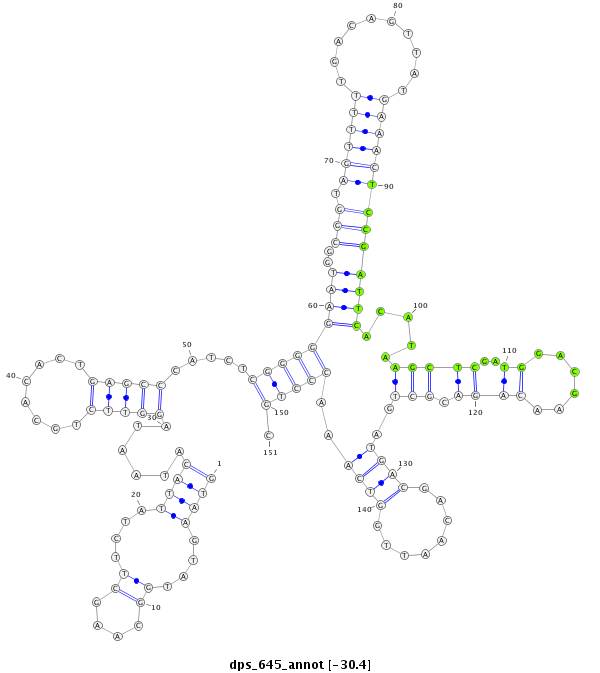
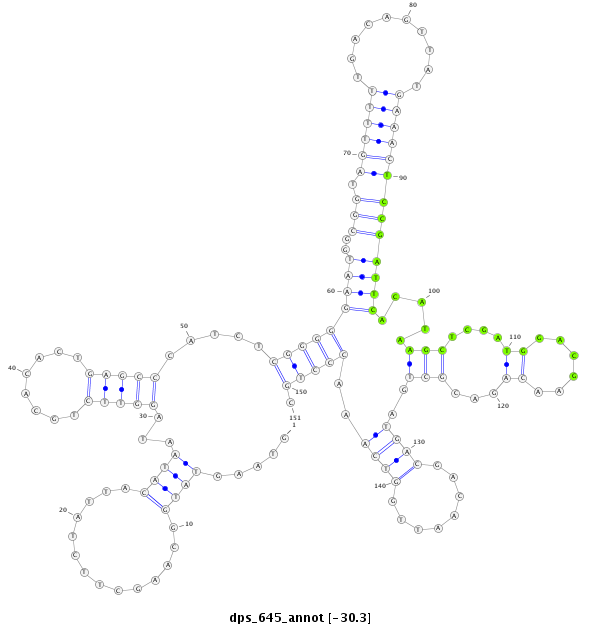
ID:dps_645 |
Coordinate:2:18366330-18366480 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -30.4 | -30.4 | -30.3 |
 |
 |
 |
CDS [Dpse\GA30173-cds]; exon [Dpse\GA30173:9]; intron [Dpse\GA30173-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AAAAATTTCACAACTGTTTTCAATACACCGTTGCTTCGAACTATTCAACAGTAAGTATGGCAAGCTTCTATTACATAATAGGTTCTGCACACTGAGCCCATCTCGGGGGAATGGCGGTAGTTTTTGACAGTTATGAAACTCCGATTCACATAAGCTCGATGGACGAACAGACGCTGATGACGACAATTGGTCAAACCCTGCCTGCAATGCTTGTATCATTACTTCGGTCTGTCGAGCTGCGGCCTGTGCCA **************************************************((((....((....))....))))......(((((........))))).....(((((((((..(((.((((((..........))))))))))))).....(((....((......))...)))..((((........))))..))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M040 female body |
SRR902011 testis |
V112 male body |
GSM444067 head |
|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................................TCCGATTCACATAAGCTCGATGGACG...................................................................................... | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .........................................................................................................................................................GCTCGATGGACGAACAGACGCTG........................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| .....................................................................................................CTCGGGTAAATGGCGGGAGT.................................................................................................................................. | 20 | 3 | 11 | 0.73 | 8 | 8 | 0 | 0 | 0 |
| ..............................................................................................................................................................................TGATGATGACATCTGGTCAA......................................................... | 20 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 |
| ......................................................................................................TCGGGGCAATGGAGGTAG................................................................................................................................... | 18 | 2 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 |
| ...................................................................................................ACCTCGGGGGGATGACGGT..................................................................................................................................... | 19 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 |
| .....................................................................................................CTCGGGGAAACGGCGGGAGT.................................................................................................................................. | 20 | 3 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 |
| ............................................................................................................................................................................ACTGACGACGTCAATTGGT............................................................ | 19 | 3 | 16 | 0.06 | 1 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................................TGTCGAGCTGTGGCCTGTG... | 19 | 1 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 |
| ..........................................................................................................GGCAATGGCGGCAGTATTTG............................................................................................................................. | 20 | 3 | 18 | 0.06 | 1 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................GTAGAGCTGTGGCCTGTGCC. | 20 | 2 | 18 | 0.06 | 1 | 0 | 0 | 0 | 1 |
| ....................................................................................................................................................................................................................................CTGTGGAGCTGCGGCCTGTGTT. | 22 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 1 |
|
TTTTTAAAGTGTTGACAAAAGTTATGTGGCAACGAAGCTTGATAAGTTGTCATTCATACCGTTCGAAGATAATGTATTATCCAAGACGTGTGACTCGGGTAGAGCCCCCTTACCGCCATCAAAAACTGTCAATACTTTGAGGCTAAGTGTATTCGAGCTACCTGCTTGTCTGCGACTACTGCTGTTAACCAGTTTGGGACGGACGTTACGAACATAGTAATGAAGCCAGACAGCTCGACGCCGGACACGGT
**************************************************((((....((....))....))))......(((((........))))).....(((((((((..(((.((((((..........))))))))))))).....(((....((......))...)))..((((........))))..))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M040 female body |
V112 male body |
M022 male body |
GSM343916 embryo |
M059 embryo |
|---|---|---|---|---|---|---|---|---|---|---|
| ............TGACGAATGTTATGGGGCAAC.......................................................................................................................................................................................................................... | 21 | 3 | 1 | 89.00 | 89 | 60 | 29 | 0 | 0 | 0 |
| ............TGACGAATGTTATGGGGCAA........................................................................................................................................................................................................................... | 20 | 3 | 2 | 86.50 | 173 | 126 | 46 | 1 | 0 | 0 |
| ............TGACGAATGTTATGGGGCA............................................................................................................................................................................................................................ | 19 | 3 | 14 | 4.43 | 62 | 53 | 7 | 0 | 0 | 2 |
| .............GACGAATGTTATGGGGCAAC.......................................................................................................................................................................................................................... | 20 | 3 | 7 | 1.57 | 11 | 9 | 2 | 0 | 0 | 0 |
| ............TGACTAATGTTATGGGGCAAC.......................................................................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...........ATGACGAAAGTTATGGGGCAA........................................................................................................................................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 |
| ............TGACTAATGTTATGGGGCAA........................................................................................................................................................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 |
| ............TGACGAACGTTATGGGGCAAC.......................................................................................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...........................GGCAAATAAGCTAGATAAGT............................................................................................................................................................................................................ | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..............ACGAATGTTATGAGGCAAC.......................................................................................................................................................................................................................... | 19 | 3 | 15 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................CCGTTCGAAAATGCTGTAT.............................................................................................................................................................................. | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| dp5 | 2:18366280-18366530 - | dps_645 | AAAAATTTCACAACTGTTTTCAATACACCGTTGCTTCGAACTATTCAACAGTAAGTATGGCAAGCTTCTATTACATAATAGGTTCTGCACACTGAGCCCATCTCGGGGGAATGGCGGTAGTTTTTGACAGTTATGAAACTCCGATTCACATAAGCTCGATGGACGAACAGACGCTGATGACGACAATTGGTCAAACCCTGCCTGCAATGCTTGTATCATTACTTCGGTCTGTCGAGCTGCGGCCTGTGCCA |
| droPer2 | scaffold_3:745372-745521 - | AAACATTTCACAACTGTTTTCAATACACCGTTGCTTCGAACTATTCAACAGTAAGTATGGCAAGCTTCTATTACATAATAGGTTCTGCACACTGAGCCCATCTCGGGGGAATGGCGGTAGTTTTTGACAGTTATGAAACTCCGATTCACA----------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004943:13877281-13877337 - | AAGGAATATACAGTGCTTTTTAATACACCATTGGAAAGGACCTTAGAACAGTAAGTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droVir3 | scaffold_12958:792271-792327 + | AGGCATTTAATTACCATTTTTAATACACCGGTTGCTCGGACAAGTGAACAGTAAGTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droMoj3 | scaffold_6540:4211762-4211819 + | AGCCATTTAATTACGATTTTCAACACTCCGCTTAATCGAACGATAAATCAGTAAGTAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15116:517407-517464 - | AAGGATTTTGTTACGGTATTTAATACAGCACTAGCTCGATCTGATGAACAGTAAGTAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13082:629248-629303 - | CAGGAGTATATCGCGGTTTTTAATACAATATTAAACAAAAGCATTCAACAGTAAGT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000391495:18321-18378 + | GAGGAATATGTTGCTGTTTTTAATACAATGTTGAATGAAACCCTTCAACAGTAAGTGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302745:5502-5578 - | TAATACGGT----------------------CTTCCCACACAGCGCATCTGTGAGGA-----------------------------------------------------ACGGTTGGGATTTTTCGCAGATGCGAAACTCGTGTTCCCACA--------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000451999:4424-4496 - | AAGGACTACATCACCATTTTTCACACCCCATTGCTTAACACAATACAGCAGTAAGTAC-------CTTTAATATTTAATA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000776721:6873-6999 - | TCTTCCCATA-----------------------------------------------------------------------------------GAGTCCATCCGTTCGAAACGGTTGAGATTTTTGACAAATATGAAACACCGATTCCCACAAAGCACATCCACCATC---------CGATAACAGCTGATCAAAT--------------------------------CATTCTAGCCCGTTGCACTTGCA | |
| droBia1 | scf7180000299317:18134-18190 - | CAGGAATACACAACACTTTTCAATACTCGACTTCATCATACAATTCAACAGTAAGTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000412857:7343-7399 - | AAGGAATATATAACTCTTTTCAATACACAACTGCAGCATACAACTCAGCAGTAAGTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000405977:547-553 + | AAAAATT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 3l:23180892-23180949 - | AAAGGTTATATTACACTATTTAATACTCCTCTGCATAATTCAATAAATCAGTAAGTAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_64:72996-73053 - | AAAGGTTATATTACACTATTTAATACTCCTCTGCATAAATCAATAAATCAGTAAGTAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | v2_chrUn_3671:910-932 - | AAAAATATCAAAACATTTTTCAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/17/2015 at 10:54 PM