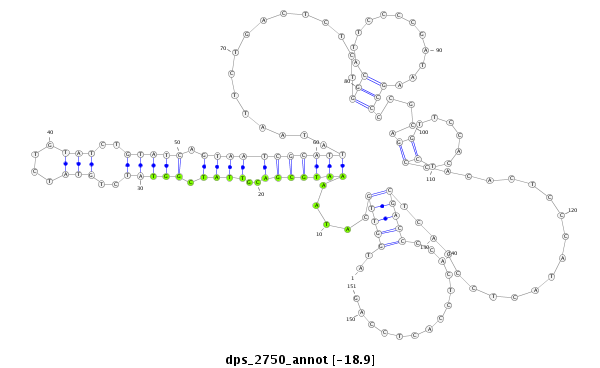
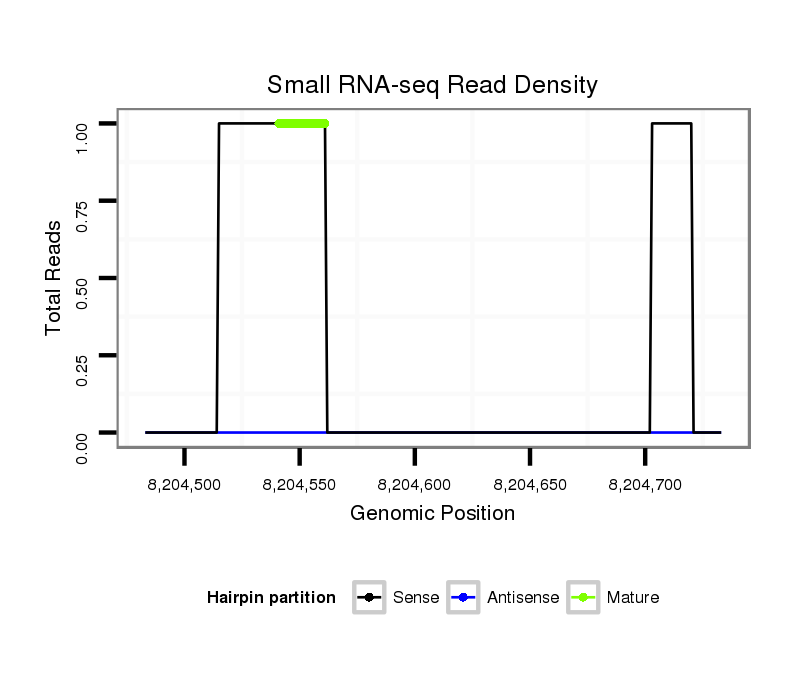
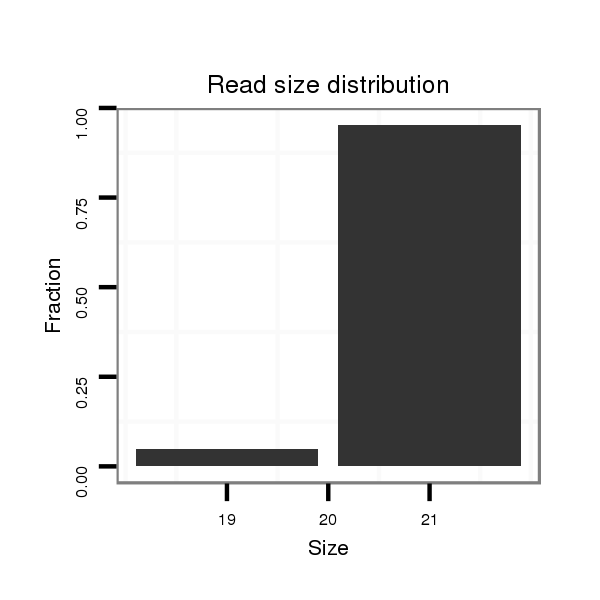
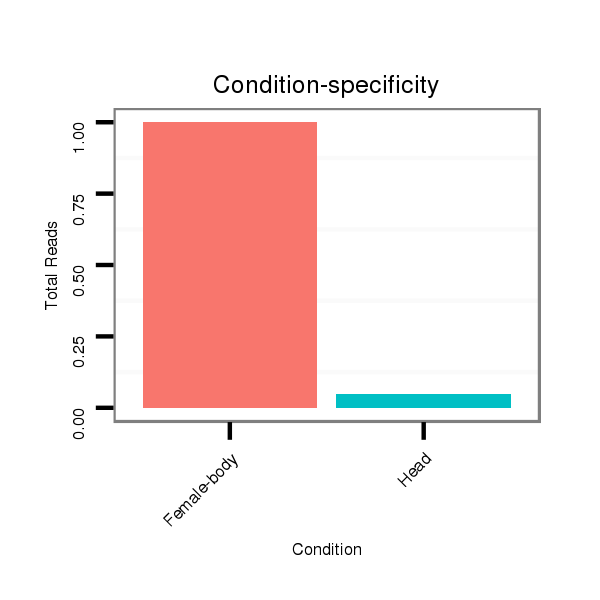
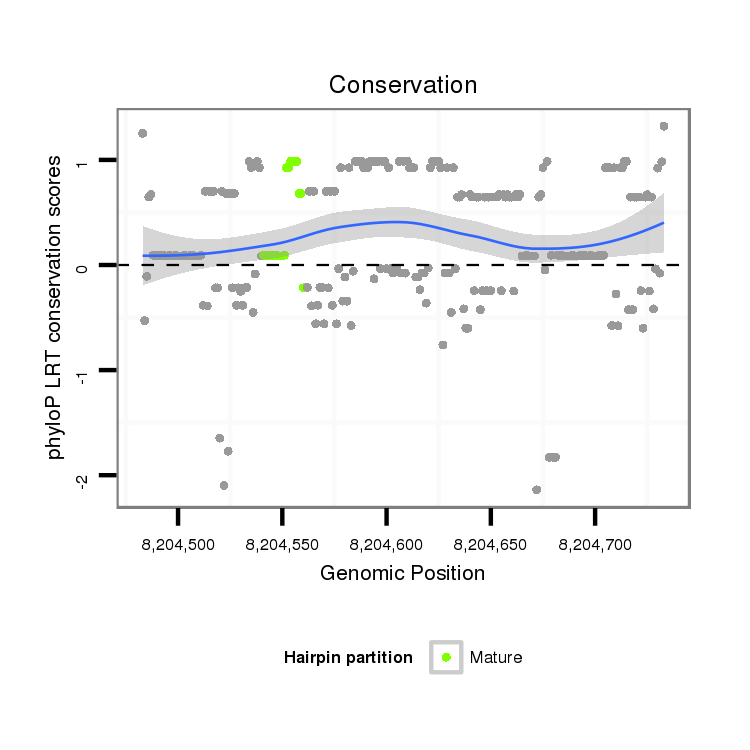
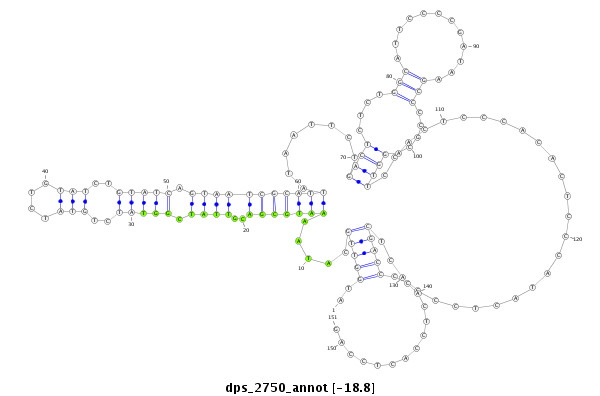
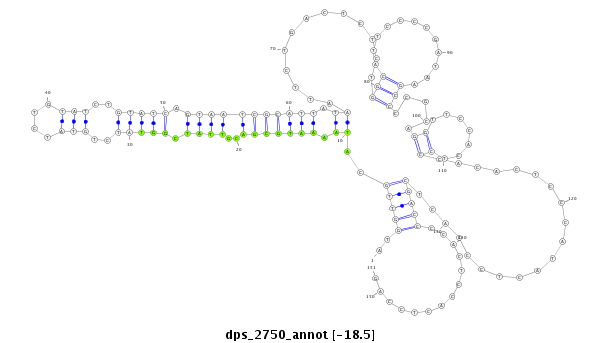
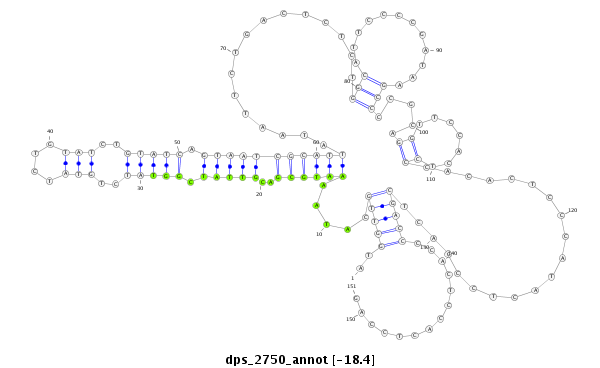
ID:dps_2750 |
Coordinate:XL_group1e:8204533-8204683 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -18.8 | -18.5 | -18.4 |
 |
 |
 |
CDS [Dpse\GA23038-cds]; exon [dpse_GLEANR_1149:2]; intron [Dpse\GA23038-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GCCCAACACAAGGTCGTCCGTCCATGGCTTTCTTTCCATGGTGCGAGTTCATGGTTGCATAAAATGCGACGTTATCGGTATCTGTATCTGTATCTGTATCAGTAATCGCATTATAATTCTGACTCTCTGGCATTCCCCGATAAGCCCCGCAGGTTCCACTCCCACACTCCCATACTCCCACTCGACCCCACTCCACTCCAGTCCCCTGCCAGTGGAGGCACTCCCTCCTCCAACGCGCCAGGTGGATGGTT **************************************************..(((((.....(((((((..((((.(((((..(((....)))..))))).)))))))))))................(((............))).....((.......))....................)))))..............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M040 female body |
V112 male body |
GSM444067 head |
|---|---|---|---|---|---|---|---|---|
| .........................................................................................................................................................................................................................................CGCGTCAGGTGGATGGTT | 18 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 |
| ................................TTTCCATGGTGCGAGTTCATGGTTGC................................................................................................................................................................................................. | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 |
| ............................................................................................................................................................................................................................CTCCCTCCTCCAACGCGC............. | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 |
| ..........................................................ATAAAATGCGACGTTATCGGT............................................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 |
| ................................................................................................................................................................................................................CCAGTGCAGGCGCTCCCT......................... | 18 | 2 | 11 | 0.45 | 5 | 5 | 0 | 0 |
| ................................................................................................................................................................................................................CCAGTGCAGGCGCTCCCTA........................ | 19 | 3 | 20 | 0.30 | 6 | 6 | 0 | 0 |
| ......................................................................................................................................................................................................................................CAACGGGACAGGTAGATGGTT | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 |
| ................................................................................................................................................................................................................CCAGTGCAGGCGCTCCCTACT...................... | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 |
| .................................................................................................................................................................................................................CAGTGCAGGCACTCCCTATT...................... | 20 | 3 | 8 | 0.13 | 1 | 1 | 0 | 0 |
| ..............................TCTGTGCATGGGGCGAGTT.......................................................................................................................................................................................................... | 19 | 3 | 17 | 0.06 | 1 | 1 | 0 | 0 |
| ...............................................................................ATCTGTATCTGTATCTGTA......................................................................................................................................................... | 19 | 0 | 20 | 0.05 | 1 | 0 | 0 | 1 |
|
CGGGTTGTGTTCCAGCAGGCAGGTACCGAAAGAAAGGTACCACGCTCAAGTACCAACGTATTTTACGCTGCAATAGCCATAGACATAGACATAGACATAGTCATTAGCGTAATATTAAGACTGAGAGACCGTAAGGGGCTATTCGGGGCGTCCAAGGTGAGGGTGTGAGGGTATGAGGGTGAGCTGGGGTGAGGTGAGGTCAGGGGACGGTCACCTCCGTGAGGGAGGAGGTTGCGCGGTCCACCTACCAA
**************************************************..(((((.....(((((((..((((.(((((..(((....)))..))))).)))))))))))................(((............))).....((.......))....................)))))..............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M040 female body |
V112 male body |
GSM343916 embryo |
|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................TTCGGGTCGTCCAAGGGGA........................................................................................... | 19 | 2 | 1 | 2.00 | 2 | 0 | 2 | 0 |
| .............................................................................................................................................TTCGGGGCGTCCAAGGGGATGC........................................................................................ | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 |
| ......................................................................CAAGAGCCATATACATAGAGATAGAC........................................................................................................................................................... | 26 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 |
| ................................................................................................................................................................................................................GGTCACGTCCGCGAGGGAT........................ | 19 | 3 | 20 | 0.85 | 17 | 17 | 0 | 0 |
| .........................................................................................................................................................................GGTATGAGGGTGGTCTTGGGT............................................................. | 21 | 3 | 5 | 0.80 | 4 | 4 | 0 | 0 |
| .............................................................................................................................................TTCGGGTCGTCCAAGGGGATG......................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 |
| .............................................................................................................................................TTCGGGGCGTCTAAGGCG............................................................................................ | 18 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 |
| ................................................................................................................................................................................................................GGTCACGTCCGCGAGGGA......................... | 18 | 2 | 11 | 0.36 | 4 | 4 | 0 | 0 |
| ..............................................................................................................................................TCGCGGCGTCCAAGGGGATG......................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 |
| ......................................................................................................................................................................................................................................GTTGTCCTGTCCACCTACCAA | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 |
| ................................................................................................................................................................................................................GGTCACGTCCGCGAGGGATGA...................... | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 |
| ......................................................................................................................................................................................................................................GTTGCCCTGTCCATCTACCAA | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 |
| .........................................................................................................................................................AAAATGAGGGTGTGAGGGT............................................................................... | 19 | 2 | 7 | 0.14 | 1 | 1 | 0 | 0 |
| .........................................................................................................................................................................GGTATGAGGGTGGTCTTGGG.............................................................. | 20 | 3 | 10 | 0.10 | 1 | 1 | 0 | 0 |
| ..............................................................................................................................................CCGCGGCGTCCGAGGTGAGG......................................................................................... | 20 | 3 | 14 | 0.07 | 1 | 1 | 0 | 0 |
| .............................................................................................................................................TCCGTGGCGTCCAAGGGGA........................................................................................... | 19 | 3 | 14 | 0.07 | 1 | 1 | 0 | 0 |
| .........................................................................................................GGCGTAAAATTAGGACTGA............................................................................................................................... | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 1 |
| ........................................................................................................................................................................................TGGGCTGAGGTGAAGTCA................................................. | 18 | 2 | 18 | 0.06 | 1 | 0 | 0 | 1 |
| ...............................................................................................................................................................................................................TGGTCACGTCCGCGAGGGA......................... | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 |
| .............................................................................................................................................TCGGGGGCGTCCAAGGGGA........................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| dp5 | XL_group1e:8204483-8204733 + | dps_2750 | GCCCAACACAAGGTCGTCCGTCCATGGCTTTCTTTCCATGGTGCGAGTTCATGGTTGCATAAAATGCGACGTTATCGGTATCTGTATCTGTATCTGTATCAGTAATCGCATTATAATT------------CTGACTCTCTGGCATTCC--CCGATAAGCCCCGCAGGTTCCACTCCCACACTCC----CATACTCCC---ACTCGACCCCACTCCACTCCAGTCCCCTGCCAGTGGAGGCACTCCCTCCTCCAACGCGCCAGG----------------T----------GGATGGTT |
| droPer2 | scaffold_13:362226-362476 - | GCCCAACACAAGGTCGTCCGTCCATGGCTTTCTTTCCGTCGAGCGAGTTCATGGTTGCATAAAATGCGACGTTATCGGTATCTGTATCTGTATCTGTATCAGTAATCGCATTATAATT------------CTGACTCTCTGGCATTCC--CCGATAAGCCCCGCAGGTTCCACTCCCACACTCC----CATACTCCC---ACTCGACCCCCCTCTATTTTAGTCCCCTGCCAGTGGAGGCACTCCCTCCTCCAACGCGCCAGG----------------T----------GGATGGTT | |
| droBip1 | scf7180000396431:535535-535719 + | GTACA----------------------------------------------TGCATG------------CGTTAT-------------------CGGGGCGATAATCGCATAATGATCCATTAAAAAGTGCCAGCTTTTTGGCGCTTT--GCGATAAGATCTCCGGGTGGGACCCCAGCGCCCCACCCCACACTCCTTGCACT-------TCTCCA---------------------------CCCCCAACCAAAGAGCCGCGAATAACCAAAACCAGTTGGCCGGCCCCAGCCGATT | |
| droEle1 | scf7180000491006:2436509-2436512 + | GAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T | |
| droBia1 | scf7180000301760:2656456-2656459 + | GAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T | |
| dm3 | chrX:8926759-8926762 + | GAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T | |
| droSim2 | x:8475050-8475053 + | GAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T | |
| droSec2 | scaffold_34:289110-289113 + | GAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T | |
| droYak3 | X:12386115-12386264 - | GCT--------------------------GTATTTTTGTGGTGTGCAAGTGTGGATG------------CGTTATCGACGTATCGGCGTATCTGCGTATCTGTAATCGCATTATGATCCATTAAAATGCGCCAGTTTTTTGGCATGTTAAGTGATAAGGTCTGCG------------------------------------------------CCA---------------------------CCCACACCCAA---------------------------GCCGCTCCC-------- | |
| droEre2 | scaffold_4690:6187392-6187398 - | GA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGATT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/18/2015 at 05:22 AM