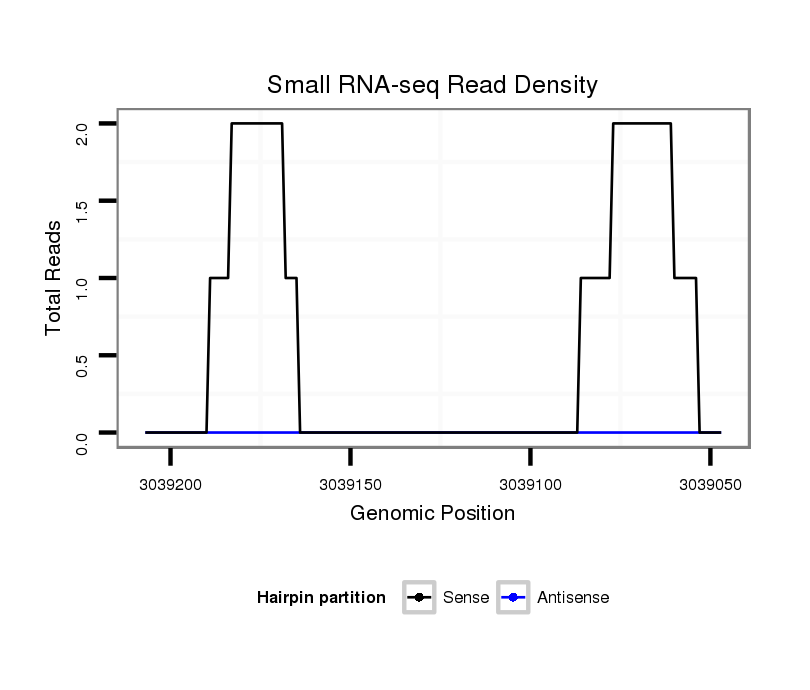
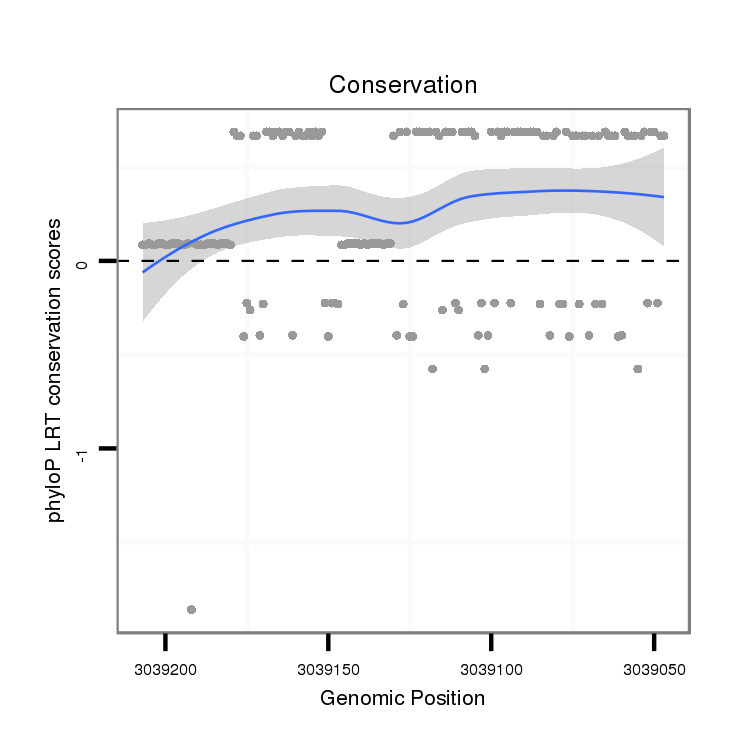
ID:dps_1387 |
Coordinate:4_group1:3039097-3039157 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dpse\GA25230-cds]; exon [dpse_GLEANR_16624:3]; CDS [Dpse\GA25230-cds]; exon [dpse_GLEANR_16624:4]; intron [Dpse\GA25230-in]
No Repeatable elements found
| ##################################################-------------------------------------------------------------################################################## CCTGCAAGGATACCATTCGCAAAGTATCAGCCAACGACATGAAGTTTCTGGTGAGTAGATCGGAAAACTGAATTCTTGAACAGCAAAAACTCATTTATATTTCTTCTATAGATTATTTTATTCGCTCTGCTGGCCGCTGCCCTGGCCTTGCCCCAATATGG **************************************************(((((((((((((....))))..)))............)))))).................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M059 embryo |
M040 female body |
GSM343916 embryo |
V112 male body |
SRR902012 CNS imaginal disc |
|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................................................TCGCTCTGCTGGCCGCTGCCCTGGCC.............. | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..................GCAAAGTATCAGCCAACGACA.......................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| ........................TATCAGCCAACGACATGAA...................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................TGGCCGCTGCCCTGGCCTTGCCCC....... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................GCTCTGCTGGCCGCTGCCCTGGCCC............. | 25 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .............................................TTTTGGTGAGTGGGTCGGAAA............................................................................................... | 21 | 3 | 5 | 0.40 | 2 | 0 | 0 | 0 | 2 | 0 |
| ..............................................................................................................GATTATTTTATTCGGTTTGATG............................. | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 |
| .........................................AATTATCTGGTGAGTAGAGC.................................................................................................... | 20 | 3 | 6 | 0.33 | 2 | 0 | 0 | 0 | 0 | 2 |
| ........................TAACAACCAACGACATGA....................................................................................................................... | 18 | 2 | 13 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 |
|
GGACGTTCCTATGGTAAGCGTTTCATAGTCGGTTGCTGTACTTCAAAGACCACTCATCTAGCCTTTTGACTTAAGAACTTGTCGTTTTTGAGTAAATATAAAGAAGATATCTAATAAAATAAGCGAGACGACCGGCGACGGGACCGGAACGGGGTTATACC
**************************************************(((((((((((((....))))..)))............)))))).................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M040 female body |
V112 male body |
|---|---|---|---|---|---|---|---|
| ......................................................................TTAAGAACATGTCGTTTACGAG..................................................................... | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 |
| ..............TATGCGCGTCATAGTCGGT................................................................................................................................ | 19 | 3 | 14 | 0.07 | 1 | 1 | 0 |
| ...................GCTATATAGTCGGTTGCTGT.......................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| dp5 | 4_group1:3039047-3039207 - | dps_1387 | CCTGCAAGGATACCATTCGCAAAGTATCAGCCAACGACATGAAGTTTCTGGTGAGTAGATCGGAAAACTGAATTCTTGAACAGCAAAAACT----------------------CATTTATATTTCTTCTATAGATTATTTTATTCGCTCTGCTGGCCGCTGCCCTGGCCTTGCCCCAATATGG |
| droPer2 | scaffold_5:2181528-2181688 + | CCTGCAAGGATACCACTCGCAAAGTATCAGCCAACGACATGAAGTTTCTGGTGAGTAGATCGGAAAACTGAATTCTTGAACAGCAAAAACT----------------------CATTTATATTTCTTCTATAGATTATTTTATTCGCTCTGCTGGCCGCTGCCCTGGCCTTGCCCCAATATGG | |
| droVir3 | scaffold_12963:4989817-4989955 + | dvi_10523 | ----------------------------AGCAGTCGCTATGAAGTTGCTGGTGAGTGTGCT----------------GCATATAAAAAAGTCTAAACATACTATTGGTATTTACTTTTGAATTTCGCGGACAGATCATTTTATTTGCGCTATTTGCTGCGGTCTTGGCAGTGCCGCAGTACGG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
Generated: 05/18/2015 at 12:34 AM