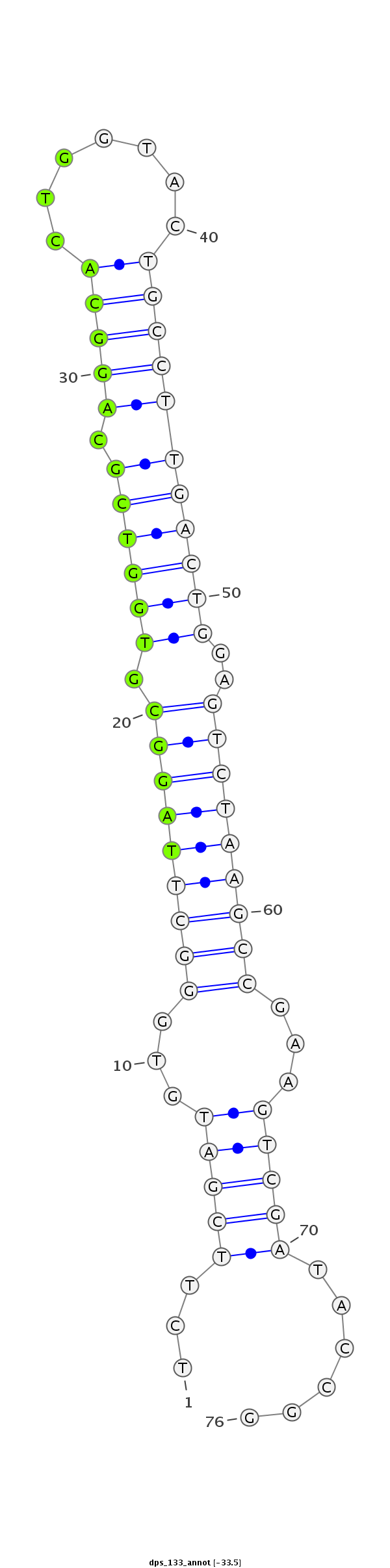
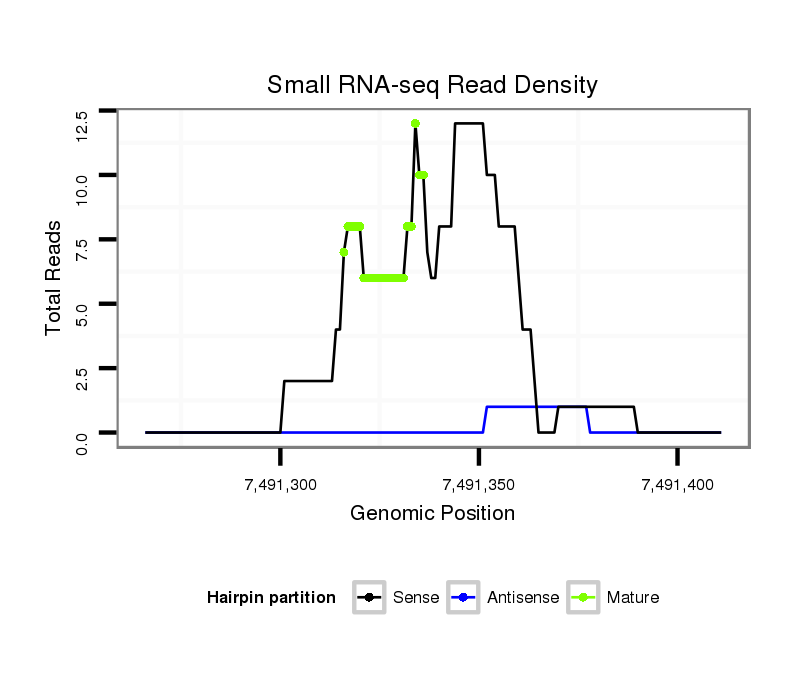
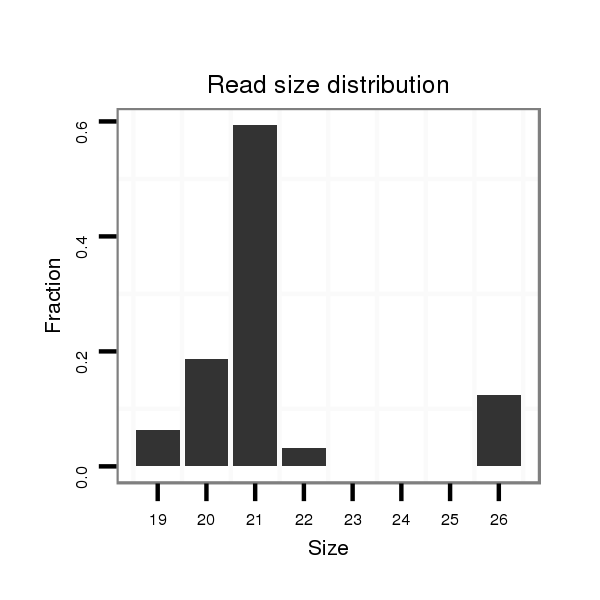
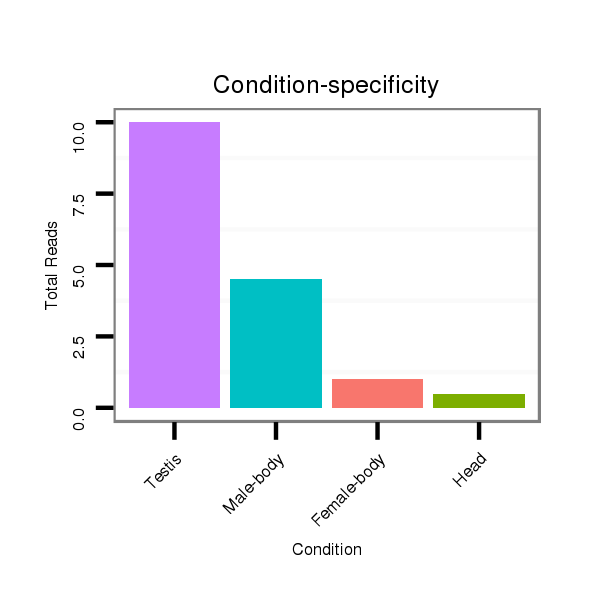
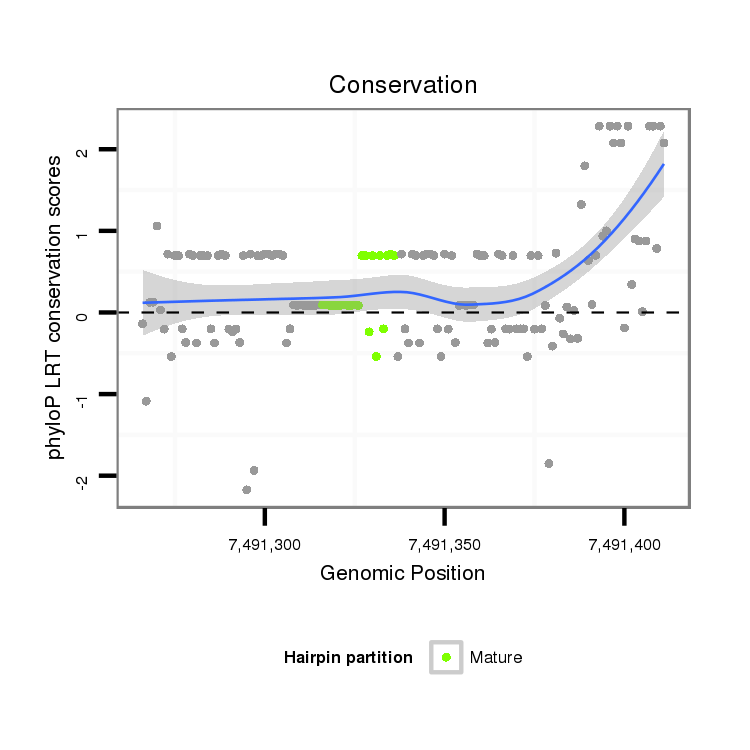
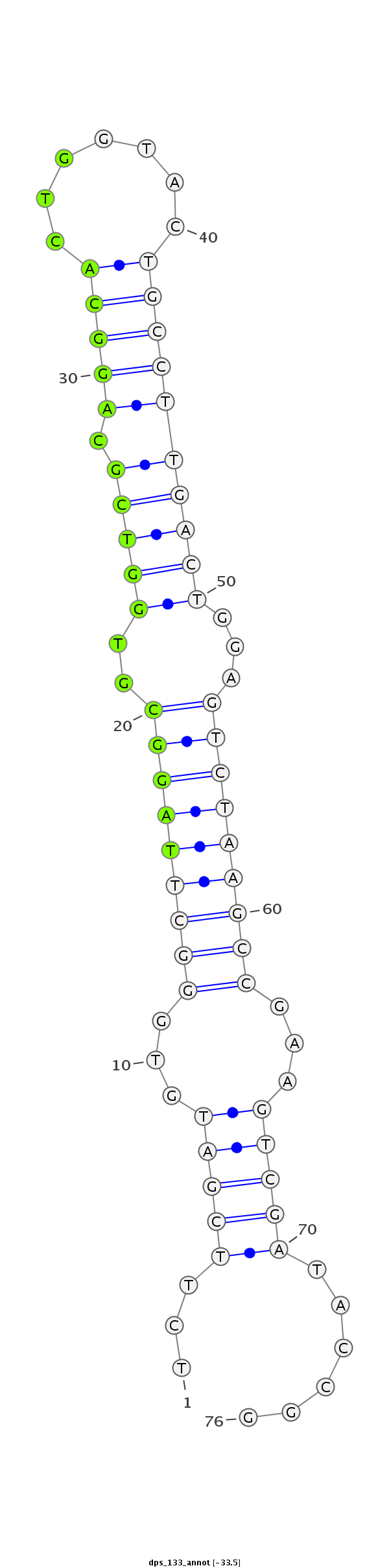
ID:dps_133 |
Coordinate:XL_group1a:7491316-7491361 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -33.5 |
 |
intergenic
No Repeatable elements found
| mature | star |
|
CTCCTGGTGGCAAAGGGCCTGCTGCACTGCATGCTTCTTCGATGTGGGCTTAGGCGTGGTCGCAGGCACTGGTACTGCCTTGACTGGAGTCTAAGCCGAAGTCGATACCGGACCCGTAGTGACGGAGTCCTGTGCAACTGTTTCTG
***********************************...(((((...(((((((((.((((((.(((((.......)))))))))))..)))))))))...)))))......*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR902011 testis |
V112 male body |
M040 female body |
GSM444067 head |
M022 male body |
|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................TAGGCGTGGTCGCAGGCACTG........................................................................... | 21 | 0 | 2 | 3.00 | 6 | 0 | 4 | 1 | 0 | 1 |
| ....................................................................CTGGTACTGCCTTGACTGGAGTCTAA.................................................... | 26 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 |
| ..............................................................................CTTGACTGGAGTCTAAGCCGA............................................... | 21 | 0 | 1 | 2.00 | 2 | 1 | 0 | 1 | 0 | 0 |
| ..............................................................................CTTGACTGGAGTCTAAGCCG................................................ | 20 | 0 | 1 | 2.00 | 2 | 1 | 1 | 0 | 0 | 0 |
| ..................................................................CACTGGTACTGCCTTGACTG............................................................ | 20 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 |
| ....................................................................CTGGTACTGCCTTGACTGGAG......................................................... | 21 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 |
| ...................................TCTTCGATGTGGGCTTAGGC........................................................................................... | 20 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 |
| ................................................CTTAGGCGTGGTCGCAGGCAC............................................................................. | 21 | 0 | 1 | 2.00 | 2 | 1 | 1 | 0 | 0 | 0 |
| ..........................................................................CTGCCTTGACTGGAGTCTAAG................................................... | 21 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 |
| ...................................................AGGCGTGGTCGCAGGCACTGG.......................................................................... | 21 | 0 | 2 | 1.50 | 3 | 2 | 1 | 0 | 0 | 0 |
| .....................................................................................................................AGTGACGGAGTCTTGTGCAA......... | 20 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................CTGCCTTGACCGGAGTCTAAG................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................ATACCGGACCCGTAGTGACG...................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| .....................................................GCGTGGTCGCAGGCACTGGTAC....................................................................... | 22 | 0 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................................ATACCGGAGCCGTAGCGA........................ | 18 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 |
| .....................................................GCGTGGTCGCAGGCACTGGTA........................................................................ | 21 | 0 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..................................................TAGGCGAGGTCGCTGGCACTG........................................................................... | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 |
| ........................................................TGGTCGCAGGCACTGGTAC....................................................................... | 19 | 0 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ......................................................CGTGGTCGCAGGCACCGGT......................................................................... | 19 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| .......................................................GTGGTCGCAGGCACTGGTA........................................................................ | 19 | 0 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 |
| ......................................................CGTGGTCGCAGGCACTGGTAC....................................................................... | 21 | 0 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ....................................................GGCGTGGTCGCAGGCACTGG.......................................................................... | 20 | 0 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| .....................................................GCGTGGTCGCAGGCACTGGT......................................................................... | 20 | 0 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................ACCCGCAGTTTCGGAGTCCTG.............. | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................GGTACCGCCTTGGCTGGCGT........................................................ | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 |
|
GAGGACCACCGTTTCCCGGACGACGTGACGTACGAAGAAGCTACACCCGAATCCGCACCAGCGTCCGTGACCATGACGGAACTGACCTCAGATTCGGCTTCAGCTATGGCCTGGGCATCACTGCCTCAGGACACGTTGACAAAGAC
***********************************...(((((...(((((((((.((((((.(((((.......)))))))))))..)))))))))...)))))......*********************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR902011 testis |
M062 head |
GSM343916 embryo |
M040 female body |
|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................CTCAGATTCGGCTTCAGCTATGGCCT.................................. | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .....................................................................................TCTCAGATTCGGCTTCAGCTA........................................ | 21 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .........................................................CCAGCGTCCGTGACCATGA...................................................................... | 19 | 0 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 |
| ......................................................GCACCAGCGTCCGTGACCATGAC..................................................................... | 23 | 0 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 |
| .......................................................CACCAGTGTCCGTGACCATGA...................................................................... | 21 | 1 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 |
| ...............................................................................................GGCTTCATCTTTGGCCCGG................................ | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 |
| ..............................................................................................CGGCTTAAGCTATCGTCTG................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| dp5 | XL_group1a:7491266-7491411 + | dps_133 | CTCCTGGTGGCAAAGGGCCTGCTGCACTGCATGCTTCTTCGATGTGGGCTTAGGCGTGGTCGCAGGCACTGGTACTGCCTTGACTGGAGTCTAAGCCGAAGTCGATACCGGACCCGTAGTGACGGAGTCCTGTGCAACTGTTTCTG |
| droPer2 | scaffold_13:2133628-2133773 + | CTCCTGGTGGCAAAGGGCCTGCTGCACTGGAGGCTTCTTCGATGTGGGCTTAGGCGTGGTCGCAGGCACTGGTACTGCCTTGACTGGAGTCTAAGCCGAAGTCGATACCGGACTCGTAGTGACGGAGTCCTGTGCAACTGTTTCTG | |
| droVir3 | scaffold_12932:1545826-1545849 - | C--------------------------------------------------------------------------------------------------------------------------GGCTTCTTGTGCAGCCGCTTGTG | |
| droMoj3 | scaffold_6473:3063194-3063310 + | CGCCTAATCGCGCAGTGCCCTCTGTTTGGGATGCTTCTTCTG-------------------GCTGCCGCTGCTGATGACTTGGGTAGC-----AGCAACAGCTGGCGGCAGG----T-GGCGCGACTTCTTGTGCAGCCGCTTGTG | |
| droAna3 | scaffold_13117:4673182-4673200 + | -------------------------------------------------------------------------------------------------------------------------------TCTTGTGCAGCCTCTTGTG | |
| droKik1 | scf7180000302592:1568613-1568638 + | G------------------------------------------------------------------------------------------------------------------------ACGGTTTCTTGTGCAGTCGCTTGTG | |
| droFic1 | scf7180000453882:502956-502974 + | -------------------------------------------------------------------------------------------------------------------------------TTTTGTGCAGCCGCTTGTG | |
| droEle1 | scf7180000491023:441498-441521 + | C--------------------------------------------------------------------------------------------------------------------------GGCTTTTTGTGTAGCCGCTTGTG | |
| droBia1 | scf7180000302126:3584645-3584668 + | C--------------------------------------------------------------------------------------------------------------------------GGCTTTTTGTGCAGCCTCTTGTG | |
| droTak1 | scf7180000415394:490939-490972 + | CCCCTG----------------------------------------------------------------------------------------------------------------TCGCCGGCTTTTTGTGCAGCCGCTTGTG | |
| droEug1 | scf7180000409538:33178-33201 - | C--------------------------------------------------------------------------------------------------------------------------GGCTTTTTGTGTAACCGCTTGTG | |
| dm3 | chrX:10654594-10654629 + | TACT--------------------------------------------------------------------------------------------------------------GGGCTCGCCGGCTTTTTGTGAAGCCTCTTGTG | |
| droSim2 | x:10113118-10113152 + | ACT---------------------------------------------------------------------------------------------------------------GGGCTCGCCGGCTTTTTGTGAAGCCTCTTGTG | |
| droSec2 | scaffold_15:1352989-1353023 + | ACT---------------------------------------------------------------------------------------------------------------GGGCTCGCCGGCTTTTTGTGAAGCCTCTTGTG | |
| droYak3 | X:19223874-19223897 + | C--------------------------------------------------------------------------------------------------------------------------GGCTTTTTGTGAAGCCTCTTGTG | |
| droEre2 | scaffold_4690:4475864-4475887 - | C--------------------------------------------------------------------------------------------------------------------------GGCTTTTTGTGGAGCCTCTTGTG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/15/2015 at 02:40 PM