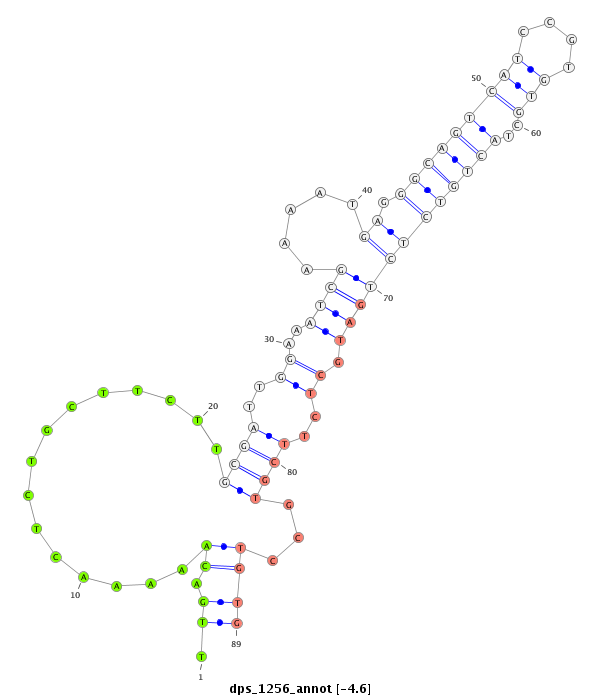
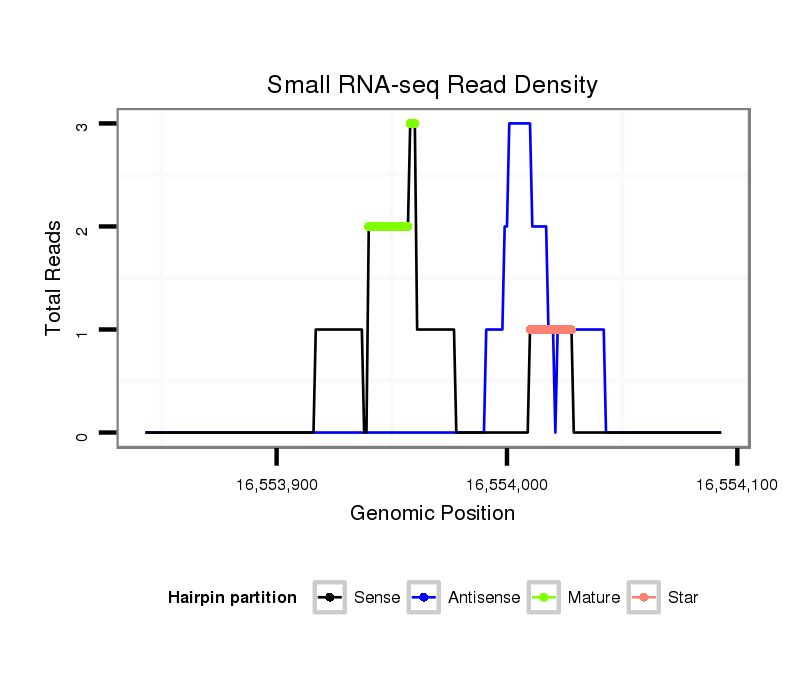
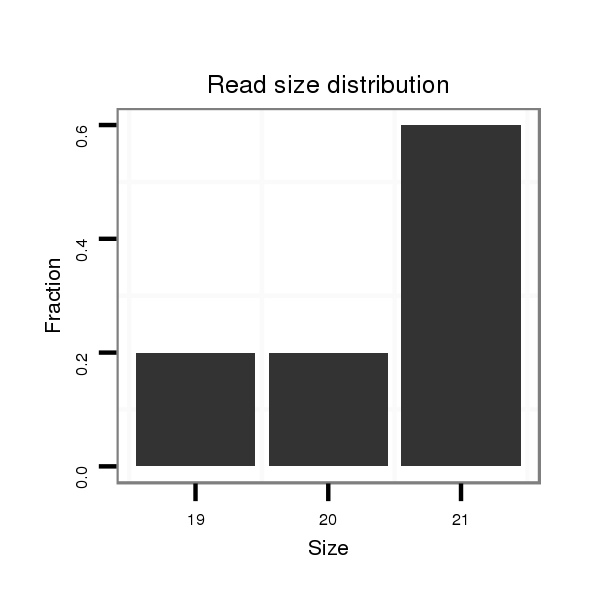
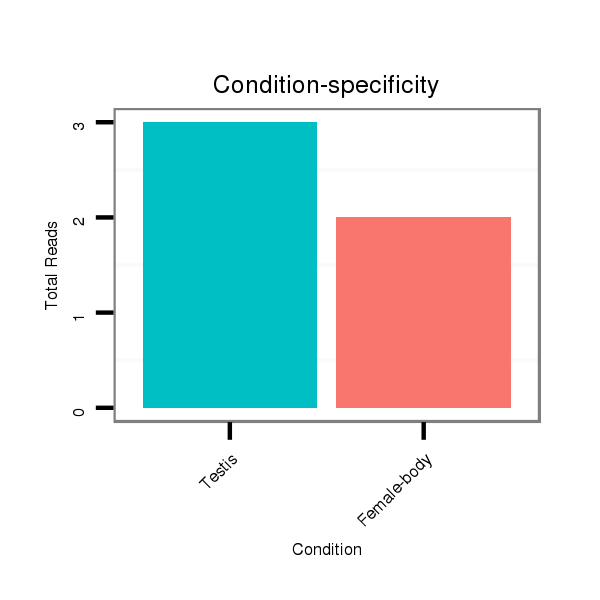
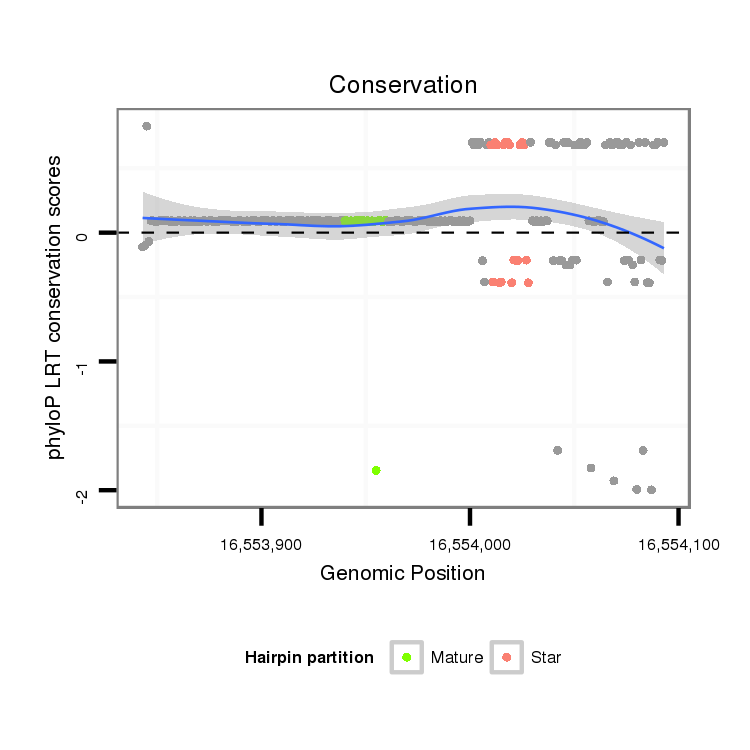
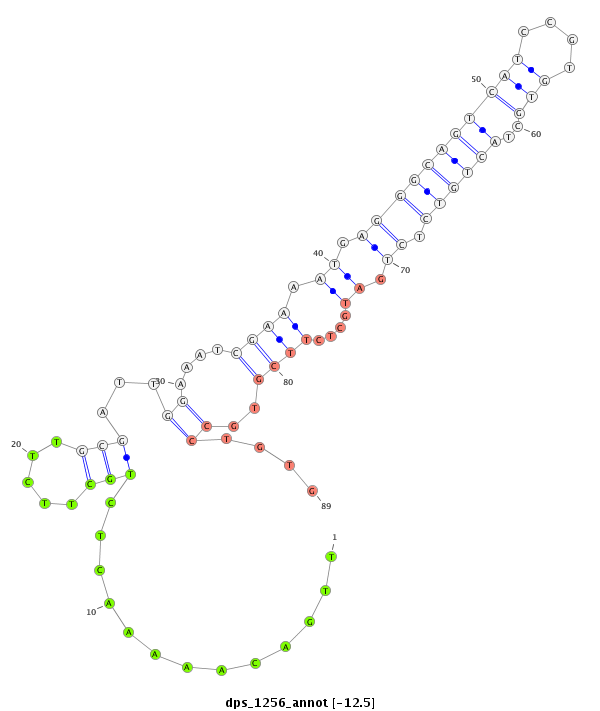
ID:dps_1256 |
Coordinate:3:16553893-16554043 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -12.5 | -12.5 | -12.4 | -12.4 |
 |
 |
 |
 |
CDS [Dpse\GA15761-cds]; exon [dpse_GLEANR_16248:2]; intron [Dpse\GA15761-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AGTTTTCCTTTAACAGCATCACCAGTCGCTTCTAGGCAAATATGCCATCGTGTTTGCTATAGATGATCCAATACATTTCTCTTTATCCTTTCCGGAATTGACAAAAACTCTGCTTCTTGCGATTGGAAATCGAAAATGAGGGCAGTCATCCGTGTGCTACTGTCTCTGATGCTCTTCGTGCCTGTGTTCTGTGAAATCCAGGTTATAGACAGGAAACCATCGGTAGATTGGGATCAAAATATCCTGGATGA *************************************************************************************************.((.((...............((((..((..((((.....((.(((((((((....)))..)))))))))))).))..))))...))))***************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M040 female body |
SRR902011 testis |
M059 embryo |
M062 head |
V112 male body |
|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................................................................GAGGTTATAGACCCGAAACCA................................ | 21 | 3 | 1 | 9.00 | 9 | 9 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................GGTTATAGACCCGAAACCA................................ | 19 | 2 | 1 | 7.00 | 7 | 7 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................CGAGGTTATAGACCCGAAACC................................. | 21 | 3 | 1 | 4.00 | 4 | 3 | 0 | 1 | 0 | 0 |
| .....................................................................................................................................................................................................CGAGGTTATAGACCCGAAACCA................................ | 22 | 3 | 1 | 3.00 | 3 | 3 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................AGGTTATAGACCCGAAACCA................................ | 20 | 2 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 |
| .................................................................................................TTGACAAAAACTCTGCTTCTT..................................................................................................................................... | 21 | 0 | 1 | 2.00 | 2 | 0 | 2 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................GAGGTTATAGACCCGAAACC................................. | 20 | 3 | 2 | 2.00 | 4 | 4 | 0 | 0 | 0 | 0 |
| ...................................................................................................................CTTGCGATTGGAAATCGAAA.................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................GATGCTCTTCGTGCCTGTG................................................................. | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................AGGTTATAGACCCGAAACCAG............................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................AGGTTATAGACCCGAAACC................................. | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................ATTTCTCTTTATCCTTTCCGG............................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................GGTTATAGACCCGAAACCAG............................... | 20 | 3 | 5 | 0.80 | 4 | 4 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................GAGGTTATAGACCCGAAAC.................................. | 19 | 3 | 6 | 0.67 | 4 | 4 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................CGAGGTTATAGACCCGAAA................................... | 19 | 3 | 7 | 0.29 | 2 | 1 | 0 | 0 | 1 | 0 |
| ..........................................................................................................................................................................................................................ATCGGTAGATCGGGATGAT.............. | 19 | 3 | 20 | 0.10 | 2 | 1 | 0 | 0 | 0 | 1 |
| ..........................................................................................................................TTGGAAACCGAAGATGTGGG............................................................................................................. | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 1 |
| .....................................AAATATTCCATCGTCGTTGC.................................................................................................................................................................................................. | 20 | 3 | 18 | 0.06 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................................................GATCCAATACCGTGCTCT......................................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
|
TCAAAAGGAAATTGTCGTAGTGGTCAGCGAAGATCCGTTTATACGGTAGCACAAACGATATCTACTAGGTTATGTAAAGAGAAATAGGAAAGGCCTTAACTGTTTTTGAGACGAAGAACGCTAACCTTTAGCTTTTACTCCCGTCAGTAGGCACACGATGACAGAGACTACGAGAAGCACGGACACAAGACACTTTAGGTCCAATATCTGTCCTTTGGTAGCCATCTAACCCTAGTTTTATAGGACCTACT
*****************************************************************.((.((...............((((..((..((((.....((.(((((((((....)))..)))))))))))).))..))))...))))************************************************************************************************* |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR902011 testis |
V112 male body |
M040 female body |
GSM343916 embryo |
|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................................................CAGCTACTAGGAGAAGCACGG..................................................................... | 21 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| ....................................................................................................................................................AGGCACACGATGACAGAGAC................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ............................................................................................................................................................GATGACAGAGACTACGAGA............................................................................ | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................................................CGGACACAAGACACTTTAGGT................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................................................TGACAGAGACTACGAGAAGC......................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................................................TGCCATAGACTACTAGAAGC......................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 |
| ..........................................................GATCTATTAAGTTATGTAAAGA........................................................................................................................................................................... | 22 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 |
| .......................................................................................................................................................................................CATATGACACTTTAGGTGC................................................. | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 1 |
| ...............................................................................................................................................................................................................................CCCTAACCCTAGTTATATAG........ | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 |
| ...........................................................................................................................................................CGATGTCAGAGAATTCGAG............................................................................. | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 1 | 0 |
| ..............................................................................................................................................................TGCCATAGACTACTAGAAG.......................................................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 |
| ........ATTCTGTCGTAGTGGTCAG................................................................................................................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| dp5 | 3:16553843-16554093 + | dps_1256 | AGTTTTCCTTTAACAGCATCACCAGTCGCTTCTAGGCAAATATGCCATCGTGTTTGCTATAGATGATCCAATACATTTCTCTTTATCCTTTCCGGAATTGACAAAAACTCTGCTTCTTGCGATTGGAAATCGAAAATGAGGGCAGTCATCCGTGTGCTACTGTCTCTGATGCTCTTCGTGCCTGTGTTCTGTGAAATCCAGGTTATAGACAGGAAACCATCGGTAGATT----GGGATCAAAATATCCTGGATGA |
| droPer2 | scaffold_4:207711-207961 - | AGTTTTCCTTTAACAGCATCACCAGTCGCTTCTAGGCAAATATGCCATCGTGTTTGCTATAGATGATCCAATACATTTCTCTTTATCCTTTCCGGAATTGACAAAAACTCTGTTTCTTGCGATTGGAAATCGAAAATGAGGGCAGTCATCCGTGTGCTACTGTCTCTGATGCTCTTCGTGCCTGTGTTCTGTGAAATCCGGGTTATAGACAGGAATCCATCGGTAGCTT----GGGATCAAGATGTCCAGGATGA | |
| dm3 | chr2R:13740937-13740940 + | ATTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 2r:14430729-14430732 + | ATTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_1:11236701-11236704 + | ATTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 2R:15441999-15442081 - | CT------------------------------------------------------------------------------------------------------------------------------------------------------------ACTGTTGCTGCTGAGCTTCTCATCTGCTT--------ATTCAAATAAAGGGCAGGA--------GGAGCTTCAGAGGAACCTCTACATAACGGACAA | |
| droEre2 | scaffold_4845:7968070-7968073 + | ATTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/18/2015 at 12:18 AM